GÉNOMIQUE ET INFORMATIQUE : L'impact sur les thérapies et sur l'industrie pharmaceutique
SERUSCLAT (Franck)
RAPPORT 20 (1999-2000) - OFFICE PARLEMENTAIRE D'EVALUATION DES CHOIX SCIENTIFIQUES ET TECHNOLOGIQUES
Fichier
au
format
Acrobat
( 1097 Ko )
Table des matières
-
1. PREMIÈRE PARTIE : UNE RÉVOLUTION SCIENTIFIQUE
- 1.1. DES CONNAISSANCES ET DES TECHNIQUES ENTIÈREMENT NOUVELLES
- 1.2. DE MULTIPLES APPLICATIONS
-
2. DEUXIÈME PARTIE : DE NOUVEAUX CHOIX À FAIRE
- 2.1. POUR LA RECHERCHE
- 2.2. POUR L'INDUSTRIE
- 2.3. POUR LA SOCIÉTÉ
|
N° 1871
|
|
N° 20
|
|
|
Enregistré à la Présidence de l'Assemblée nationale |
Annexe au procès-verbal de la séance du |
||
|
le 15 octobre 1999 |
14 octobre 1999 |
||
|
|
|||
OFFICE
PARLEMENTAIRE D'ÉVALUATION
DES CHOIX SCIENTIFIQUES ET TECHNOLOGIQUES
RAPPORT
sur
GÉNOMIQUE ET INFORMATIQUE : L'IMPACT SUR LES THÉRAPIES
ET SUR L'INDUSTRIE PHARMACEUTIQUE
par
M. Franck SÉRUSCLAT,
Sénateur.
|
Déposé sur le
Bureau de l'Assemblée nationale
|
Déposé sur le
Bureau du Sénat
|
Recherche - Biologie - Génétique - Médicaments - Santé.
INTRODUCTION
INTRODUCTION
I. STRUCTURE DU RAPPORT
I. STRUCTURE DU RAPPORT
À l'évidence, nous assistons à un bouleversement des moyens utilisés pour
guérir les
hommes malades et non seulement les soigner.
Chaque jour, la presse, spécialisée ou non, fait état des recherches et de leurs
résultats prévisibles ; la presse spécialisée en confirme la plupart tout
en
restant prudente dans ses audaces.
L'Office parlementaire d'évaluation des choix scientifiques et technologiques a
adopté
un rapport destiné à faire le point sur les enjeux, déjà perceptibles ou
prévisibles,
de cette rencontre entre l'informatique et la génomique.
Ce rapport, réalisé à partir d'informations recueillies en France comme aux
États-Unis, nourri de la lecture de la presse spécialisée et de la consultation
d'une
documentation scientifique comprend deux parties :
La première partie
décrit la véritable
RÉVOLUTION SCIENTIFIQUE
qui
bouleverse depuis quelques années le domaine de la santé.
De façon aussi précise et détaillée que le permettent les informations
recueillies,
sont présentées en deux chapitres :
-
LES CONNAISSANCES ET LES TECHNIQUES ENTIÈREMENT NOUVELLES
récemment
maîtrisées :
|
La génomique : |
Étude des génomes des organismes, en particulier de l'ensemble des gènes et de leur disposition sur les chromosomes ;
|
|
La bio-informatique : |
Combinaison de l'informatique et de la biologie, qui permet de déchiffrer les génomes et d'analyser l'information génétique ;
|
|
Les biopuces : |
Supports issus de la micro-électronique classique, mais sur lesquels sont fixés des fragments d'ADN permettant d'analyser d'autres brins d'ADN ;
|
|
La chimie combinatoire et le criblage à haut débit : |
Synthèse par combinaison chimique de très nombreuses molécules constituant des candidats-médicaments et tri rapide de ces molécules en fonction de leur action sur les cibles que constituent, par exemple, les protéines.
|
- LEURS MULTIPLES APPLICATIONS :
|
L'utilisation pour la recherche pharmaceutique de cibles issues de la génomique : |
Chaque gène code pour une protéine ; la déficience ou l'excès de protéine est à l'origine de nombreuses pathologies. L'utilisation des protéines identifiées grâce à la génomique, permet de mieux orienter la recherche pharmaceutique ;
|
|
La thérapie génique : |
Réparation d'un gène ou apport in situ d'un gène fonctionnel ;
|
|
Les nouveaux vaccins : |
Nouvelles techniques d'immunothérapie ; vaccins à base d'ADN ; vaccins traditionnels découverts grâce à la connaissance du génome des bactéries ;
|
|
La pharmacogénomique : |
Adaptation des traitements aux malades en fonction de leur profil génétique ;
|
|
Le diagnostic moléculaire : |
Tests ciblant le patrimoine génétique et permettant de détecter les maladies infectieuses ou génétiques ;
|
|
Les protéines thérapeutiques : |
L'utilisation des techniques du génie génétique pour la production de protéines par des bactéries ou des levures et, plus récemment par des animaux génétiquement modifiés. |
La deuxième
partie
présente les
NOUVEAUX CHOIX À FAIRE EN FRANCE
, pour
bénéficier de
cette révolution scientifique, dans trois domaine :
LA RECHERCHE, ses STRUCTURES et ses ORIENTATIONS fondamentales, notamment
vers la
protéomique
(étude des protéines) ;
L'INDUSTRIE, les ACTIONS EN FAVEUR DES JEUNES ENTREPRISES DE
BIOTECHNOLOGIE, les
BIOPÔLES ainsi que le problème des BREVETS ;
LA SOCIÉTÉ, avec les deux aspects spécifiques de la FORMATION
PROFESSIONNELLE et
de la MÉDECINE PRÉDICTIVE (étude génétique des prédispositions à certaines
pathologies).
La
conclusion
propose une série de recommandations pour profiter de la
révolution
génomique et en maîtriser les conséquences.
II. PROPOS LIMINAIRES : GÉNOMIQUE INTIME
II.
PROPOS
LIMINAIRES : GÉNOMIQUE INTIME
Je tiens avant tout à remercier
M. Jacques DANGOUMAU
, professeur
des
universités, praticien hospitalier, pharmacologue et
M. Yves
CHAMPEY
,
président de la Fondation Rhône-Poulenc Rorer, qui ont bien voulu constituer,
pour
m'assister dans l'élaboration de ce rapport, un comité de pilotage dont les
conseils
m'ont été infiniment précieux.
Dès le début de la préparation de ce rapport, et la rédaction de l'étude de
faisabilité préalable, j'ai découvert combien avaient progressé les
connaissances sur
la nature intime de l'être humain. Les scientifiques sont arrivés à identifier,
ou vont
y parvenir, la composition du génome humain et, bientôt, ils n'hésiteront pas à
le
prendre comme matrice de médicaments spécifiques, en feront une méthode
thérapeutique
ordinaire adaptée à une maladie pour un malade personnalisé. Décrypter ces
données
nouvelles est indispensable, l'objectif du rapport étant de mettre à la portée
de
chacun des informations claires sur des sujets complexes.
" L'élucidation de la structure de la double hélice de l'ADN, la
découverte de l'ARN messager, le déchiffrement du code génétique, le décryptage
de la
mécanique de la synthèse protéique, la compréhension des grands principes de la
régulation des gènes sont des morceaux d'anthologie, désormais classiques [...].
L'étude approfondie des systèmes n'a cessé de progresser, appuyée par une
impressionnante avancée des méthodes et des techniques. La plus spectaculaire a
été
sans doute l'avènement du génie génétique qui a offert aux biologistes une
méthode
quasi-générale pour isoler et purifier des gènes spécifiques, donc de les
analyser et
les manipuler à des fins cognitives ou productrices [...]. Dotée de concepts et
d'outils
performants, la recherche peut, au niveau moléculaire, aborder la confondante
diversité
du vivant [...] (en découvrant en même temps) l'homogénéité moléculaire du
vivant
qui contraste avec la grande diversité des formes [...]. À peine repère-t-on,
sur
quelques organismes très différents un trait marquant, que l'on peut formuler
une loi
générale s'appliquant à l'ensemble ou à des sous-ensembles du monde vivant
[...]. Un
biologiste moléculaire, aujourd'hui, raisonne et se documente de façon quasiment
indifférente sur des données obtenues chez des bactéries, des levures, la
mouche du
vinaigre, l'oursin, la torpille, le crapaud, le poulet, le lapin, la souris ou
l'homme
[...]. Jamais peut-être l'impact de découvertes fondamentales dans ses
applications n'a
été si rapide [...]
1(
*
)
.
L'origine de la vie, son évolution font étonnement, certes ; les causes
n'en sont,
cependant, plus ignorées et des connaissances scientifiques et techniques
peuvent prendre
place dans des débats philosophiques ou théologiques.
La vie des êtres humains, des végétaux comme des animaux, est portée par les
mêmes
substances chimiques, substances que l'homme sait synthétiser dans ses
cornues :
l'ADN, deux bases puriques, deux pyrimidiques, 24 acides aminés, c'est
tout. Avec
des milliards de combinaisons, l'homme ne reproduit que des être humains, tous
différents entre eux, noirs, jaunes, métis, blancs, avec des yeux bridés ou
non, hommes
grands ou petits, plus ou moins bancals, beaux ou vilains, mais toujours, et
seulement,
des êtres à visages, corps et comportements humains. Végétaux et animaux
gênèrent
des millions de différences, de l'arbre immense au pissenlit à la fleur d'une
délicatesse surprenante, dans le règne végétal, de l'éléphant au virus du SIDA
dans
le règne animal.
Les plus fins détails de l'organisation du génome humain se sont précisés. Les
milliards de nos cellules sont, avec des fonctions précises, réparties dans
tous nos
organes, peau et muscles, coeur, foie, cerveau, rein, pancréas, etc. Ces
cellules, sauf
celles qui nagent dans le sang, ont des ponts entre elles pour être maintenues
en
place ; dans le noyau de chacune d'entre elles est pelotonné l'ADN avec
ses bases
puriques et pyrimidiques organisées selon la séquence née de la fusion des
patrimoines
parentaux. Chacune de ces cellules pourrait intervenir dans la naissance et la
vie de
n'importe quel organe, mais chacune d'elle est limitée au rôle nécessaire pour
l'organe
où elle se trouve logée ; celles qui sont dans le tissu musculaire ne
fourniront
pas du tissu cérébral, cardiaque, ou rénal, etc. Le détail de l'intervention de
l'ARN
messager, la présence de verrous répressifs ne laissant s'exprimer que la
séquence
nécessaire d'ADN ne sont pas encore parfaitement connus.
L'objectif des scientifiques est d'acquérir la connaissance intime de ces
mécanismes et
leurs maîtrises ; un jour, ils construiront l'homme à leur manière pour
créer
" leur meilleur des mondes ". Fol espoir ! Terrible
inquiétude !
Dégager les conséquences du recours au génome comme matière pour des
" médicaments génétiques " conduit à se demander si l'homme
ne
serait qu'une étonnante " machine " faite de moteurs
chimiques pour
penser, créer, imaginer, aimer ou détester, caresser, torturer, tuer ou
protéger,
enchanter ou effrayer... La vie ne serait-elle plus ce "
miracle "
impressionnant tant Menuhin (
" Je suis né avec un héritage qui
date de
milliers d'années. L'enfant est l'incarnation de vies antérieures ; on
croît qu'il
est nouveau-né, mais il est le miracle d'une vie qui n'a pas été interrompue
depuis
l'origine de l'homme "
). Vie de l'homme, vie des animaux, vie des
plantes,
toutes ont les mêmes éléments pour accomplir les actes les plus essentiels
comme les
plus subtils de la vie à la mort. Chaque seconde, fraction de seconde, fraction
de
fraction de seconde, nos comportements les plus secrets, les plus ordinaires
comme les
plus compliqués en seraient-ils dépendants ? La chimie supplanterait-elle
toutes
les autres hypothèses ? Rendrait-elle caduc ce qui était légende ou
mystère ?
Les premiers éléments réunis pour l'élaboration de ce rapport ont mis en
lumière une
fulgurante évolution démultipliée par le développement parallèle des moyens
informatiques mis à la disposition de la recherche, et celui des connaissances
de la
structure intime de l'être humain.
Génomique, chimie combinatoire, informatique, thérapie génique, sont des mots
qui
aujourd'hui enthousiasment les uns, inquiètent les autres, font naître des
espérances
ou des inquiétudes ; jusqu'où l'homme va-t-il oser aller, sans risques
majeurs ? Quelles chances, quels risques pour l'espèce humaine au moment
où il
disposera des clefs de son évolution dès avant la naissance, jusqu'à la fin de
la
vie ?
Pour en bien comprendre les rôles respectifs des composantes de ce génome
humain, il m'a
été utile d'en faire un recensement explicatif.
Chaque cellule humaine a un noyau et un cytoplasme, sauf les globules rouges.
Chaque cellule contient dans son noyau les 46 chromosomes porteurs des
facteurs
déterminant de l'hérédité
Le chromosome :
Au début du XIXe siècle, l'examen
microscopique
de cellules animales et végétales traitées par certains colorants révéla la
présence
dans leurs noyaux de corps colorés qu'on appela chromosomes, du grec
khrôma,
couleur,
et
soma,
corps.
Chaque chromosome est porteur de nombreux gènes originaux mais il n'a pas de
fonction
propre pour autant. Il n'y a pas un gène qui soit porteur d'une finalité type
par
exemple " yeux bleus ". C'est la conjugaison des gènes de
plusieurs
chromosomes qui permettra que ce caractère héréditaire apparaisse.
Ceci est vrai pour tous les chromosomes, sauf les chromosomes sexuels.
Chaque chromosome n'a pas de spécificité par lui-même, sauf sa forme.
Le composant principal des chromosomes est l'ADN (acide désoxyribonucléique) qui
possède une structure en double hélice lui permettant de stocker et de
transmettre une
information génétique
" À n'en pas douter, l'élucidation de
la
structure de l'ADN marque une étape majeure dans la compréhension du vivant et
ouvre
pour la recherche une nouvelle ère : celle de la biologie moléculaire du
gène. Ce
dernier, support de l'hérédité, vient en effet de trouver sa
nature... "
2(
*
)
.
Cette découverte,
le 25 avril 1953, par CRICK et
WATSON fut saluée par les plus brillants biologistes du monde et par Salvador
DALI :
" Aujourd'hui, les dernières découvertes de la science nous
prouvent que
les lois de Dieu sont celles de l'hérédité contenue dans l'acide
désoxyribonucléique,
ADN, et que l'acide ribonucléique, ARN, n'est que le messager chargé de
transmettre le
code génétique, qui est le
legi intimus
des deux acides en question
formant ici
l'échelle de Jacob de CRICK et WATSON "
3(
*
)
.
Cette première découverte n'était, pourtant, pas suffisante pour connaître et
comprendre le mécanisme intime de la transmission de l'information génétique.
Il a fallu que SANGER, prix Nobel en 1958, interprète le rôle des protéines à
partir
des premiers travaux de BANTING et BEST en 1922 sur l'insuline, première
protéine
isolée à l'état pur, composée de 177 acides aminés ; il démontra que
ces
acides aminés n'étaient pas dans un ordre aléatoire mais en séquence bien
déterminée
et, si une seule erreur intervient dans cet agencement, l'insuline perd ses
propriétés.
Il en conclut que ces protéines étaient de grosses molécules chimiquement
définies.
Ce sont, enfin, les travaux de J. MONOD, F. JACOB et A. WOLF,
prix Nobel en
1965, qui ont permis de percer les mystères du mécanisme de la régulation
génétique
au niveau de la cellule : on leur doit la découverte de l'ARN messager
ainsi que
celle des étapes de la participation des acides aminés à la constitution des
protéines
du vivant, qu'il s'agisse des être humains, des animaux, des bactéries, des
microbes
mais aussi des plantes : pour tous ADN, ARN messager, protéines et acides
aminés,
en des quantités totalement différentes, sont les supports de la transmission
de tous
leurs caractères, jusque dans leurs moindres détails. Cet ADN est localisé dans
le
noyau des cellules et sert de matrice pour la synthèse des différents types
d'acide
ribonucléique, ou ARN, par le processus de transcription. Il est le support de
la
transmission héréditaire.
La molécule d'ADN est une molécule codée que l'on peut considérer comme un
" mot " formé de plusieurs millions de lettres écrites avec
un
alphabet réduit de 4 lettres : A, C, G et T (adénine, cytosine,
guanine et
thymine). L'ordre dans lequel sont placés ces 4 constituants s'appelle une
information
codée.
Le génome
est composé de tous les chromosomes d'un organisme
vivant,
humain, animal vertébré ou non, plante..., donc de tous les gênes qu'il
contient, sans
qu'il y ait de mélange.
La double hélice est constituée par le génome, donc par tous les chromosomes ,
donc par
tous les gènes. Mais cette double hélice n'a pas pour autant un contenu physique
unique ; elle est faite des 46 chromosomes restant
individualisés : elle
est faite de 46 fragments.
Le gène
est la portion d'un chromosome qui commande l'expression
d'un
caractère héréditaire précis : c'est un tout petit fragment du chromosome
Y qui
détermine le sexe ; si ce petit fragment est présent dans la cellule oeuf,
il
entraîne la " fabrication " d'un garçon ; s'il est
absent,
d'une fille... La précision de leur localisation, permet d'établir, peu à peu,
les
cartes génétiques.
Chaque gène est constitué par une séquence de 4 bases allant toujours deux
à
deux : adénine et thymine , bases puriques, et guanine et cytosine, bases
pyramidiques. On évalue à environ 80 000 à 100 000 le nombre de
gènes.
Dans le cytoplasme sont produits et assemblés une
vingtaine d'acides
aminés
déterminant la constitution des caractéristiques génétiques ; On distingue
des :
- acides aminés essentiels apportés par l'alimentation et que l'individu
ne peut
synthétiser
- acides aminés ordinaires synthétisés par les cellules.
Ces acides aminés seront ordonnés (= mis en ordre) par l'ARN messager qui, sous
la
dictée de l'ADN, aura copié cet ordonnancement. Pour cela, l'ARN messager, avec
sa
copie, quitte le noyau, met en ordre les acides aminés contenus dans le
cytoplasme selon
les directives recopiées sur le fragment d'ADN dont il est le correspondant.
Ainsi se construisent les protéines utiles :
- les unes aux formes de la cellule (ce sont des briques de
construction) ;
- les autres aux fonctions assurées par les cellules.
L'altération, l'absence d'un des éléments ou d'un ensemble d'entre eux peuvent
être à
l'origine de maladies. La découverte de la cause génétique d'une maladie, sauf
dans le
cas où celle-ci serait ou paraîtrait être due à un seul gène, est
particulièrement
difficile, tant le nombre de facteurs est élevé.
40 mille milliards de cellules (peau, muscles, nerfs...) contiennent,
chacune dans
leur noyau, 23 paires de chromosomes. Ces 23 paires sont enfermées
dans un
zygote totipotent et omnipotent, cellule initiale née du mariage entre le
spermatozoïde
du père et l'ovule de la mère ; celui-ci contient toutes les instructions
nécessaires à notre création puis survie.
Chaque chromosome est un long filament d'acide désoxyribonucléique (ADN). Cette
molécule est un serpentin à deux bandes formées d'une longue suite d'unités
fondamentales, les minuscules nucléotides, eux-mêmes constitués de trois
molécules : un phosphate, un sucre et une base. Phosphates et sucres
forment le
serpentin qui s'enroule pour former une double hélice. Les bases, associées
deux à
deux, forment des petits liens perpendiculaires aux deux bandes comme les
barreaux d'une
échelle torsadée.
La totalité de notre matériel génétique est constituée par quelques trois
milliards
de ces barreaux sur ces minces filaments d'un millième de millimètre
d'épaisseur.
Les quatre bases (adénine, cytosine, guanine, thymine, désignés par leurs
initiales A,
C, G, T), sont disposées, par paires, en vis-à-vis, sur chacune des deux
bandes, selon
une règle immuable : l'adénine est toujours associée à la thymine (A-T) et
la
cytosine à la guanine (C-G). À chaque gène correspond une information génétique
définie par le nombre et l'ordre de succession des paires de bases au sein du
gène ; le nombre de paires de bases sur ces gènes peut aller de 800 à plus
d'un
million par gène.
Les lettres A, C, G, T, peuvent être considérés comme quatre notes pour écrire
la
partition de la vie. La lecture de cette partition se traduit par la
fabrication de
protéines (ou d'enzyme, protéine ayant une fonction d'un type précis,
catalytique)
toutes constituées d'une chaîne d'acides aminés qui, au départ, nagent dans la
cellule ; ils sont alignés dans un ordre bien déterminé et caractéristique
de la
protéine en question. Chaque assemblage de bases puriques correspond à un acide
aminé
particulier : le codon CGA code pour l'acide aminé alanine, le triolet CCA
pour la
proline...
Dans la cellule, ces opérations sont dirigées par un organite appelé
ribosome : il
déchiffre un assemblage de bases puriques et ordonne à l'ARN messager d'aller
chercher
l'acide aminé correspondant ; il lit un assemblage suivant, fait chercher
l'acide
aminé pour le faire accrocher au précédent, jusqu'à lire le dernier de la
phrase ; ce gène est limité à ses deux extrémités par deux triolets
caractéristiques, au début ATG qui code pour l'acide aminé méthionine ; à
la
fin, le triolet " dit non sens ", TAA, ne correspond à
aucun acide
aminé : il ferme le gène.
La molécule d'ADN mesure 1,80 m de long pour 2 millièmes de millimètre
d'épaisseur ; elle se pelotonne dans un espace de quelques micromètres.
Pour une compréhension plus facile de la fonction de chacun des composants,
génome,
chromosome, gène, base purique, acide aminé, protéine, le recours à des images
plus
familières peut être utile.
Comparaison avec une bibliothèque et des livres :
Le génome,
enfermé dans le noyau, ressemble à une bibliothèque avec 2 fois
23 rangées
de livres édités, les uns par la mère, les autres par le père ; la
confrontation
de ces deux éditions d'un même texte (la vie humaine) est à l'origine de la
diversité
humaine. Chaque édition a des petites différences, des petites erreurs dont le
mélange,
au fil des générations et des mariages, aboutit à créer nos particularité. Seul
l'ADN
mitochondrial échappe à cette règle. Chaque rangée correspond à un chromosome
constitué d'ADN et porteur de caractères héréditaires spécifiques à ce
chromosome
;
ces livres ne peuvent pas quitter cette bibliothèque sans le secours de
l'ARN
messager ; celui-ci prend une copie d'une partie de livre et la transfère
dans le
cytoplasme ; les acides aminés, nageant dans ce cytoplasme sont
comparables à des
briques ; ils sont assemblés selon un plan porté par l'ARN messager ;
celui-ci
transfère des plans ou fragments de plans exprimés en combinaison des
4 bases
puriques entre elles.
On peut comparer cette combinaison à un code comme le sont le morse ou un
langage ;
ce code sert à ordonnancer les acides aminés considérés comme des briques
capables de
construire des protéines adaptées aux multiples fonctions nécessaires à la vie.
III. PROPOS INTRODUCTIFS
III. PROPOS
INTRODUCTIFS
Pendant des siècles et des siècles, faisant référence à Hippocrate et Esculape,
médecins et apothicaires ont fait usage de plantes et de produits d'origine
animale pour
tenter d'adoucir les symptômes de maladies et la souffrance des malades. En ces
débuts,
la thériaque était une mixture complexe ayant vocation de médicament à effets
généralistes et, surtout, antidote des poisons les plus divers ; peu à
peu, des
onguents, des pommades, des sirops, des extraits et autres formes galéniques
ont pris
place dans les prescriptions médicales ; puis les Diafoirus, tant moqués et
décriés par Molière, ont usé et abusé de la saignée et du lavement.
La notion de " principes actifs " et leur extraction datent
du début
du XIXe siècle. Nombreux d'entre eux ont été isolés des végétaux, tels les
alcaloïdes (morphine en 1805, strychnine et quinine en 1818 et 1920, puis
cocaïne,
codéine...) et les hétérosides (digitaline cristallisée). En 1889, année de
l'exposition universelle à Paris, la recherche industrielle a fait ses premiers
pas et a
abouti à la découverte des antiseptiques, des digitaliques et des
antirhumatismaux.
En 1907, le médecin allemand, P. EHRLICH, en découvrant les arsenicaux de
synthèse
efficaces contre la syphilis et la maladie du sommeil, a donné naissance à la
chimiothérapie
.
Celle-ci a pris son essor rapidement, permettant la découverte
d'antiparasitaires, de
barbituriques, d'antipaludéens et de sulfamides.
Pendant la seconde guerre mondiale, l'ère des antibiotiques a commencé avec la
préparation à grande échelle de la pénicilline, puis la mise au point de la
streptomycine et des tétracyclines.
Sont ensuite apparus les psychotropes (phénothiazine, benzodiazépine), les
antituberculeux (isoniazide) les corticoïdes (prednissone) et, vers les années
soixante-dix, les médicaments cardio-vasculaires modernes (bêtabloquants).
Tous ces médicament ont permis d'élargir considérablement la palette des
possibilités thérapeutique, sans toutefois
répondre complètement aux
exigences
déjà exprimées par Pierre LAROUSSE dans son Grand Dictionnaire Universel du
XIXe siècle
:
" La vraie classification des médicaments reposera sur la
connaissance de
leurs effets précis et bien déterminés. Elle doit être établie, non d'après les
symptômes de guérison qu'il font apparaître dans les diverses maladies, mais
sur la
nature des modifications qu'ils déterminent dans tel ou tel tissu malade.
Chaque
tissu malade a son médicament
, comme chaque tissu sain a son
poison "
.
Nous assistons actuellement à la naissance de ces médicaments :
certains
seront administrés comme leurs prédécesseurs mais atteindront des cibles très
précises, d'autres seront de nature très différente, par exemple dans le
domaine de la
thérapie génique.
Cette rupture a pour origine la rencontre entre l'informatique et la génomique.
Elle va
bouleverser les modes de production et d'administration des substances
thérapeutiques,
avoir des répercussions considérables .
Voilà que depuis quelques 20 ans, l'homme découvre sa composition intime,
son
génome.
Il devient maintenant capable d'avoir la connaissance des rôles et des effets
de l'ADN,
des gènes des protéines, des allèles comme des microsatellites, de préciser
quelle est
la responsabilité de chacun de ses composants dans la quasi-totalité des
maladies qui
ont, donc, une cause cernable : une faiblesse ou une absence
génétique ;
voilà aussi qu'il peut les remettre en état de bon fonctionnement.
La connaissance des gènes provoque un bouleversement des connaissances et des
comportements en médecine courante, dans l'industrie pharmaceutique, comme dans
toutes
les activités qui gravitent autour de la vie et de la maladie des hommes.
Il apprend même la " dualité " des gènes, celui de la
prédisposition à la longévité étant également, par exemple, celui de la
prédisposition à l'infarctus du myocarde précoce...
Il commence à connaître la composition des protéines codées par les gènes, à
déterminer les conséquences pathologiques de l'excès ou du déficit de production
protéinique ; il peut essayer d'enrayer les maladies en agissant sur leur
cause
c'est-à-dire en régulant le niveau d'expression des protéines.
Cette véritable
Révolution scientifique
prélude
à de Nouveaux choix à
faire.
POUR LES TERMES
TECHNIQUES,
UN GLOSSAIRE EST CONSULTABLE EN FIN DE RAPPORT
1. PREMIÈRE PARTIE : UNE RÉVOLUTION SCIENTIFIQUE
1.1. DES CONNAISSANCES ET DES TECHNIQUES ENTIÈREMENT NOUVELLES
Elles sont apparues dans les domaines de la génomique, la bio-informatique, les biopuces et la chimie combinatoire associée au criblage à haut débit .
1.1.1. LA GÉNOMIQUE :
1.1.1.1. Définition et procédés
La génomique :
C'est l'étude
exhaustive des génomes et en particulier de l'ensemble des gènes, de leur
disposition
sur les chromosomes, de leur séquence, de leur fonction et de leur rôle.
Le génome des organismes vivants est l'ensemble de leur matériel génétique. Il
assure
le fonctionnement des cellules et la transmission des caractères héréditaires
au cours
des générations. Il est constitué de molécules d'acides nucléiques (ADN),
enchaînements d'unités élémentaires, les nucléotides. Les nucléotides sont
constitués d'un sucre, d'un phosphate, et d'un élément variable, la base, qui
peut
être l'adénine, la guanine, la cytosine ou la thymine. Les gènes, c'est-à-dire
les
parties d'ADN porteuses d'une information génétique, ne constituent qu'une
partie du
génome.
Les génomes des organismes vivants ont des tailles considérables allant d'une
centaine
de millions à des milliards de nucléotides. Le génome humain, par exemple, est
composé
d'environ 3 milliards de bases. L'étude d'un génome passe donc par des
opérations
de cartographie puis de séquençage ainsi que par l'interprétation des
séquences.
La cartographie physique :
C'est le
positionnement de repères sur le génome.
On commence par couper l'ADN en grands fragments. Les grands fragments clonés
de cette
collection sont ensuite ordonnés (cartographiés) les uns par rapport aux
autres, au
moyen de points de repère (courtes séquences d'ADN) qui servent de balises
identifiant
les grands fragments. Lorsque plusieurs fragments ont une balise en commun, on
en conclut
qu'ils ont une partie d'ADN en commun. On dit que les fragments sont
partiellement
recouvrants ou chevauchants.
En analysant l'ensemble des fragments d'ADN en fonction de leur contenu en
balises, on
peut reconstituer l'enchaînement des balises et des fragments d'ADN, tels
qu'ils existent
dans la molécule d'ADN de départ.
La reconstitution de la molécule d'ADN de départ sous la forme d'un ensemble de
fragments chevauchants constitue la carte physique. C'est à partir de cette
carte que
sera choisi l'ensemble minimal de fragments assurant la couverture complète du
génome à
séquencer.
Le séquençage :
Pour connaître
les " instructions " que renferme un fragment d'ADN, on lit
la
succession des bases puriques et pyrimidiques (A, T, G, C)
4(
*
)
de l'enchaînement. Cette lecture est appelée séquençage.
Un fragment d'ADN à séquencer est constitué de l'enchaînement de centaines
d'exemplaires de nucléotides dans un ordre défini. Séquencer une telle
molécule, c'est
déterminer cet ordre.
Le principe utilisé consiste à réaliser, à partir d'un point fixe, des copies
partielles de la molécule, interrompues au hasard. On synthétise toutes les
copies
intermédiaires possibles à partir du point fixe.

Puis on les sépare selon leur taille par une migration électrophorétique dans
un gel
poreux. Ces gels permettent de séparer deux intermédiaires consécutifs qui ont
une
différence de taille d'un seul nucléotide. Si l'on peut identifier le
nucléotide du
point d'interruption sur chacune de ces copies partielles, de la plus petite à
la plus
grande, il devient possible de reconstituer la succession des nucléotides tout
au long de
la copie.

Dans la pratique, pour identifier les nucléotides terminaux, l'ADN à séquencer
est
recopié à l'aide d'un composé chimique qui provoquera l'interruption au hasard,
mais
systématiquement à la suite d'un seul des 4 nucléotides A, T, G ou C. On fera
donc, en
parallèle, 4 séries de copies. Dans chaque série, toutes les copies seront
interrompues
derrière un seul type de nucléotide ; par exemple, toutes les copies
intermédiaires
d'une série seront terminées par un A. En outre, le composé provoquant
l'interruption
est fluorescent pour pouvoir être détecté automatiquement à l'aide d'un système
optique qui balaye le bas du gel d'électrophorèse dans les séquenceurs
automatiques. Le
signal obtenu est interprété par un programme informatique qui reconstituera la
séquence originale du fragment d'ADN analysé
5(
*
)
.
La rapidité du séquençage :
Les centres
publics ou privés de séquençage utilisent des outils de plus en plus
perfectionnés,
des séquenceurs à haut débit. Les deux séquenceurs les plus rapides sont
actuellement :
-
MegaBace 1000
, de la société américaine Molecular
Dynamics-Amersham
Pharmacia-Biotech qui permet de séquencer 96 échantillons par réaction et
1 100 par 24 heures (les premiers appareils ont été installés en
Europe en
août 1997).
-
Abi Prism 3700
, de la société américaine Perkin Elmer Applied
Biosystems, qui permet de séquencer 96 ou 384 échantillons par réaction,
et 760 à
1240 par 24 heures (les premiers appareils ont été installés en Europe en
janvier
1999).
Il peut être également intéressant, pour des raisons de rentabilité et de
flexibilité
de coupler plusieurs séquenceurs. La firme canadienne Visible Genetics a mis au
point le
Virtual DNA Sequencer. Ce système organise une connexion en réseau de plusieurs
séquenceurs automatisés rapides. La centralisation dans l'ordinateur des données
d'analyse issues de chacun de ces appareils permet de faire fonctionner
l'ensemble comme
un seul séquenceur très rapide.
L'interprétation des séquences :
La séquence d'un fragment d'ADN contient une série d'informations qu'il faut identifier et interpréter. Les éléments de séquences les mieux connus correspondent aux gènes, délimités par des signaux de début et de fin. Ces gènes ne s'expriment pas tous en permanence dans une cellule. Leur expression est régulée par des éléments de contrôle, situés dans leur voisinage, qui augmentent ou diminuent leur niveau d'expression en fonction du besoin. Grâce à des programmes informatiques, l'interprétation des séquences permet le repérage des gènes, des éléments de contrôle et de leurs relations.
La cartographie génétique :
Elle constitue une autre façon d'étudier les génomes. Compte tenu de la complexité des procédés déjà exposés (cartographie physique, séquençage, interprétation des séquences), il est évident que des approches différentes peuvent se révéler intéressantes pour la connaissance des génomes. On peut, sans disposer d'un séquençage complet ou de cartes physiques très précises, étudier un caractère physiologie ou pathologie particulier. On fait alors appel aux méthodes de cartographie génétique pour identifier les gènes qui contrôlent ces caractères. Ces méthodes consistent à détecter directement au niveau de l'ADN les polymorphismes, c'est-à-dire les variations génétiques différenciant un individu d'un autre.
1.1.1.2. L'état des connaissances
1.1.1.2.1. La soudaine accélération du séquençage du génome humain
Le projet
international " Génome Humain " a été lancé dès 1990 avec,
aux
États-Unis, un budget de 18 milliards de francs sur quinze ans.
Ce programme se fondait notamment sur une carte physique, localisant
géographiquement les
gènes sur la molécule d'ADN : elle avait été dressée à 70 % par le
Professeur Daniel COHEN, chercheur au Centre d'études du polymorphisme humain
(CEPH) et
au Généthon, le laboratoire de l'Association française contre les myopathies
(AFM),
puis achevée avec le concours des chercheurs de l'Institut de technologie du
Massachusetts (MIT).
Il se fondait également sur une carte génétique, situant les gènes selon leur
fonction, établie par le Professeur Jean WEISSENBACH, actuellement directeur
général du
Centre national français de séquençage.
Afin que les travaux soient menés le plus rationnellement possible, tous les
responsables
des centres nationaux d'étude du génome se réunirent aux Bermudes en 1996 et
procédèrent au " partage " du génome afin de répartir son
séquençage, chaque équipe étant chargée d'un chromosome entier ou d'une région
particulière du génome.
La France, à cette époque, se montra hésitante et ce n'est qu'en 1998 qu'elle
se vit
confier le séquençage des chromosomes 3 et 14 (elle a ensuite renoncé au
séquençage du chromosome 3).
Jusqu'en 1998, dans le cadre du programme international public Génome Humain,
une
fraction de 10 % des gènes a été séquencée.
Or, depuis un an, ce programme fait l'objet d'une accélération fulgurante. Le
15 mars 1999, les Instituts nationaux de la santé américaine (NIH) ont
annoncé que
le projet international de décryptage du génome humain avait achevé avec succès
sa
phase d'essai et que le financement du séquençage de l'ADN à grande échelle
était
décidé. Les échéances annoncées sont proches : un an pour l'ébauche
globale,
prévue pour le printemps 2000 et trois à quatre ans pour l'aboutissement d'un
séquençage définitif de grande qualité (moins d'une erreur tous les cent mille
nucléotides), prélude à la compréhension des protéines sécrétées.
Pour parvenir à ces résultats, les NIH ont réparti 493 millions de francs
entre
les trois plus grands groupes publics impliqués dans le séquençage :
- Whitehead Institute à Cambridge (Massachusetts)
- Washington University School of Medicine à Saint-Louis (Missouri) ;
- Baylor College of Medicine à Houston (Texas).
L'institut américain du génome, du département de l'énergie (Joint Genome
Institute of
the US Department of Energy, à Walnut Creek, California) a associé ses efforts
à ceux
de ces trois grands centres.
Dans le même temps, la fondation britannique Wellcome Trust a annoncé le
versement, dans
les douze mois à venir, d'une somme de 460 millions de francs au Centre
Sanger
(Royaume-Uni). Le Centre Sanger a été fondé en 1993 par le Wellcome Trust (la
plus
grande association mondiale pour la recherche médicale) et le Medical Research
Council.
C'est l'un des centres les plus productifs du monde ; il devrait produire,
à lui
seul, un tiers du séquençage du génome humain en 2001.
Les raisons de cette brusque accélération du décryptage du génome humain sont
de deux
ordres.
- Tout d'abord, le progrès technique a permis d'accroître les vitesses de
séquençage : en 1992 les chercheurs identifiaient un million de bases par
an. À ce
rythme, il aurait fallu près d'un siècle pour achever le séquençage du génome
humain.
À l'heure actuelle, la vitesse de séquençage est dix fois plus élevée, grâce à
des
appareils tels que les Mega Bace 1000 ou les Abi Prism 3700. Non
seulement on
peut séquencer beaucoup plus vite, mais aussi beaucoup moins cher : le
coût de la
base séquencée est passé de 5 dollars en 1990 aux États-Unis à
50 cents
aujourd'hui.
- Par ailleurs, cette accélération est liée à la
" course "
récemment née entre la recherche publique internationale et le secteur privé.
Le généticien américain Craig VENTER a fait sensation en annonçant, le
9 mai
1998 : " J'ai un plan pour achever de façon substantielle le
séquençage
du génome humain dans les trois ans à venir ". Pour ce faire, le
fondateur de
l'Institut de recherche génomique (TIGR à Rockville, Maryland) a créé une
société
privée, la firme Celera Genomics, en s'associant au géant américain de
l'électronique,
Perkin Elmer. Celera Genomics s'est équipée de 230 séquenceurs
Abi-Prism 3700
dont le prix unitaire est de 300 000 dollars.
Puis la société Incyte Genetics créée en août 1998 par Incyte Pharmaceuticals,
pour
concurrencer Celera Genomics, a annoncé qu'elle séquencerait et
cartographierait le
génome humain d'ici 2001. Elle utilise des séquenceurs Mega Bace 1000 et a
déjà
établi de fortes relations commerciales avec plus de vingt grandes compagnies
pharmaceutiques à travers le monde pour leur vendre les informations issues de
ses
recherches.
Cette émergence du secteur privé explique en grande partie le récent et massif
engagement de la recherche publique internationale : il existe de grandes
différences entre les objectifs des uns et des autres.
1.1.1.2.2. La divergence des approches publiques et privées dans le séquençage du génome humain
1.1.1.2.2.1. Deux logiques de recherche différentes
Les sociétés
privées ont une stratégie de séquençage aléatoire sans cartographie préalable
qui se
veut rapide et puissante. Toutefois leur technique peut éventuellement se
révéler peu
efficace et en tout état de cause elle produit un séquençage " à
trous ".
Le séquençage aléatoire rapide sans cartographie préalable n'a jusqu'alors
démontré
son efficacité que sur des génomes simples et pourrait marquer ses limites pour
des
génomes plus grands.
Aussi, Celera Genomics, avant de décrypter le génome humain, va tester sa
méthode en
séquençant le génome de la mouche du vinaigre
Drosophila melanogaster
(120 millions de bases). Si cette technique échoue pour la drosophile,
elle a peu de
chance de réussir pour le génome humain, infiniment plus important et plus
complexe. Et
même si elle donne satisfaction pour la drosophile, elle ne sera pas forcément
transférable pour le génome humain.
Quant à Incyte Genetics, elle a déjà testé sa technique en menant à bien le
séquençage complet d'une levure (
Candida albicans
: 17 millions de
bases).
Pour cette société se posera aussi le problème de la taille du génome humain
composé
de 3 milliards de bases.
De toute façon, ce type de séquençage est effectué fragment par fragment, chacun
d'entre eux comprenant environ 500 bases. Cela aboutit à une séquence très
morcelée du génome constituée de dizaines voire de centaines de petits
segments, non ou
mal positionnés sur les cartes existantes.
Pour reconstituer dans ce puzzle des morceaux cohérents correspondant aux
séquences des
gènes, un gigantesque travail de réassemblage restera à faire. Il manquera
inévitablement des morceaux importants, le résultat étant un séquençage
" à trous ".
Selon Jean WEISSENBACH,
" On ne peut pas croire que, comme le dit
Craig
VENTER, les " trous " restants au terme de son travail ne
représenteront que moins de 10 millièmes du génome humain. Son programme
comporte
de nombreux mystères d'un point de vue méthodologique. En réalité, tout laisse à
penser que cette équipe entend réaliser un " écrémage " lui
permettant de trouver toute une série de choses intéressantes à breveter
rapidement "
6(
*
)
.
Au contraire, les laboratoires du programme international public isolent des
fragments de
chromosomes et les ordonnent entre eux avant le séquençage, ce qui nécessite, au
préalable, une cartographie fine des chromosomes. De plus, ils procèdent à dix
vérifications pour chaque séquence, quand les équipes du secteur privé n'en
font que
trois. Cette technique est moins rapide mais le succès est assuré, ainsi que la
qualité
des résultats obtenus
7(
*
)
.
Toutefois, il semblerait que dans un premier temps les chercheurs publics aient
décidé,
en ce qui concerne le génome humain, de procéder de manière prioritaire au
séquençage
aléatoire à faible profondeur en continuant toutefois à ordonner les clones de
fragments d'ADN sur une carte de préséquençage.
Cette technique permettrait d'obtenir pour le printemps 2000 l'ébauche globale.
Cette
étape sera bien entendu immédiatement suivie par un séquençage définitif de
grande
qualité. Retenir cette solution d'un séquençage en deux temps à pour avantage de
fournir rapidement des données partielles mais suffisantes pour des projets de
recherche
de gènes responsables de maladies dans des régions données. L'offre de ces
données,
par le secteur public est essentielle car, là encore, les stratégies du privé
et du
public divergent.
1.1.1.2.2.2. Deux logiques d'accès aux connaissances
Les
chercheurs du public craignent que les grandes entreprises privées de génomique
ne
confisquent l'information, alors que l'accès à celle-ci est un élément de base
indispensable pour la communauté scientifique.
En 1992, Craig VENTER, alors chercheur aux NIH, avait déposé des centaines de
demandes
de brevets sur des gènes dont l'intérêt biologique n'était pas prouvé. Devant la
polémique internationale déclenchée par cette initiative, les NIH avaient
renoncé,
sans que les règles du jeu de la brevetabilité aient été pour autant
clarifiées.
Aujourd'hui Craig VENTER et ses associés ont l'intention de créer une banque de
données
sur le génome humain dont personne ne sait exactement quelles seront les
conditions
d'accès.
Par ailleurs, l'Office américain des brevets a accordé en mars 1999 à la société
Incyte le premier brevet sur des marqueurs d'expression du génome (il s'agit de
portions
d'ARN messagers, molécules indispensables à l'expression des gènes, appelées
" Expressed
Sequence Tags "
EST).
Or, la position des chercheurs publics est toute autre. Selon des accords
internationaux
issus des réunions des centres d'études du génome aux Bermudes, toute portion
d'ADN
séquencée à l'aide de fonds publics doit être publiée dans la littérature
scientifique et diffusée rapidement sur Internet afin d'être disponible pour la
communauté des chercheurs.
De plus, tout récemment, les cinq plus grands laboratoires publics de séquençage
(Whitehead Institute, Washington University School of Medicine, Baylor College
of
Medicine, Joint Genome Institute, Sanger Centre) se sont engagés à rendre
publics leurs
résultats dans un délai de vingt-quatre heures :
" Par cet
effort
majeur de financement public, nous permettons que les résultats restent dans le
domaine
public, en libre accès pour les chercheurs qui mettent au point les traitements
du futur.
C'est crucial pour en recueillir de manière efficace les vrais bénéfices
médicaux "
a indiqué Michael MORGAN, directeur du Wellcome Trust
Genome
Campus
8(
*
)
.
1.1.1.3. Les résultats déjà obtenus ou attendus et l'intérêt de ces résultats
1.1.1.3.1. Le génome humain
Bien que son
séquençage complet ne soit pas réalisé, les chercheurs ont déjà identifié de
très
nombreux gènes impliqués dans les processus pathologiques. Il est très
difficile d'en
présenter une liste exhaustive. On ne peut que citer les découvertes les plus
récentes :
Une équipe française vient de démontrer que le cancer du sein de type
médullaire
est une entité biologique dans laquelle on trouve 100 % de mutation du gène
p 53, déjà suspecté précédemment d'avoir un rôle important dans le
processus
cancéreux.
L'unité de génétique des déficits sensoriels de l'Institut Pasteur vient
de
mettre en lumière le fait que plus de la moitié des surdités héréditaires de
l'enfant
sont dues à des mutations dans un gène unique, le DFNB1.
Aujourd'hui, on estime à 1 500 le nombre de gènes responsables de maladies
strictement génétiques identifiés. Mais il est évident que des milliers d'autres
gènes, en partie identifiés, sont impliqués dans des pathologies plus courantes
(cancer, diabète, maladies cardio-vasculaires ou neurologiques).
Par exemple, au 1
er
mars 1999, 487 gènes de maladies ont
été
localisés et 77 gènes de maladies ont été identifiés avec l'aide de l'AFM
et/ou
de Généthon.
Ces gènes se répartissent ainsi :
|
- Maladies neurologiques et psychiatriques |
28 % |
|
- Malformations
congénitales
|
13 % |
|
- Maladies oculaires |
11 % |
|
- Maladies neuromusculaires |
8 % |
|
- Maladies métaboliques et endocriniennes |
6 % |
|
- Maladies systémiques |
5 % |
|
- Maladies cardiovasculaires |
5 % |
|
- Maladies dermatologiques |
5 % |
|
- Maladies ostéo-articulaires |
4 % |
|
- Surdité |
4 % |
|
- Maladies cancéreuses |
4 % |
|
- Maladies urogénitales |
3 % |
|
- Maladies de l'appareil digestif |
3 % |
|
- Maladies hématologiques |
1 % |
1.1.1.3.2. Le génome d'agents responsables de maladies
Si l'on excepte
celui des très petits virus, le premier séquençage complet remonte à 1995.
C'est celui
d'
Haemophilus
influenzae
(1,93 million de bases), suivi en
1996 par
celui de
Mycoplasma genitalium
(9,58 millions de bases). Puis, à
partir de
1996, ont été séquencés les génomes de :
-
Mycoplasma pneumoniae
(810 000 bases) ;
-
Helicobacter pylori
(1,66 million de bases), tenu depuis peu
pour
responsable de l'ulcère de l'estomac ;
-
Escherichia coli
(4,6 millions de bases) ;
-
Borrelia burgdorferi
(1,44 million de bases), agent
pathogène de la
maladie de Lyme ;
-
Mycobacterium tuberculosis
ou bacille de Koch. Le génome de ce
bacille,
composé de 4,41 millions de bases formant 4 000 gènes, a fini d'être
séquencé en juin 1998 par une équipe de 42 chercheurs dirigés par le
Professeur
Stewart COLE, chef de l'unité de génétique moléculaire à l'Institut Pasteur de
Paris
et par Bart BASSEL du Centre Sanger au Royaume-Uni.
On peut espérer d'autres découvertes dans un avenir assez proche :
- Des biologistes américains ont réalisé la carte chromosomique de la
bactérie
responsable de la syphilis et vont commencer son séquençage.
- Les génomes d'agents pathogènes tels que
Streptococcus pneumaniae
(2,2 millions de bases) et
Rickettsia prowazekii
(1,1 million
de bases)
sont à l'étude, de même que ceux de
Vibrio cholerae
(2,5 millions
de
bases), responsable du choléra et
Plasmodium falciparum
, responsable du
paludisme.
- Les génomes dont l'étude donnera des résultats un peu plus tard sont ceux
d'agents pathogènes responsables de maladies malheureusement bien
connues :
Listeria
monocytogènes
,
Candida albicans
,
Legionella pneumophila
(maladie du
légionnaire),
Mycobacterium leprae
(lèpre),
Neisseria gonorrhoeae
(gonococcie),
Staphylococcus aureus
(infections graves, notamment la
septicémie),
Trypanosoma
brucei rhodosiense
(maladie du sommeil)
Yersinia pestis
(peste).
Les génomes des organismes eucaryotes 9( * )
Là encore, il
est impossible d'être exhaustif mais l'on peut citer notamment les séquençages
sur
lesquels travaille le Génoscope d'Évry :
- l'
Arabidopsis
thaliana
(arabette, petite crucifère de la
famille du
colza et du chou) ;
- Le
Tetraodon fluviatilis
, un poisson à génome
" compact " c'est-à-dire débarrassé de l'ADN
" superflu " (non codant).
Il convient également d'évoquer la levure
Saccharomyces cerevisae
, le
premier
organisme eucaryote dont le génome ait été séquencé, en 1996, grâce à un
programme
international placé sous la responsabilité du professeur A. GOFFEAU de
l'Université de Louvain en Belgique.
Enfin, il faut souligner l'importance exceptionnelle d'un récent
succès : le
séquençage du génome d'un animal a été achevé au début de l'année 1999 ;
c'est celui du ver
Caenorhabditis elegans
.
Ses 97 millions de bases forment plus de 19 000 gènes dont
12 000
encore inconnus. Ce travail considérable a été réalisé par l'Université
Washington
de Saint-Louis et le Sanger Center du Royaume Uni.
L'intérêt de ces séquençages
En ce qui
concerne les génomes des bactéries pathogènes, l'utilité de leur décryptage est
évidente. Un exemple en a été fourni très récemment avec le séquençage du
Mycobacterium
tuberculosis
ou bacille de Koch. La tuberculose connaît aujourd'hui une
inquiétante
recrudescence et tue chaque année plus de 3 millions de personnes dans le
monde, les
vaccins demeurant bien faibles devant la maladie. Or le séquençage du
Mycobacterium
tuberculosis
a permis, en octobre 1998, à des chercheurs de l'unité de
génétique
mycobactérienne de l'Institut Pasteur de Paris d'identifier un gène responsable
de la
virulence du bacille de la tuberculose. Appelé
erp
, ce gène commande la
production d'une protéine dont le bacille a besoin pour se multiplier dans les
cellules
qu'il infecte. Inactiver ce gène pourrait permettre d'atténuer la virulence du
bacille
et de produire de nouveaux vaccins, en particulier des vaccins vivants
atténués.
D'une façon plus générale, il est certain que connaître l'ensemble des gènes et
donc
des protéines d'un organisme pathogène est un préalable indispensable à la
compréhension des mécanismes pathologiques induits par ces espèces.
" Cette connaissance devient cruciale à l'heure où l'on assiste à
une
généralisation du phénomène de résistance aux antibiotiques et aux moyens de
lutte
contre les parasites. Il devient essentiel d'inaugurer de nouvelles voies de
lutte contre
les pathogènes. On peut même penser qu'en raison de leur extraordinaire capacité
d'évolution, de nouvelles variétés insensibles aux nouveaux agents
anti-pathogènes ne
vont cesser d'apparaître en réponse à l'utilisation de ces agents. La
connaissance du
génome permettra néanmoins de connaître rapidement les changements clés chez ces
variants et de prendre des mesures appropriées "
10(
*
)
.
- En ce qui concerne les génomes d'organismes eucaryotes, leur intérêt réside
essentiellement dans les possibilités de comparaison avec le génome humain
qu'ils
offrent. L'utilité de génomes d'espèces utilisées comme modèles expérimentaux,
comme
la souris, dont la physiologie est proche de l'homme, est évidente. Mais les
génomes
d'organismes très éloignés de l'homme peuvent être très intéressants également.
Si l'on prend l'exemple de la levure
Saccharomyces cerevisiae
, on
constate que
certaines protéines humaines ont une séquence en acides aminés qui ressemble de
façon
significative à celle d'une protéine de levure : ces protéines sont
" homologues ". Selon les scientifiques, près de 40 %
des gènes
connus pour être impliqués dans une maladie génétique humaine ont un homologue
chez la
levure
11(
*
)
. Mais l'on ignore souvent le rôle des
protéines que codent ces gènes humains. La levure peut alors fournir une
indication sur
la fonction des protéines. Le schéma de recherche est le suivant : le gène
responsable d'une maladie génétique humaine est identifié ; la fonction de
la
protéine qu'il code est inconnue ; un homologue du gène existe chez la
levure ; on utilise alors la levure comme une " éprouvette
biologique " car il est aisé de détruire ou remplacer un gène précis
dans un
organisme tel que la levure et cela permet de commencer à décrypter le rôle et
le
fonctionnement des gènes dont l'équivalent humain provoque une maladie
génétique.
Cette méthode a, par exemple, été utilisée pour étudier l'ataxie de Friedreich
(maladie due à une dégénérescence des neurones entraînant des handicaps
physiques
graves et une cardiomyopathie).
De même, le séquençage du génome du
Caenorhabditis elegans
aura
des
conséquences importantes, toujours grâce au caractère homologue de nombreux
gènes
humains avec ceux d'espèces bien différentes ; grâce à des années de
recherche
intensive, la fonction de nombreux gènes du ver est déjà connue. Les
possibilités
d'études comparatives seront donc nombreuses.
En ce qui concerne le génome humain, l'utilité de son décryptage est
évidente,
ainsi que le rappelle le Professeur Jean WEISSENBACH.
"
Plus de 6 000 maladies d'origine clairement
génétique,
conséquence d'un défaut au niveau d'un gène, ont été répertoriées à ce
jour.
Ces maladies génétiques souvent incurables sont cependant rares, elles
affectent un
nouveau-né sur 1 000 à 100 000, voire moins. Depuis une dizaine
d'années, les
gènes responsables des maladies génétiques les plus fréquentes sont
progressivement
identifiés.
Ils constituent le point de départ à une approche rationnelle de la thérapie.
Cette
identification est considérablement facilitée lorsqu'on dispose de la séquence
de l'ADN
de la région dans laquelle le gène a pu être localisé. Cette localisation,
elle-même
encore très laborieuse il y a quelques années, s'est considérablement améliorée
grâce à la cartographie du génome humain, préalable indispensable au
séquençage. À
ce jour, près de 1 500 gènes responsables de maladies génétiques ont
été
identifiés.
À côté de ces maladies strictement génétiques, d'autres pathologies beaucoup
plus
communes comme le diabète, les maladies cardiovasculaires, neuropsychiatriques,
etc., ont
elles aussi une composante génétique
dans leur origine en général
complexe. La
recherche des gènes prédisposant à ces pathologies fréquentes devrait permettre
de
disposer de nouvelles cibles pour
les médicaments du futur. Ces gènes
représentent donc des enjeux majeurs pour l'industrie pharmaceutique, et la
plupart des
grands groupes internationaux se sont lancés dans de grands programmes visant à
identifier les facteurs génétiques prédisposant aux pathologies communes. Ces
travaux
n'ont pas encore abouti à des découvertes majeures mais la séquence complète du
génome humain devrait aussi considérablement faciliter la recherche de ces
gènes.
Le diagnostic de maladies et de prédispositions génétiques reposera lui aussi
sur la
séquence du génome. À ce jour, cette activité, qui a bénéficié de nombreux
progrès
technologiques, est déjà largement répandue. La connaissance de la séquence
complète
du génome va cependant provoquer une véritable explosion dans le domaine du
diagnostic
génétique dans le but d'orienter de manière beaucoup plus ciblée les
traitements et
éventuellement de
mettre en place de nouveaux modes de
prévention
12(
*
)
".
CARTE DU
GÉNOME HUMAIN
Localisation sur les chromosomes de certains gènes
dont les mutations, associées ou non à d'autres mutations et mécanismes, sont
impliquées dans l'apparition, l'évolution et la gravité de certaines
maladies
|
Chromosome 1
Cataracte
congénitale
|
|
|
|
Chromosome 2
Glaucome
congénital
|
|
|
|
Chromosome 3
Prédisposition
à la schizophrénie
|
|
|
|
Chromosome 4
Nanisme
(forme achondroplasie, hypochondroplasie)
|
|
|
|
Chromosome 5
Déficit
de l'attention et hyperactivité
|
|
|
|
Chromosome 6
Prédisposition
à la schizophrénie
|
|
|
|
Chromosome 7
Cancer du
côlon (non polyposique)
|
|
|
|
Chromosome 8
Épilepsie
précoce
|
|
|
|
Chromosome 9
Albinisme
oculo-cutané
|
|
|
|
Chromosome 10
Fente
labiopalatine
|
|
|
|
Chromosome 11
Prédisposition
au diabète sucré insulino-dépendant
|
|
|
|
Chromosome 12
Prédisposition
aux maladies inflammatoires de l'intestin
|
|
|
|
Chromosome 13
Surdité
dominante
|
|
|
|
Chromosome 14
Prédisposition
à l'atopie (eczéma)
|
|
|
|
Chromosome 15
Susceptibilité
à la dyslexie
|
|
|
|
Chromosome 16
Psychose
maniaco-dépressive
|
|
|
|
Chromosome 17
Prédisposition
au cancer du sein (gène BCCR)
|
|
|
|
Chromosome 18
Psychose
maniaco-dépressive
|
|
|
|
Chromosome 19
Migraine
hémiplégique
|
|
|
|
Chromosome 20
Déterminant
quantitatif de la stature
|
|
|
|
Chromosome 21
Maladie
d'Alzheimer
|
|
|
|
Chromosome 22
Cardiopathies
congénitales
|
|
|
|
Chromosome X
Prédisposition
à la schizophrénie
|
|
|
|
Chromosome Y
Facteur
déterminant dans la régulation des gènes contrôlant le développement des
testicules ; dysgénésie gonadique (femme XY)
|
Documentation AFM.
1.1.2. LA BIO-INFORMATIQUE
La
bio-informatique, combinaison de l'informatique et de la biologie, est la
science sans
laquelle aucune des découvertes évoquées ci-dessus ni aucune de celles qui en
découleront n'aurait été possible. Domaine fondateur de la génomique, la
bio-informatique est constituée de l'ensemble des concepts et des techniques
nécessaires
à l'acquisition et à l'interprétation de l'information génétique. Elle seule
permet
de gérer cette information, qui est quantitativement considérable.
La bio-informatique, véritable clé de la génomique, en est un outil
indispensable : à partir d'une séquence d'ADN nouvellement identifiée,
elle permet
de retrouver les séquences similaires déjà décrites dans des banques de données,
construire des séquences " virtuelles " issues de leur
assemblage,
déduire quels sont les gènes associés et leur distribution au niveau d'un
organe ou
d'un tissu, établir un lien entre des gènes présents dans une pathologie et la
présence en surabondance d'une certaine protéine in situ, prédire la structure,
et
même la fonction de cette protéine, cible potentielle pour un futur médicament.
La bio-informatique est rendue indispensable par la quantité même
d'informations qu'elle
permet de recueillir en décryptant le génome.
" Le nombre de
données est
tellement important
qu'il est impensable qu'on soit aujourd'hui en
mesure de les
interpréter dans toute leur complexité et ainsi parvenir à les intégrer dans un
schéma global de fonctionnement de la cellule. C'est dire, dans le long terme,
l'importance du pari de la bio-informatique pour tirer le meilleur parti de
l'utilisation
de cette technologie révolutionnaire. "
13(
*
)
1.1.2.1. L'acquisition et l'analyse de l'information génétique
L'activité
de séquençage consiste à transformer de la matière en information. Dans un
premier
temps, il s'agit d'utiliser des programmes informatiques qui, intégrés aux
séquenceurs
permettent de décrypter les fragments d'ADN, de les
" lire ".
Interviennent ensuite des moyens d'analyse intensive de la
séquence :
- Les supercalculateurs :
Des moyens de calculs importants mettant en oeuvre des techniques d'analyse
d'images et de
compression de données permettent non seulement d'organiser la grande quantité
de
données brutes générées quotidiennement, mais aussi de reconstituer la séquence
de
longues régions du génome à partir d'éléments beaucoup plus petits. On est ainsi
amené à reconstituer des puzzles dont chacune des pièces est une séquence que
l'on
comparera à toutes les autres. Pour un génome bactérien entier, par exemple, la
reconstitution du puzzle peut demander plus de
deux cent mille milliards de
comparaisons de caractères
.
Les séquences reconstituées doivent ensuite être comparées à celles déterminées
par
des milliers de chercheurs à travers le monde et stockées dans des bases de
données
internationales : ces comparaisons sont actuellement le meilleur moyen
d'attribuer
une fonction biologique aux séquences.
- L'algorithmique du génome :
C'est ce qui permet le calcul de certaines occurrences, de relations
phylogénétiques, et
la mise en évidence de phénomènes inattendus par l'examen systématique des
données.
Pour la détection et la prédiction des gènes, pour la connaissance des
structures et
des fonctions des protéines correspondantes, il est indispensable, en effet, de
rechercher des événements tels que la présence simultanée de plusieurs motifs
dans une
configuration donnée.
Enfin, l'ultime étape de la connaissance génomique, c'est-à-dire la
découverte
de la structure des protéines passe aussi par l'informatique. Celle-ci permet
en effet,
à partir des données sur les séquences ou motifs de séquences, de reconstruire
la
structure spatiale des protéines. Cette structure en trois dimensions est
indispensable
à la conception de molécules capables d'interagir avec les protéines.
1.1.2.2. L'organisation et la conservation de l'information
C'est ce que
certains appellent l'informatique d'intégration. L'informaticien essaie
d'assister le
biologiste dans l'organisation des résultats et la mise en concordance
d'observations
scientifiques distinctes, qui lui permettent d'émettre une hypothèse et de la
valider
sur de grands ensembles de données. Elle consiste notamment à constituer de
gigantesques
bases de données, à permettre la recherche d'informations ("
data
mining
")
et la mise en évidence de " voisinage " à partir des
connaissances
scientifiques publiées et de banques de données spécialisées.
Par ailleurs, la découverte des séquences génomiques de plus en plus nombreuses
suppose
que soit rapidement assurées, par la constitution de bases de données
" d'archives ", la conservation et bien entendu la mise à
jour des
séquences, ainsi que le classement par niveaux de complexité croissants, des
résultats
de la démarche génomique d'ensemble (séquences, structures, fonctions,
propriétés
physiologiques, etc.).
-
La communication de l'information
Elle suppose la constitution de réseaux d'information cohérents, homogènes en
format et
en qualité permettant, par des échanges nationaux et internationaux, de mettre
en commun
les connaissances, les technologies et les compétences.
À l'heure actuelle les standards d'Internet structurent le développement des
environnements informatiques appliqués à la biologie moléculaire. C'est bien
entendu la
démarche suivie au Génoscope d'Évry :
" Les données produites au Génoscope sont mises à la disposition
des
autres membres de la communauté scientifique internationale. Réciproquement,
le
Génoscope
réactualise journellement les bases des données produites ailleurs dans le
monde,
via Internet. Chaque jour, il met ainsi de l'ordre de plusieurs millions
d'octets de
données nouvelles à disposition sur le
réseau et recueille des millions
d'octets
représentant
les
nouvelles données découvertes par des biologistes du
monde
entier. À cette fin, nous exploitons une connexion au réseau qui permet
d'échanger deux
millions de bits (Mbits) -250 000 octets- par seconde.
14(
*
)
"
1.1.3. LES BIOPUCES
1.1.3.1. Les technologies utilisées
L'hybridation : théorie
Le concept de
biopuce ou puce à ADN date du début des années 1990. Il a été à cette époque
décrit par quatre équipes différentes, en Grande-Bretagne, en Russie et aux
États-Unis. Il repose sur une technologie pluridisciplinaire intégrant la
micro-électronique, la chimie des acides nucléiques, l'analyse d'images et la
bio-informatique. Le principe de fonctionnement de ces puces repose sur un
fondement de la
biologie moléculaire : le phénomène de l'hybridation, c'est-à-dire
l'
appariement
par complémentarité des bases de deux séquences d'ADN
. Par analogie, on
peut
comparer ce mécanisme à celui de la fermeture éclair... En effet, les brins
extraits de
la double hélice d'ADN ont la capacité de reformer spontanément cette double
hélice
dès qu'ils se retrouvent face au brin complémentaire ; les quatre molécules
élémentaires de l'ADN ont la particularité de s'unir deux à deux :
l'
adénine
avec la thymine
,
la cytosine
avec la guanine
.
Le phénomène de l'hybridation permet d'identifier une séquence de nucléotides,
c'est-à-dire l'enchaînement des bases d'un fragment d'ADN en mettant ce dernier
en
présence d'autres brins d'ADN, dont la séquence est connue.
Par exemple, face à des brins d'ADN synthétique représentatifs d'une maladie,
les brins
extraits de l'ADN du patient vont hybrider si le malade est porteur de
l'affection
recherchée.
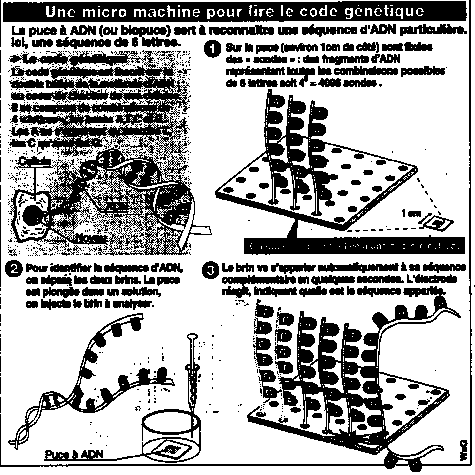
Source : Le Figaro, 13 novembre 1997.
Le problème est " d'offrir " aux brins d'ADN du patient la possibilité d'hybrider avec de nombreux brins d'ADN synthétiques représentant les différents gènes correspondant à une pathologie, sous leurs différentes formes.
Le support
Les brins
synthétiques utilisés, ceux dont on connaît la séquence, s'appellent des
sondes. Les
puces à ADN présentent, par rapport aux méthodes classiques d'hybridation un
avantage
majeur : grâce aux techniques de miniaturisation elles permettent
l'hybridation
simultanée d'un nombre de sondes considérables sur une surface totale utile
inférieure
à 1 cm
2
. Les supports sur lesquels sont fixées les sondes se
présentent
sous la forme de surfaces planes ou poreuses (percées de puits) composés de
matériaux
tels que :
-
le verre
, matériau peu onéreux, inerte et mécaniquement stable.
Il est
notamment utilisé par l'entreprise américaine Affymetrix et le consortium
Genosensor).
La société américaine Protogene (Palo Alto, Californie) avec laquelle les
chercheurs de
l'équipe du Professeur Francis GALIBERT, du CNRS, ont passé des accords de
partenariat
utilise une ingénieuse technique : la petite plaque de verre, substrat de
la puce,
est recouverte d'une grille de Teflon (qui détermine des zones hydrophiles et
hydrophobes).
-
les polymères
sont à la base des techniques développées par
l'équipe
d'Andrei MIRZABEKOV (Argonne, Illinois) ainsi que par les chercheurs de la
société CIS
Bio-international qui utilisent plus précisément le polypyrrole ;
-
le silicium
, couramment utilisé pour la fabrication des puces
électroniques à cause de ses propriétés semi-conductrices (matériau choisi par
plusieurs équipes et notamment par le laboratoire IFOS de l'École centrale de
Lyon -
UMR 5621 du CNRS).
- les métaux, notamment
l'or et le platine
(utilisés par la société
américaine Nanogen et le Consortium Genosensor).
La fixation des sondes
Quel que soit le
support choisi, il est traité pour former un réseau dense et régulier de
microsurfaces
et sur chacune de celles-ci est greffée une sonde d'ADN synthétique. Il existe
deux
méthodes : le transfert de brins d'ADN ou sa synthèse
in situ
.
- La
synthèse préalable
à la fixation des sondes permet de fixer des
sondes
relativement longues, atteignant 40 à 60 bases. Le
transfert
de ces
sondes
sur la puce peut se faire au moyen de micropipettes, de micropointes ou par des
dispositifs de type jet d'encre.
Un autre système, choisi par la société CIS Bio International en collaboration
avec les
chercheurs du CEA à Grenoble (LETI)
15(
*
)
est
l'adressage
électrochimique : la puce est composée d'un support en silicium recouvert,
à
chaque plot d'une mini-électrode en or. Chaque sonde rejoint un plot
particulier lorsque
les mini-électrodes sont mises sous tension sélective.
Les méthodes d'adressage des sondes sont fondamentales : leur performance,
leur
capacité à être automatisées et leur coût de fonctionnement pèseront lourd dans
le
développement de la technologie des puces à ADN.
-
La synthèse
in situ
:
dans ce cas, la construction
des sondes
se fait par dépôt de couches successives des quatre bases de l'ADN sur un
support en
verre. C'est un masque, dont la configuration varie à chaque dépôt d'une
couche, qui
permet aux bases de s'empiler correctement. Avec ce procédé, utilisé par
Affymetrix,
les sondes comportent au maximum 30 bases.
Là encore, la société Protogene innove : elle a mis au point un système qui
permet de déposer sur la grille de téflon de la biopuce, les quatre bases
désirées,
grâce à des injecteurs piézo-électriques, selon une technologie qui s'apparente
à une
synthèse d'ADN classique. C'est l'équivalent d'une imprimante à jet d'encre à
quatre
couleurs (chaque couleur étant en l'occurrence une base : A, G, T, C). Ce
procédé
est extrêmement flexible : il permet de fabriquer indifféremment telle ou
telle
sorte de biopuce, de même qu'une imprimante imprime indifféremment telle ou
telle page,
quels que soient les mots du texte.
L'hybridation : réalisation pratique
Le rôle de
chaque sonde est de reconnaître, dans un mélange appliqué à la surface de la
biopuce,
une séquence d'ADN. Généralement cette séquence est amplifiée par PCR
16(
*
)
puis marquée par fluorescence, préparée en solution
et mise
en contact avec la biopuce.
La phase d'hybridation est réalisée dans une sorte d'incubateur (station
fluidique) et
suivie d'un lavage destiné à débarrasser la puce des cibles nucléiques non
hybridées.
La détection des hybridations
Elle permet de
déterminer à quelle sonde s'est appariée la séquence d'ADN analysée.
Le procédé Affymetrix repose sur l'utilisation d'un scanner qui permet de
repérer les
sondes d'ADN devenues fluorescentes, c'est-à-dire ayant donné lieu à une
hybridation.
D'autres procédés sont à l'étude chez Clinical Micro Sensors (Californie) ou à
l'École centrale de Lyon et se fondent sur les modifications des charges
électriques du
support en silicium où sont fixées les sondes (système GenFet). La mesure de ces
modifications permet de repérer les associations spécifiques entre les sondes
et les
oligonucléotides cibles.
1.1.3.2. Les possibilités d'utilisation
|
LES APPLICATIONS 17( * ) |
||||
|
Diagnostic |
Pharmacie |
Recherche |
Agro-alimentaire |
Environnement |
|
Immuno-essais hormone & cancer |
Criblage médicamenteux " screening " |
Séquençage, analyse de séquences |
Classification |
Microbiologie |
|
Microbiologie ADN antigènes/anticorps |
Mise au point médicaments |
Tri moléculaire |
Contamination |
Polluants |
|
Facteurs aggravants cardio-vasculaire vieillissement |
Pharmacogénomique |
Vision intégrée des voies métaboliques |
|
Plantes |
|
Toxicologie |
|
|
||
|
Aide au suivi des essais cliniques, adaptation de la thérapie |
|
|
|
|
Le séquençage par hybridation (SPH)
Alors que la
méthode enzymatique dite méthode de Sanger peut être considérée comme une
épellation
de la séquence, c'est-à-dire une lecture base par base, le SPH procède par
lecture de
petits blocs. L'analyse porte sur des sous-séquences chevauchantes qui sont
lues et
réassemblées au moyen d'un programme informatique qui reconstitue la séquence
étudiée.
Le séquençage par biopuces constitue une alternative intéressante et plus
précise à
la méthode classique du séquençage en terme d'automatisation et de réduction des
coûts et des durées d'exécution. Cependant tous les problèmes techniques ne
sont pas
résolus.
L'identification de cibles pour la recherche thérapeutique :
La contribution des biopuces au séquençage s'accompagne d'une aide à la compréhension plus fine du génome et de sa régulation. Ainsi, grâce aux puces à ADN, ont été mis en évidence de nouveaux gènes s'exprimant spécifiquement dans le tissu cérébral de l'enfant, ou apparaissant associés à des pathologies inflammatoires rhumatismales ou intestinales. Les biopuces devraient contribuer à l'identification de cibles thérapeutiques pour la recherche pharmaceutique. Elles serviront aussi à déterminer la résistance aux antibiotiques de certaines souches microbiennes pour permettre de mieux lutter contre celles-ci.
La pharmacogénomique :
La
pharmacogénomique consiste à identifier les gènes impliqués dans l'efficacité
(ou
l'inefficacité) d'un produit, ou ses effets indésirables.
Elle conduit à une meilleure compréhension des mécanismes d'action des
médicaments. En
montrant qu'une molécule a sur une cible une action variable, la biopuce ouvre
le champ
des potentiels thérapeutiques. Elle permet aussi d'identifier les effets
secondaires d'un
produit et, lors des essais cliniques, de faire des mesures de toxicité.
Le diagnostic des maladies infectieuses et génétiques
Ce thème est
développé dans le chapitre 1.2.4.
D'un point de vue économique, il est important de noter que le marché mondial de
diagnostic médical par biopuces sera en pleine expansion d'ici quelques années.
En dehors du secteur médical, trois secteurs industriels non négligeables offrent des débouchés aux biopuces :
- L'agro-alimentaire :
le suivi des bactéries productrices de ferments lactiques, détection des
séquences
provenant d'organismes génétiquement modifiés dans les semences.
- L'environnement : l'analyse bactérienne de l'eau de consommation, la
détection
des agents infectieux dans l'alimentation, l'air ou l'eau (
Salmonella
,
Listeria
,
Legionnella
).
- La guerre bactériologique ou chimique : en déterminant par avance les
modifications du fonctionnement génétique des cellules immunitaires
occasionnées par
des agents toxiques, on peut identifier rapidement les produits chimiques
(mercure,
dioxine...) ou bactériologiques (bacille du charbon ou de la diphtérie...)
disséminés
par un éventuel agresseur.
Les multiples possibilités d'utilisation des biopuces expliquent que la
concurrence
règne entre les entreprises de biotechnologie qui souhaitent dominer un
marché très
rentable à moyen terme
.
1.1.3.3. La production des biopuces
|
LES ACTEURS 18( * ) |
||||
|
|
SYNTENI |
AFFYMETRIX |
PROTOGENE |
|
|
Méthode de fabrication |
Dépôt de sondes PCR par robot à pointes |
Synthèse in situ par photolithographie |
Synthèse in situ par microfluidique |
|
|
Nature des sondes |
Sonde PCR : spécifique d'un gène |
Sonde " oligonucléotide " |
Sonde " oligonucléotide " |
|
|
Densité |
10 000 sondes/cm 2 |
100 000 sondes/cm 2 |
100-400 sondes/cm 2 |
|
|
Coût |
$ 4000/puce |
Programme de R & D : 2,2 M$ (1 seul design $450/puce) |
Prog. de R & D : 1,5 M$ (500 designs $300/puce) |
|
|
Applications |
Recherche Pharmacie |
Recherche |
Reséquençage Diagnostic |
|
|
|
40 000 gènes analysés par puce |
1700-7000 gènes analysés par puce |
1 gène analysé par puce (BRCA1, P53, HIV) |
|
Comme dans bien
d'autres domaines biotechnologiques, les États-Unis sont en pointe pour les
puces à ADN.
La société Affymetrix reste incontournable actuellement pour plusieurs
raisons :
Elle a mis au point la première puce à ADN et bénéficie d'une expérience
technologique certaine ;
Elle a protégé ses différentes techniques, -notamment de dépôt de sondes
sur la
puce et de séquençage par hybridation- par d'innombrables brevets. Ses
concurrents
devront donc inévitablement la combattre
19(
*
)
,
s'allier
avec elle (c'est la stratégie de Bio-Mérieux) ou tenter de la contourner en
utilisant
des techniques différentes (c'est la solution adoptée par Protogene).
Le fait que Glaxo-Welcome détienne 34 % du capital d'Affymetrix lui
confère
une réelle solidité économique.
Elle commercialise aussi les scanners de lecture automatique des biopuces
(près de
700 000 F le lecteur) à ceux qui achètent ses biopuces et constituent en
quelque
sorte une clientèle captive.
Les biopuces sont aussi produites par d'autres sociétés américaines, de moindre
importance mais parfois associées à de grands groupes de pharmacie ou
d'instruments
scientifiques : Synteni (rachetée par Incyte Pharmaceuticals à la fin de
1997)
Hyseq (associée à Perkin Elmer), Molecular Dynamics, Nanogen, ...
En Europe, quelques équipes développent le procédé des puces à ADN :
- l'Institut Engelhardt de Moscou travaille avec les groupes américains
Motorola et
Packard Instrument, ainsi qu'avec le Laboratoire national d'Argonne (Illinois).
- Le centre allemand de cancérologie de Heildelberg collabore avec l'Université
danoise
de Copenhague.
- En Angleterre vient de voir le jour la société Oxford Gene Technology, qui
développe
une technique particulière de fixation des fragments d'ADN sur les puces.
- En France, la société CIS-Bio-International (Saclay) étudie une technologie
spécifique en collaboration avec le LETI (Laboratoire d'électronique, de
technologie et
d'instrumentation) et le DRFMC (département de recherche fondamentale de la
matière
condensée) du CEA (Commissariat à l'énergie atomique) à Grenoble.
Aujourd'hui, le LETI développe un nouveau programme : AMIGO, exposé dans la
deuxième partie du présent rapport.
1.1.4. LA CHIMIE COMBINATOIRE ET LE CRIBLAGE À HAUT DÉBIT
" L'apparition
de nouvelles technologies a bouleversé la recherche de nouveaux médicaments
dans sa
phase initiale. Celle-ci inclut tout d'abord la synthèse et l'isolement de
nouvelles
molécules puis leur essai sur des systèmes biologiques permettant de
présupposer d'un
intérêt thérapeutique éventuel. Cette phase était classiquement longue et
pénible.
La synthèse chimique relevait d'un art difficile ; au départ, le choix
d'une
structure de base se faisait sans guide. Les essais sur les animaux entiers ou
les organes
isolés étaient longs et complexes. Au total, malgré des progrès au fil des
années, le
processus relevait plus de la " pêche à la ligne " que de la
démarche rationnelle. Trois approches ont profondément transformé cette
recherche.
1.
Les techniques conformationnelles
La théorie des récepteurs postule que c'est l'union de la molécule de
médicament avec
une macromolécule (une protéine en général) qui est à l'origine de l'effet
pharmacodynamique et plus généralement de la réponse thérapeutique. Cette union
est
fortement spécifique : seules quelques molécules privilégiées en sont capables.
On dit
que le médicament est comme " la clé dans la serrure ". On
sait
maintenant déterminer la conformation dans l'espace des protéines, notamment
grâce à
la radiocristallographie aux rayons X, donc celle des récepteurs. On peut donc
prévoir
quelles structures devront présenter les molécules pour pouvoir s'unir à eux.
Cette
recherche est aidée par les programmes informatiques qui permettent de
visualiser les
molécules et de les faire tourner dans l'espace (conception assistée par
ordinateur).
Bien que hautement sophistiquée et évidemment plus ardue que ces quelques lignes
pourraient le laisser croire, cette approche permet de ne plus s'en remettre au
hasard
dans la recherche des séries chimiques intéressantes. On voit, cependant, qu'il
est
indispensable de connaître au départ le récepteur pertinent, c'est-à-dire
d'avoir une
hypothèse physio-pathologique et d'avoir été capable d'identifier et d'isoler la
protéine qui le porte. Là aussi, des progrès décisifs ont été faits dans
l'isolement
des protéines et, mieux -encore, dans le repérage et le clonage des gènes qui
commandent leur synthèse.
2.
La chimie combinatoire
Il est désormais possible de synthétiser en une seule opération plusieurs
centaines de
molécules c'est ce que l'on appelle la chimie combinatoire. On part de la
structure de
base déterminée a priori comme il vient d'être dit et on génère systématiquement
toutes les variations possibles en greffant des radicaux chimiques, des chaînes
latérales, en modifiant le squelette, etc. Ceci se fait non plus étape par
étape, mais
en mettant en présence les réactifs nécessaires. On obtient ainsi d'un seul coup
plusieurs centaines de molécules. Toutes les opérations, synthèse, isolement et
identification, sont miniaturisées et robotisées. Le gain de temps et
l'abaissement des
coûts sont considérables. On peut ainsi constituer une bibliothèque de plusieurs
milliers de dérivés en quelques mois.
3.
Le criblage à haut débit
Le problème est alors d'identifier parmi toutes ces molécules celles qui sont
pourvues
des propriétés biologiques les plus intéressantes. Le gain de temps et
l'augmentation
de la productivité apportés par la chimie combinatoire l'auraient été en vain
si la
productivité de cette phase de repérage appelée " criblage "
n'avait pas été aussi améliorée. Aux essais longs et limités de la pharmacologie
expérimentale classique, a succédé une technique qui permet d'essayer dans le
minimum
de temps des milliers de molécules.
Le test consiste à mette en présence la substance à tester et un système
biochimique
(une enzyme par exemple) et de mesurer l'importance de la réaction éventuelle.
L'essai
peut être fait simultanément avec un grand nombre de systèmes, de
significations très
diverses. En fait, tout est miniaturisé et robotisé. Les opérations se
déroulent sans
intervention humaine et les résultats sont lus sur l'imprimante.
Tout dépend de ce que l'on met dans les tubes et, une fois de plus, on ne
trouvera que ce
que l'on cherche. C'est dire l'importance des hypothèses physiopathologiques.
Les
systèmes biologiques testés ne sont pas indifférents : ce sont ceux dont
on pense
qu'ils interviennent de manière cruciale dans le déterminisme de la maladie. On
voit
donc bien que cette recherche appliquée, dont les progrès sont si remarquables,
est
totalement tributaire de la recherche fondamentale d'amont.
L'opération finale est celle du choix. La plupart du temps, toutes les molécules
intéressantes ne peuvent pas passer en développement. Il faut donc sélectionner
les
plus prometteuses, compte tenu de leurs résultats aux tests, mais aussi d'autres
considérations comme leur facilité de synthèse ou leur pharmacocinétique. Si les
opérations répétitives ont été automatisées, l'inventivité de l'esprit humain
intervient donc toujours dans les choix des hypothèses, des tests et in fine des
molécules. "
( 20( * ) )
1.1.4.1. Chimie combinatoire et constitution de chimiothèques
La chimie combinatoire
Le principe de la chimie combinatoire est d'accroître les chances de trouver une molécule active, un candidat-médicament, en multipliant le nombre de molécule étudiées 21( * ) .
La synthèse
C'est une sorte
de jeu de construction qui permet de synthétiser de très nombreuses molécules
par
combinaison. Les chimistes, en cela, imitent la nature qui, à partir de quelques
molécules élémentaires, a construit toute la variété des gènes et des
protéines.
Au cours des années 1980, les immuno-chimistes cherchaient à établir la
structure des
fragments des protéines virales (peptides viraux) qui déclenchent les réactions
du
système immunitaire d'un organisme infecté par un virus. Pour synthétiser de
nombreux
peptides de 5 à 12 acides aminés, ils ont mis au point une méthode appelée
synthèse en parallèle.
La synthèse en parallèle
Dans la
synthèse en parallèle, une petite bille de polystyrène est fixée à une tige de
polyéthylène ou
" pin "
. Chacun des puits de la
plaque
contient un seul acide aminé.
On plonge tous les pins dans les puits, et l'acide aminé présent dans chaque
puits se
greffe sur la bille immergée, par l'intermédiaire d'un
" bras " (le
cylindre). Puis on change les acides aminés contenus dans les puits, et les
billes sont
à nouveau immergées. Le nouvel acide aminé se lie au premier, formant un
dipeptide.
Après
n
étapes, on obtient un oligopeptide qui est libéré dans le
puits
après rupture du bras qui le reliait à la bille de polystyrène.
Par cette méthode, on obtient autant de peptides différents qu'il y a de puits,
et ces
molécules sont formées par la succession des acides aminés qui ont été
successivement
introduits dans les puits.
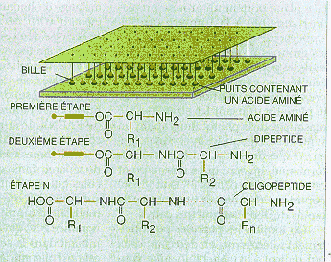
(22( * ))
La méthode
combinatoire est dérivée de cette synthèse mais elle est beaucoup plus
puissante car
elle produit un nombre de molécules croissant exponentiellement à chaque étape
de la
synthèse. Il s'agit alors d'une synthèse dite divergente ou dissociée.
LES DIFFÉRENTES MÉTHODES DE SYNTHÈSE DES BANQUES COMBINATOIRES
La synthèse en
parallèle divergente
La synthèse par répartition et mélange
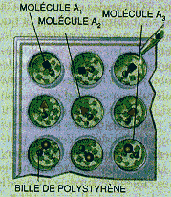


Étape 1
On dispose d'un jeu de trois molécules A 1 , A 2 , A 3 . Ces molécules sont fixées sur des billes de polystyrène différentes.
Étape 2
On ajoute dans chacun des puits le mélange des trois molécules A 1 , A 2 , A 3 . On obtient les molécules A 1 A 1 , A 1 A 2 , A 1 A 3 ; A 2 A 1 , A 2 A 2 , A 2 A 3 ; A 3 A 1 , A 3 A 2 , A 3 A 3 (pour simplifier, on n'a représenté qu'un type de molécules par puits, mais, en réalité, les trois types de molécules sont mélangés, sur chaque ligne).
Étapes suivantes
Au bout de deux
étapes, on a synthétisé 3
2
, soit neuf molécules différentes (en
recommençant
n
fois la même opération, on obtient
3
n
molécules).
Toutefois, les billes portent toutes un mélange de molécules, ce qui rend
l'identification de la molécule active difficile.
MOLÉCULES
A
1
A
1
A
1
A
2
A
1
A
3
A
2
A
1
A
2
A
2
A
2
A
3
A
3
A
1
A
3
A
2
A
3
A
3
Étape 1
Des billes de polystyrène sont introduites dans les tubes à essai. Les molécules A 1 sont placées dans le premier tube, les molécules A 2 dans le deuxième, etc. On n'a représenté que trois tubes, mais on peut en utiliser des dizaines.
Étape 2
Le contenu des tubes est filtré et rincé, et toutes les billes sont regroupées et mélangées.
Étape 3
Le mélange est partagé en trois, et des molécules A 1 , sont introduites dans le tube 1, des molécules A 2 dans le tube 2, etc.
Étapes suivantes
On recommence les opérations de mélange et de partage autant de fois qu'il est nécessaire pour ajouter toutes les sous-unités voulues. Une bille ne porte qu'un seul type de molécules, car, à chaque étape, les billes ne sont en contact qu'avec un seul réactif. Un test colorimétrique identifie la bille qui porte la molécule active.
(23( * ))
Ce type de
synthèse a été rendu possible par le fait que B. MERRIFIELD
24(
*
)
a réussi à développer des peptides sur de petites billes de polymère
chimiquement
inertes.
La chimie combinatoire utilise ces billes de polymère de la façon
suivante :
placées dans un solvant, les billes gonflent, de sorte que l'intérieur devient
accessible aux réactifs dissous dans ce solvant. Chaque bille porte environ
10
13
amorces qui se combinent avec les réactifs. Par réactions successives on
obtient des
oligomères, des molécules constituées d'un enchaînement de plusieurs
sous-unités.
Le marquage des molécules
La seconde
étape consiste à " étiqueter " les molécules au fur et à
mesure
de leur élaboration, de façon à pouvoir identifier chacune d'elles à la fin du
processus. Plusieurs techniques sont utilisées :
- La première est l'ajout, à chaque stade de la synthèse, d'un composé
inerte sur
un site distinct de la bille ; cela crée une sorte de signature chimique
de la
molécule ;
- La seconde est l'ajout, à chaque stade également, d'une base (A, T, G ou
C) qui
permettra ultérieurement de reconnaître la molécule en lisant la séquence après
amplification par PCR (
polymerase chain reaction
) ;
- La société française CEREP utilise des codes barres pour assurer la
traçabilité des produits.
" Chaque opération, chaque produit est
référencé. L'ordinateur lit les codes et permet au chimiste d'accéder
directement aux
informations qu'il recherche ou au robot de savoir quels échantillons il
traite "
.
25(
*
)
- La société californienne Irori a lancé, en juin 1996, le système
Accutag :
il s'agit de développer des molécules sur des billes contenues dans une capsule
où se
trouve également un microprocesseur. La paroi de chaque capsule est comme un
filet qui
laisse passer les réactifs chimiques nécessaires à la synthèse, mais retient les
billes de polymère.
À chaque étape de la synthèse, un signal radio est envoyé à chaque
microprocesseur
afin d'enregistrer en détail la réaction à laquelle la bille est exposée. Ces
données
peuvent ensuite être lues sur les microprocesseurs lorsque les molécules en sont
arrivées à leur stade final.
Toutefois, le système électronique utilisé reste trop volumineux pour permettre
de
produire des centaines de milliers de composés.
C'est pourquoi, Irori a signé des accords avec Rhône-Poulenc Rorer et
Bristol-Myers
Squibb pour mettre au point un nouveau procédé qui devrait, à terme, permettre
de
synthétiser près de 25 000 composés par semaine. L'idée est
d'utiliser des
étiquettes code à barres à deux dimensions et à lecture optique au lieu du
système
des étiquettes à radiofréquence c'est-à-dire des
" volumineux "
microprocesseurs contenus dans la capsule.
Les nouvelles molécules créées par la chimie combinatoire sont rassemblées dans
de
vastes chimiothèques. Les chimiothèques " généralistes "
fournissent une grande diversité de structures moléculaires. Toutefois, ces
molécules
sont d'un intérêt inégal et il est indispensable de les trier pour déterminer
les plus
actives et quel type de cible elles peuvent atteindre.
UNE NOUVELLE
FAÇON DE CRÉER DES MOLÉCULES INÉDITES :
LES MOLÉCULES DARWINIENNES
" Faisant
appel aux principes de la sélection naturelle, un logiciel engendre des
molécules-modèles inédites, futures candidates au statut de médicament.
Plusieurs modèles chimiques permettent déjà de trouver informatiquement de
nouvelles
molécules actives. Le dernier logiciel en date propose d'appliquer la sélection
naturelle darwinienne sur des modèles de molécules. Les résultats de David
NOEVER lui
ont valu le titre d'innovator de l'année 1998, décerné par le magazine américain
Discover. Grâce à cette idée, David NOEVER a fondé une start-up qui travaille
notamment avec le National Cancer Institute (NCI) et une dizaine d'entreprises
privées.
Son logiciel part d'une molécule fournie par un laboratoire, connue pour son
action sur
certaines maladies. Il en construit ensuite jusqu'à trente
" parents ", des molécules élaborées automatiquement selon
des
critères géométriques, physico-chimiques ou autres.
A chaque molécule est associé un " Chromosome " fictif
représentant
ses caractéristiques. L'algorithme déclenche alors un processus de reproduction
sur
cette population de chromosomes, en autorisant les mutations (changement
ponctuel et
aléatoire) et les cross-over (échange d'une séquence entre deux chromosomes). À
chaque
génération, le logiciel choisit les meilleurs reproducteurs, par exemple les
molécules
ayant la configuration de plus basse énergie. En calculant quelques milliers de
générations, on voit apparaître de nouvelles molécules, parmi lesquelles seules
les
meilleures " survivent ". David NOEVER précise que,
" lors
des expériences réalisées pour le National Cancer Institute, les experts du NCI
ont
toujours vérifié que les molécules proposées n'avaient jamais été observées
auparavant ". En jouant sur les paramètres de sélection, NOEVER peut
aussi
modéliser la fixation de ligands sur une protéine, ou encore prédire la forme
exacte
d'une molécule d'après sa formule chimique, en un temps record: quelques
minutes sur une
station de travail. "
La Recherche. N° 313 . Octobre 1998 .
1.1.4.2. Le criblage à haut débit
Les laboratoires pharmaceutiques ont tout intérêt à restreindre leur champ d'investigation expérimentale en le ciblant correctement pour aboutir à de nouveaux médicaments le plus vite possible à un moindre coût. C'est pourquoi ils passent au crible les nouvelles molécules ; ce criblage doit être réalisé à haut débit.
Le criblage virtuel
Cette technique relativement récente est notamment mise en oeuvre par la société Syntem. L'ordinateur aide le biochimiste à faire le tri parmi les molécules. " Par une évaluation rationnelle de l'activité biologique des molécules, on peut diminuer le nombre de molécules à synthétiser en laboratoire d'un facteur mille ou plus " 26( * ) . Puis les nouvelles molécules sont modélisées : leurs propriétés globales (exemple : affinité pour les graisses, solubilité...) sont analysées et ces descriptions sont communiquées à l'ordinateur qui réalise un premier tri, permettant de sélectionner les molécules qui, compte tenu de ces propriétés, ont le plus de chance d'être actives en fonction de telle ou telle cible.
Le criblage biologique
C'est
actuellement le procédé le plus utilisé. Il consiste à identifier les molécules
actives par mise en contact avec la cible biologique. Ces cibles peuvent par
exemple être
des protéines dont on a identifié expérimentalement l'implication dans tel ou
tel
processus pathologique.
Jusqu'au début des années 1990, les essais traditionnels en éprouvette
permettaient à
une personne de tester 2000 composés par an. L'emploi de microplaques à
96 puits a ensuite permis de réaliser 6 000 essais par jour et
par robot
sur une même cible. Aujourd'hui les microplaques comportent 384 puits,
certaines
pouvant aller jusqu'à 1 536 puits. Ces progrès ont permis de
multiplier les
tests et, également, de réduire les coûts car les essais sont
" miniaturisés " et utilisent des volumes d'échantillons
très
réduits.
Le criblage à haut débit se caractérise par l'utilisation de robots capables de
gérer
simultanément de grands nombres de microplaques, facilement manipulables et
stockables,
où sont effectués des milliers de tests par jour.
Le criblage à haut débit est issu des progrès de la robotique. Il repose sur des
systèmes capables de réaliser des taches séquentielles indépendantes telles que
dilution, pipettage et répartition de composés dans des cupules ou puits,
agitation,
incubation, lecture de résultats. Ils sont pilotés par des logiciels
spécifiquement
adaptés au type d'analyse à réaliser. Les essais sont effectués dans des
microplaques
standards identifiées par un code à barres et manipulées par la
" main " d'un robot : celle-ci prend la plaque vide,
ajoute les
réactifs nécessaires et les composés à tester, gère le temps de la réaction puis
passe la plaque à un lecteur pour connaître le résultat. Pour visualiser les
réactions
issues de la mise en contact, dans les puits, de la molécule et de la cible, on
utilise
des méthodes basées sur la radioactivité ou, bien souvent, la fluorescence. Ces
résultats sont stockés et analysés grâce à des ordinateurs.
1.2. DE MULTIPLES APPLICATIONS
La génomique, la bioinformatique, les biopuces et la chimie combinatoire vont offrir des possibilités thérapeutiques remarquables selon des approches très diverses : la sélection de cibles d'intérêt pour la mise au point de médicaments traditionnels, la thérapie génique, les vaccins, la pharmacogénomiques, le diagnostic moléculaire et la production de protéines thérapeutiques.
1.2.1. L'UTILISATION POUR LA RECHERCHE PHARMACEUTIQUE DES CIBLES ISSUES DE LA GÉNOMIQUE
L'efficacité d'un criblage est directement liée à la pertinence de la cible choisie. On comprend aisément, que tester les réactions de milliers de molécules sur des produits biologiques non impliqués dans des processus pathogènes n'offre aucun intérêt. Le choix des cibles est donc essentiel et l'apport de la génomique est considérable. La génomique permet de mieux connaître les bases moléculaires des maladies.
1.2.1.1. La connaissance du génome humain
Les informations
issues de l'analyse systématique du génome humain vont permettre d'identifier
les gènes
liés à certaines maladies et leur fonction biologique via leurs produits :
les
protéines.
" Les gènes deviennent la voie royale pour découvrir des
médicaments : connaître la séquence d'un gène, en déduire la structure de
la
protéine qu'il code, c'est identifier autant de cibles biologiques, autant de
sites
d'interventions sur lesquelles ces médicaments pourront agir.
Sur les milliers de maladies connues, seules une centaine (au plus 150)
représentent un
enjeu majeur de santé publique et nécessitent un traitement (ou une
amélioration du
traitement). La majorité de ces maladies, comme le cancer, l'hypertension,
l'athérosclérose ou certains (troubles mentaux, ont un déterminisme génétique
multifactoriel.
En estimant que le nombre de gènes impliqués dans de telles maladies oscille
entre 5 et
10 (ce qui est généralement admis), on voit que le nombre de gènes liés à une
pathologie se situe entre 500 et 1 000. Si l'on considère que ces gènes et
leurs
produits interagissent avec 3 à 10 autres produits, qui sont autant de sites
d'intervention potentiels, on aboutit à un total de 3 000 à
10 000 cibles
d'intérêt pour des médicaments. Or, c'est précisément le nombre de sites
moléculaires qui sont susceptibles, d'ici six ans, d'émerger du décryptage du
génome
humain !
Autrement dit, c'est par au moins un facteur 10 que l'on peut espérer pouvoir,
en
quelques années, multiplier le nombre de cibles actuellement exploitées (417
seulement,
rappelons-le) ! Sans compter que la plupart des cibles fournies par la
génomique
(comme des récepteurs nucléaires, l'ADN ou les canaux ioniques) sont peu
explorées
aujourd'hui dans le processus de mise au point des
médicaments. "
27(
*
)
La création de cibles issues de la génomique passe généralement par la création
d'ADN
complémentaires (ADNc). En effet, les ARN messagers (ARNm), ces copies d'ADN,
porteuses
de l'information nécessaire pour fabriquer les protéines correspondant aux
gènes,
pourraient être analysés et conservés pour fournir en temps utile des protéines
cibles. Mais ce sont des molécules très fragiles. On utilise une astuce
technique :
à partir des ARNm, on synthétise, en jouant sur la complémentarité des bases
nucléotidiques des ADN complémentaires (ADNc), copies stables des ARNm, offrant
l'intérêt majeur de pouvoir être stockées, copiées et séquencées.
Ces ADN permettent, lorsque l'on veut effectuer un criblage précis, de produire
les
protéines codées par les gènes correspondants, afin de tester les
molécules : il
s'agit principalement de protéines spécifiques comme des enzymes et des
récepteurs,
cibles privilégiés pour la recherche pharmaceutique.
1.2.1.2. La connaissance des génomes bactériens
La connaissance
des génomes bactériens permet également de cribler des familles de molécules de
type
antibactérien et des vaccins.
Un exemple de cibles potentielles pour de nouveaux antibactériens permettant de
contourner les phénomènes de résistance aux antibiotiques : le blocage du
processus de réplication de l'ADN bactérien, déduit de la connaissance du
génome d'
Escherichia
coli
.
" Autre piste, le processus de réplication de l'ADN bactérien
offre aussi
des cibles intéressantes. Chez Escherichia coli, par exemple, plus de trente
protéines
sont impliquées dans la réplication de l'ADN. La mutation des gènes codant ces
protéines entraîne généralement un blocage de la réplication, suivi d'un arrêt
de la
croissance et souvent de la mort bactérienne. D'autre part, la machinerie de
réplication
est similaire chez la plupart des bactéries. De ce fait, un inhibiteur de la
réplication
a toutes les chances d'avoir un spectre d'activité large. A l'heure actuelle
une famille
d'antibactériens, les quinolones, a pour cible les topo-isomérases, enzymes
impliquées
dans les changements de conformation de l'ADN. Mais on connaît maintenant une
vingtaine
d'enzymes (polymérase III holo-enzyme et ses sous unités, ou encore DnaA,
DnaB et
DnaC...) qui interviennent dans la phase initiale de réplication de
1'ADN : ce sont
autant de cibles potentielles pour de nouveaux agents
antibactérien. "
28(
*
)
Depuis le premier séquençage d'un génome bactérien, celui d'
Haemophilus
influenzae
en 1995, de nombreux autre ont été totalement séquencés ou vont l'être
prochainement.
Certains chercheurs estiment que les génomes de la majorité des bactéries
pathogènes
pour l'homme seront séquencés au début des années 2000. Il est certain qu'un
génome
bactérien correctement reconstitué et annoté apporte de nouvelles cibles
permettant le
criblage, soit de nouveaux produits antibactériens, soit de vaccins (ainsi que
cela est
exposé au chapitre 1.2.3., notamment en ce qui concerne les génomes
d'
Helicobacter
Pylori
et de
Mycobacterium tuberculosis
).
1.2.2. LA THÉRAPIE GÉNIQUE
La thérapie génique est la fille de la génomique, qui en est l'indispensable préalable.
1.2.2.1. Origine et application de la thérapie génique
Le défi
de la thérapie génique est de parvenir à corriger, à l'intérieur des cellules
d'un
organisme humain, les anomalies qui, en affectant son génome, sont responsables
de
pathologies graves et, le plus souvent, actuellement incurables.
L'objectif est d'atteindre et de supprimer la cause de la maladie et de ne plus
se
contenter d'atténuer ou d'effacer les symptômes.
" La thérapie génique est l'insertion délibérée de matériel
génétique
dans l'organisme d'un patient pour corriger un défaut précis à l'origine d'une
pathologie, que ce soit à titre curatif ou préventif "
29(
*
)
.
Cette définition inclut à la fois la thérapie du gène (réparation de gènes dont
l'altération est responsable de maladies), objectif qui prévalait pour les
maladies
génétiques en 1983, après la mise au point des premiers vecteurs, et
l'utilisation de
gènes comme nouveaux types de médicament.
À l'origine, la cible la plus logique de la thérapie génique a paru être le
domaine
des maladies monogéniques héréditaires : pour ces affections, on était
certain
que les personnes porteuses d'un gène sain ne développeraient pas la maladie.
Par
ailleurs, on connaissait précisément les gènes responsables de nombreuses
maladies
génétiques grâce à l'action menée notamment par l'Association françaises contre
les
myopathies. Mais les champs d'application de la thérapie génique se sont ensuite
diversifiés.
Le principe de cette thérapie, est d'introduire un gène-médicament à
l'intérieur de la cellule cible afin de :
- corriger une maladie génétique en introduisant dans les cellules malades
un
gène-médicament faisant défaut ;
- inhiber ou stimuler la synthèse d'une protéine donnée.
Il existe trois méthodes de thérapie génique :
- La thérapie génique
ex vivo
consiste à prélever sur le patient les
cellules cibles, à les modifier génétiquement avec le vecteur viral porteur du
gène
thérapeutique puis à les réintroduire chez le patient.
Cette méthode est utilisée en particulier pour les cellules sanguines qui sont
faciles
à prélever et à réintroduire.
- La thérapie génique
in situ
consiste à placer directement au sein du
tissu
cible le vecteur de transfert. Cette technique est expérimentée, notamment,
dans les cas
de mucoviscidose (transfert de vecteurs dans la trachée et les bronches), de
cancer
(injection dans la tumeur d'un vecteur portant le gène d'une toxine, par
exemple), ou de
dystrophie musculaire (injection dans le muscle d'un vecteur porteur du gène de
la
dystrophine) ;
- La thérapie génique
in vivo
consiste à injecter le vecteur portant le
gène
thérapeutique directement dans la circulation sanguine ; le vecteur est
alors censé
atteindre spécifiquement les cellules cibles.
1.2.2.2. Les obstacles techniques
La thérapie
génique suppose nécessairement :
- un gène-médicament ;
- un vecteur pour le transporter ;
- une cellule cible où le gène puisse s'exprimer.
Aujourd'hui, l'évolution de la thérapie génique repose essentiellement sur le
développement des systèmes de transfert des gènes : ils doivent être sûrs,
efficaces, capables d'exercer leur fonction dans des cellules qui ne se
divisent pas et
d'assurer la stabilité de l'expression du gène thérapeutique.
Les principaux types de vecteurs
Les virus ont la
capacité de franchir, dans certains conditions, les barrières de protection que
dresse
le corps humain en cas d'introduction d'ADN étranger dans son génome. Ils sont
capables
d'introduire leur matériel génétique dans les cellules qu'ils infectent. C'est
pourquoi
les chercheurs ont eu l'idée de les utiliser pour transférer les gènes
thérapeutiques
dans les cellules des patients.
Bien entendu, les virus utilisés ne doivent présenter aucun danger : on
transforme
donc génétiquement les virus en ôtant, dans leur propre génome, les séquences
nécessaires à leur réplication et leur virulence (les gènes E 1 et
E 4).
Différents types de virus sont utilisés comme vecteurs :
-
Les rétrovirus
ont été les premiers virus testés. Leur qualité
principale
est de pouvoir intégrer leur matériel génétique de façon permanente dans le
génome
des cellules qu'ils infectent. Actuellement, 60 % des protocoles cliniques
sont
fondés sur l'utilisation de vecteurs rétroviraux, dérivés des rétrovirus de la
leucémie murine (MLV, virus de Moloney en particulier). Ils peuvent contenir un
ADN
exogène de taille relativement grande : huit kilobases
30(
*
)
.
À l'exception du VIH (virus de l'immunodéficience humaine), ces virus ont
évolué sous
des formes peu pathogènes et leur utilisation présente des risques limités.
Ils sont utilisés selon la technique
ex vivo
. La pénétration des
rétrovirus
dans les cellules cibles se fait grâce à la reconnaissance, par un récepteur
cellulaire, d'une protéine présente sur l'enveloppe virale. Mais, pour être
efficace,
c'est-à-dire pour s'intégrer dans le chromosome cellulaire, le vecteur ne peut
se
contenter d'être entré dans le cytoplasme de la cellule : il doit pénétrer
jusqu'au noyau de la cellule. Ce n'est possible qu'au moment où la cellule se
divise pour
se reproduire (mitose), car la membrane du noyau est alors momentanément
rompue. Cette
particularité explique que l'on doive utiliser la technique
in vivo
car,
lorsque
l'on cultive, en laboratoire, les cellules humaines, la plupart d'entre elles
sont
réceptives aux rétrovirus MLV et se divisent activement lors de l'exposition aux
rétrovirus qui peuvent ainsi pénétrer dans leurs noyaux.
Mais l'approche
ex vivo
a ses limites. Les cellules sanguines qui
pourraient être
ainsi traitées, car elles sont faciles à prélever et à réintroduire dans
l'organisme,
sont malheureusement peu réceptives au MLV : elles expriment peu le
récepteur à ce
virus, qui pénètre donc difficilement en elles.
L'utilisation des rétrovirus
in vivo
est encore plus délicate :
- Les vecteurs doivent atteindre principalement les cellules cibles ;
or, les
cellules endothéliales expriment naturellement un récepteur au MLV ; de ce
fait,
les rétrovirus modifiés peuvent pénétrer dans ces cellules rencontrées sur le
chemin
qui doit les mener aux cellules cibles. Dans la mesure où les vecteurs
rétroviraux ne
peuvent pas être produits à des concentrations élevées, le fait qu'ils se fixent
partout dans la circulation sanguine les empêche de parvenir en quantité
suffisante pour
une transduction
31(
*
)
efficace dans les cellules
cibles.
- La plupart des cellules considérées comme des cibles potentielles pour
une
thérapie génique ne prolifèrent pas activement
in vivo
. Les rétrovirus ne
peuvent profiter de la mitose pour s'introduire dans leur noyau. La solution de
l'avenir
sera peut-être l'utilisation des lentivirus, (tel que le VIH) qui sont capables
de
pénétrer dans le noyau des cellules ne se divisant pas. Le risque principal
présenté
par ces virus est une éventuelle recombinaison entre le génome viral et le
génome des
cellules transduites, susceptible de produire un virus pathogène.
C'est pourquoi les recherches portent sur des systèmes hybrides incluant le
génome
modifié du VIH (débarrassé des gènes responsables du caractère pathogène du
virus)
dans un vecteur rétroviral. Cette méthode paraît intéressante mais n'en est
qu'à ses
balbutiements.
-
Les adénovirus
ont des caractéristiques intéressantes : leur
grande
taille permet le transfert de très larges séquences d'ADN (plus de
35 kb) ;
ils sont capables d'infecter un grand nombre de types de cellules différentes,
même si
elles ne sont pas en phase de mitose ; ils peuvent être produits à des
concentrations élevées. Ils ont aussi des défauts, notamment celui de provoquer
de
fortes réactions inflammatoires et immunitaires. C'est pourquoi les vecteurs
adénoviraux
de deuxième génération contiennent des génomes réduits des virus. On doit
toutefois
noter qu'ôter des séquences des génomes viraux présente des inconvénients :
ainsi, il peut être nocif de retirer les séquences correspondant à des régions
dites
" activatrices " qui aident à maintenir la stabilité du
génome
viral dans la cellule.
Aujourd'hui, pour lutter contre ces réactions, l'utilisation des vecteurs
adénoviraux
suppose d'administrer des gènes viraux contenant des gènes immunosuppresseurs.
Une autre
solution consisterait à administrer un traitement
" classique " de
produits immunodépresseurs au patient parallèlement au traitement par thérapie
génique. Parvenir à supprimer les réactions immunitaires est essentiel car les
vecteurs
adénoviraux ne s'intègrent pas dans le génome de la cellule cible et ont
tendance à
disparaître au fil des divisions cellulaires. Il faut pouvoir les administrer
de façons
répétées sans déclencher de réaction immunitaire.
-
Les adéno-associated virus (AAV)
sont des virus non pathogènes très
répandus
chez l'homme. Ils ne peuvent se répliquer qu'en s'associant avec des adénovirus
ou des
virus de l'herpès. Ils peuvent transduire efficacement les cellules du cerveau,
du foie
et certaines cellules sanguines. Ils peuvent infecter des cellules en dehors
des phases de
mitoses. Malheureusement cette qualité essentielle disparaît lorsque l'on
modifie le
génome des AAV pour y introduire le gène-médicament : les vecteurs restent
capables d'infecter des cellules ne proliférant pas mais ne s'intègrent plus
dans leur
génome. Par ailleurs, les AAV présentent un autre inconvénient : celui de
ne
pouvoir contenir que des petites séquences d'ADN (4,8 kb).
Ces AAV pourront donner des vecteurs peu dangereux et présentant certains
avantages mais
ils sont actuellement inexploitables dans le domaine clinique.
-
Les vecteurs synthétiques
ont plusieurs qualités : facilement
produits,
ils sont stables et peuvent contenir des séquences d'ADN de grande taille. Ce
sont des
lipides, des peptides ou des polymères dits cationiques car ils sont porteurs
d'une
charge électrique positive. Celle-ci leur permet de compacter les milliers de
paires de
bases d'une molécule d'ADN (chargées négativement) et de donner une charge
positive à
l'ensemble (vecteur + ADN) qui peut interagir avec les charges négatives
des
membranes des cellules.
La pénétration
in vitro
des vecteurs synthétiques dans les cellules ne
pose pas
de problèmes ; malheureusement les résultats
in vivo
sont très
décevants.
Il semble que, injectés par voie intraveineuse, les vecteurs s'agrègent en
particules de
grande taille mécaniquement retenues par les deux principaux filtres du corps
humain
(poumon et foie).
Compte tenu de ces difficultés, l'une des utilisations envisagée à terme, pour
les
vecteurs synthétiques est le traitement de la mucoviscidose par instillation du
gène-médicament dans les poumons (dans ce cas, il suffirait semble-t-il
d'atteindre
5 % des cellules pulmonaires) :
Les défauts actuels des vecteurs synthétiques pourraient être corrigés au prix
d'importants efforts de recherche dans le domaine de la chimie et de la
biochimie. Or
l'industrie pharmaceutique est prête à valoriser son savoir-faire traditionnel
en chimie
en étudiant de près les vecteurs synthétiques. Les retombées en seraient
positives
pour les groupes pharmaceutiques et pour la thérapie génique.
|
VECTEURS |
CARACTÉRISTIQUES |
TAILLE DE L'INSERT |
VOIX D'ADMINISTRATION |
INDICATIONS CLINIQUES |
COMMENTAIRES |
|
Adénovirus |
- Efficace (vecteur de
référence)
|
10-14 kb
|
- intratrachéale
|
- Oncologie
|
- Production industrielle
OK
|
|
Adeno Associated Virus (AAV) |
- Efficacité modérée
|
4,5 kb |
- intratrachéale
|
- Oncologie |
- Production industrielle
difficile
|
|
Rétrovirus
|
- Efficacité modérée
|
4-6 kb |
-
ex vivo
|
- Oncologie
|
- Production
industrielle : OK
|
|
Vecteurs synthétiques |
- Efficacité
modérée/limitée
|
illimitée (en théorie) |
- intramusculaire
|
- Maladies génétiques
|
- Production
industrielle : +/-
|
|
Pox Virus |
- Efficaces
|
20 kb |
- intramusculaire |
- Immunothérapie (cancers et infectieux) |
- Production
industrielle : OK
|
|
Vecteurs cellulaires |
- Efficaces
|
30 kb |
-
intramusculaire ou
|
- Immunothérapie (cancers) |
- Production
industrielle : OK
|
Source : Transgène
L'expression des gènes
Si le problème
de l'efficacité du transfert du gène-médicament par des vecteurs est résolu à
terme,
il conviendra d'obtenir du gène une expression durable et au bon niveau
.
L'intérêt de la thérapie génique, du moins telle qu'elle était conçue à
l'origine,
est en effet de traiter la cause de la maladie et non seulement ses
symptômes :
apporter dans l'organisme du patient des gènes destinés à compenser le
dysfonctionnement de certains gènes ou leur absence n'est efficace que si le
gène de
remplacement exerce réellement ses fonctions, c'est-à-dire s'il exprime les
protéines
indispensables à la santé du patient.
Ce problème est loin d'être résolu
Une expression stable et à un bon niveau
Plusieurs
facteurs empêchent le maintien de l'expression des gènes après leur
transfert :
- les séquences régulatrices
32(
*
)
contrôlant
l'expression du gène thérapeutique sont souvent reconnues comme étrangères et
inactivées par la cellule qui les reçoit ;
- si l'efficacité du gène-médicament n'est pas atteinte, c'est alors la
cellule
" d'accueil ", celle qui a reçu le gène, qui est détruite
par le
système immunitaire du patient, ce système reconnaissant et éliminant les
produits de
gènes étrangers et les cellules qui les expriment.
Une expression régulable
Si les
problèmes précédents sont résolus, il faudra obtenir une expression régulable du
gène. En effet, plusieurs gènes importants, tel que celui par exemple qui
permet la
production d'insuline, ne s'expriment pas au même taux en continu mais
répondent à des
signaux physiologiques. Il faudrait donc que les séquences génétiques insérées
puissent répondre aux signaux physiologiques du corps et fonctionnent comme les
autres
gènes de l'organisme. Devant la complexité de ce problème une autre possibilité
est
envisagée : utiliser des substances chimiques administrées de façon
traditionnelle
et permettant de contrôler le niveau d'activité du gène.
Cette combinaison du gène-médicament et du produit pharmaceutique classique est
étudiée avec intérêt par l'industrie pharmaceutique : cela lui permettrait
d'exercer un certain contrôle sur le marché de la thérapie génique. Des
recherches
sont déjà en cours : ainsi, les chercheurs de l'Institut de thérapie
génique de
l'Université de Pennsylvanie, associés à ceux de la société Ariad
Pharmaceuticals ont
conçu une approche dans laquelle un vecteur biologique (virus) est utilisé pour
délivrer aux patients des gènes thérapeutiques ayant une particularité
intéressante : ils sont inactivés au départ et les patients doivent
ingérer une
pilule composée de produits chimiques pour déclencher leur expression.
Pour conclure, on peut noter que :
- si la vectorologie progresse, la thérapie génique
ex vivo
permettra
vraisemblablement, d'ici quelques années, d'améliorer la situation clinique des
patients. En revanche, la thérapie génique
in vivo
semble beaucoup plus
difficile
à mettre en oeuvre ;
- compte tenu des problèmes de contrôle à long terme de l'expression des
gènes-médicaments, une réorientation des finalités de la thérapie génique est en
cours.
1.2.2.3. La réorientation des applications de la thérapie génique
" Il existe autant de thérapies géniques que de maladies à traiter, chaque type de tissu comportant des problèmes particuliers. Le fait de traiter une maladie acquise ou d'essayer de corriger une maladie héréditaire implique des problèmes de nature très différente " 33( * ) . Les maladies héréditaires monogéniques, les maladies acquises et les vaccins ont des horizons thérapeutiques très différents.
Les maladies héréditaires
Leur horizon
thérapeutique est lointain. La seule solution, en matière de thérapie génique
est
d'introduire une bonne version du gène déficient, ou de moduler l'expression de
certains
gènes. Or, ainsi que l'a montré le paragraphe précédent, les techniques
actuelles ne
permettent pas de contourner les réactions inflammatoires ou immunitaires ni
d'obtenir
une expression stable et persistante du gène.
Les maladies héréditaires monogéniques sont régies par deux grands mécanismes
l'un de
perte, l'autre de gain de fonction.
En cas de perte de fonction, la carence fonctionnelle d'un gène aboutit à un
déficit
(tel est le cas, par exemple, de l'hémophilie ou de la mucoviscidose) ; la
stratégie consiste alors à le remplacer par un gène en état de fonctionner.
Lorsqu'il y a gain de fonction (drépanocytose, chorée de Huntington,
polykystose rénale
héréditaire), les symptômes de la maladie sont liés à la synthèse d'une protéine
anormale à effets délétères sous la direction du gène muté ; l'apport d'un
gène fonctionnel ne suffit pas à avoir des effets thérapeutiques ; il faut
inhiber
le fonctionnement du gène muté ou inactiver son produit protéique.
Lorsqu'un effet thérapeutique peut être espéré par la production dans la
circulation
d'une substance très active, par exemple une hormone, une cytokine ou un
facteur de
coagulation, un certain optimisme est raisonnable, même si les difficultés ne
sont pas
toutes résolues.
En revanche, lorsqu'il s'agit de faire pénétrer un gène fonctionnel dans un
très grand
nombre de cellules affectées et si une expression prolongée du gène
thérapeutique est
nécessaire, on ne peut être optimiste, du moins à court et moyen terme.
Ces difficultés ne condamnent nullement les recherches. Cette solution serait
particulièrement mal venue en France, compte tenu des efforts humains,
scientifiques et
financiers des associations de malades telles que l'Association française
contre les
myopathies (AFM).
" Chaque jour apporte un lot de nouvelles découvertes. Partout
dans le
monde, des chercheurs contribuent à leur développement. Après le cancer, les
essais
cliniques débutent pour les maladies génétiques. Même si tout est loin d'être
résolu, et que des questions importantes sont encore sans réponse, nous y
croyons plus
que jamais. Parce que nous savons que de la masse critique de connaissances
accumulées
grâce à nos efforts au cours des dernières années, doivent émerger de nouvelles
idées, de nouveaux concepts qui permettront de lever les derniers obstacles à
la mise au
point de traitements. "
34(
*
)
Dans un premier temps, la thérapie génique progressera plutôt, dans le domaine
des
maladies acquises.
Les maladies acquises
Les maladies
aiguës nécessitent des vecteurs efficaces qui transfèrent de fortes quantités de
gènes n'ayant pas obligatoirement une expression prolongée. On peut noter par
ailleurs
que les réactions inflammatoires liées à l'emploi de certains vecteurs sont de
moindre
importance dans la mesure où les gènes-médicaments ne sont pas destinés à être
administrés de façon régulière et continue.
-
Le cancer
est une cible majeure de la thérapie génique.
L'oncologie
mobilise aujourd'hui la majorité des essais cliniques. Une grande variété
d'approches
sont actuellement expérimentées :
La technique du gène-suicide : on transfère dans des cellules
(généralement
des lymphocytes) le gène d'une enzyme, la thymidine-kinase (TK), du virus de
l'herpès
(HSV). Ces cellules génétiquement modifiées sont injectées dans la tumeur
maligne. Le
" gène-suicide ", c'est-à-dire le fragment d'informations
génétiques issu du virus de l'herpès, s'introduit au sein des cellules
malignes. Les
médecins administrent alors au patient un médicament qui n'est actif qu'en
présence
d'affections virales herpétiques (Cymevan ou Ganciclovir). Ce médicament
provoque la
destruction sélective des cellules tumorales.
La lutte contre l'angiogenèse :
La capacité, pour une tumeur cancéreuse, de grossir et de métastaser est
étroitement
liée à la prolifération des nouveaux vaisseaux sanguins qu'elle induit. Ce
phénomène,
appelé angiogenèse, permet la transformation d'un petit amas de cellules
anormales en
une grosse masse pouvant se disséminer dans tout l'organisme.
Bloquer l'angiogénèse est un bon moyen de lutter contre les tumeurs. Cela peut
être
réalisé grâce à l'angiostatine, une protéine existant naturellement dans le
corps. On
peut notamment citer, dans ce domaine, les travaux d'une équipe
franco-américaine
35(
*
)
qui a mis au point
une technique de thérapie génique pour
fabriquer deux anti-angiogenèses : l'angiostatine et l'endostatine et leur
permettre
d'agir efficacement ; les gènes qui codent pour la protéine inhibitrice de
l'angiogénèse sont introduits dans un rétrovirus puis transférés dans des
cellules
souches de moelles osseuses cultivées
in vitro
qui sont ensuite
transplantées
dans l'organisme. Cette technique semble toutefois plus efficace à l'heure
actuelle chez
la souris que chez l'homme.
L'immunothérapie
est également un moyen de lutte contre le cancer.
Cette
technique sera étudiée dans le chapitre consacré aux nouveaux vaccins.
La destruction spécifique des cellules tumorales
peut également
être
envisagée grâce à des produits cytotoxiques. On utilise alors la brièveté
d'expression du gène comme un atout ce qui, dans le cas d'autres thérapies
géniques
appliquées à des maladies héréditaires chroniques, est un handicap.
Certains vecteurs cellulaires mis au point par la Société Transgène ont une
expression
brève et une forte efficacité. Il s'agit de cellules dites
" vero ",
provenant d'un rein de singe ; elles ont la capacité de
" sur-exprimer " le gène-médicament avant d'être détruites
rapidement. D'où leur intérêt, par exemple, pour stimuler la fabrication d'un
cytotoxique comme l'interleukine II, au niveau d'une tumeur, sans risque
d'affecter,
à cause d'une expression trop durable du gène, les autres cellules de
l'organisme.
Le gène P 53
. Cette thérapie est actuellement en
phase III des
essais cliniques
36(
*
)
; elle semble assez
efficace
notamment contre le cancer de la sphère ORL appelé aussi cancer
" tête et
cou ". On envisage d'associer le traitement à une chimiothérapie ou
une
radiothérapie.
Il s'agit d'une injection dans les tumeurs cancéreuses du gène suppresseur de
tumeurs,
P 53, surnommé " gardien du génome ". Ce gène est
naturellement présent dans les cellules. Lorsque des mutations surviennent, il
fait en
sorte qu'elles soient aussitôt réparées. Si les dégâts sont trop importants, il
amorce le programme d'autodestruction de la cellule. Malheureusement, ce gène
est
lui-même parfois victime d'une mutation et n'effectue plus son travail. La
cellule est
alors livrée à elle-même et risque, à tout moment, de dériver vers la
cancérisation.
La moitié des cancers portent la marque de ce défaut. La thérapie génique
permet de
compenser cette mutation en introduisant un exemplaire actif du gène dans les
cellules
cancéreuses pour réactiver les mécanismes de correction ou d'autodestruction.
Des
essais cliniques de thérapies génique, avec le gène P 53, sont actuellement
conduits sur des cancers aussi variés que le cancer du poumon, du sein, de
l'ovaire, de
la prostate, du cerveau..., mais ils en sont encore à des phases précoces.
En tout état de cause, même si la thérapie génique peut offrir, à terme, des
opportunités pour le traitement des cancers, il faut rester très prudent et
garder à
l'esprit le récent abandon, par le groupe Novartis, des recherches sur le
glioblastome
(cancer du cerveau). Celles-ci avaient atteint la phase III des essais
cliniques et
les Drs David KLATZMANN et Jean-Loup SALZMANN avaient constaté des
pourcentages de
rémission jamais atteints par un autre traitement. Toutefois, l'essai de
phase III,
mené en aveugle (patients et médecins ignorant qui prend le médicament ou le
placebo) a
donné des résultats trop peu significatifs pour que Novartis envisage une
commercialisation.
-
Les maladies cardiovasculaires
pourraient aussi bénéficier de
thérapies
géniques. On évalue en particulier les possibilités de stimuler la repousse de
vaisseaux sanguins par transfert de gènes de facteurs de croissance des cellules
endothéliales vasculaires (VEGF). Une maladie vasculaire avancée peut en effet
déboucher sur la survenue d'ischémie dans les extrémités des membres ; on
pourrait remédier à celle-ci par l'injection directe de vecteurs exprimant le
VEGF
permettant ainsi d'accroître l'apport sanguin dans les territoires atteints.
Dans ce cas encore, la limitation dans le temps de l'expression de gène n'est
pas un
handicap car il suffit que celle-ci dure un mois pour que les vaisseaux
repoussent. Selon
le Dr. LEBOULCH, l'efficacité de cette technique peut être accrue si l'on
injecte
directement les vecteurs dans les zones ischémiques à l'aide d'un cathéter
spécial.
" La stratégie consistant à faire développer de nouveaux vaisseaux
sanguins (ou angiogénèse) pour pallier l'obstruction des artères est une
situation
relativement accessible à une thérapie génique
car il n'est ici pas
nécessaire
ni même souhaitable d'obtenir une expression prolongée des gènes
thérapeutiques. En
effet une expression prolongée des gènes induirait probablement la formation de
nouveaux
vaisseaux sanguins dans d'autres régions de l'organisme, ce qui pourrait
conduire à la
formation de tumeurs.
Les scientifiques de l'Université de Cornell à New-York, (US) financés par Gen
Vec ont
injecté le gène codant le VEGF (un facteur de croissance des vaisseaux sanguins)
directement dans le muscle cardiaque tout en réalisant une opération de
pontage. Les
résultats de ces essais viennent d'être rendus publics. Ils sont extrêmement
encourageants. De leur côté les chercheurs de Collateral Therapeuties ont
utilisé le
gène codant le facteur de croissance des fibroblastes. Ils prévoient
d'administrer le
gène-médicament par un
cathéter dans les artères coronaires des
patients. Les
essais cliniques de phase I/II doivent débuter au cours du premier
trimestre 1998.
Sous réserve d'un succès, des essais de phase III pourraient démarrer
dès
l'année prochaine ce qui signifie que le produit pourrait être commercialisé
dans trois
ans. Les
essais menés
par Gen Vec devraient concerner une dizaine
de
patients avec
un calendrier
relativement similaire à celui de
Collateral
Therapeutics. Les deux approches, utilisant des vecteurs adénoviraux, ont
probablement
chacune leurs avantages et leurs inconvénients. Par exemple, la
technique
d'administration de Collateral Therapeutics semble d'emblée plus simple et moins
invasive. En revanche, il est possible que l'approche de Gen Vec permette de
conserver le
gène au site où il doit s'exprimer. D'autres sociétés ont déjà démarré des
essais
utilisant le même type de gènes pour traiter des troubles vasculaires
périphériques
(autres que cardiaques). "
37(
*
)
-
Les autres pathologies :
D'autres voies thérapeutiques actuellement à l'étude visent les maladies
infectieuses
comme le SIDA et l'hépatite B, les affections articulaires inflammatoires,
la
maladie d'Alzheimer et la sclérose en plaques.
Dans le domaine du SIDA, de nombreux projets existent mais beaucoup se heurtent
à deux
problèmes : les difficultés de transfert des gènes et le peu de modèles
animaux.
Les maladies sensibles aux protéines thérapeutiques
Certaines
maladies peuvent être traitées par la sécrétion dans l'organisme d'une protéine
" manquante ".
Cette protéine peut être l'érythropoïétine, qui favorise la production des
globules
rouges ou, dans le cadre de maladies lysosomiales, l'enzyme déficiente
susceptible
d'être captée par les cellules de l'organisme. Dans le diabète
insulino-dépendant, il
faut fournir à l'organisme de l'insuline en quantité suffisante.
D'autres protéines dont l'insuffisance provoque des maladies graves peuvent être
produites
in situ
par l'organisme : la facteur VIII,
l'urokinase, la
calcitonine (maladie de Paget). On peut alors parler de protéines de troisième
génération, la première génération étant celle des protéines purifiées et la
deuxième celle des protéines recombinantes.
" Avec la thérapie génique, le gène devient un possible
médicament. La
thérapie génique pourra s'appliquer, évidemment, à des maladies génétiques, et,
plus
généralement, à toutes les affections, héréditaires ou acquises, qui peuvent
bénéficier du traitement par une protéine thérapeutique. En effet, il est, en
principe, toujours possible de remplacer un médicament protéique par le gène
qui va en
commander la synthèse dans les propres cellules du malade qu'il faut soigner.
En ce sens,
la thérapie génique constitue la troisième étape de l'utilisation des protéines
médicamenteuses. La première est celle de la purification à partir de tissus ou
de
fluides animaux ou humains ; la seconde est le génie génétique, où l'on
asservit
des micro-organismes à produire le médicament protéique en leur transférant le
gène
correspondant ; la thérapie génique, enfin, transfère directement l'ADN
dans
l'organisme qu'il faut soigner, des cellules de celui-ci devenant des
microfabriques du
médicament et des microsystèmes de sa délivrance au
malade. "
38(
*
)
Ainsi, la firme américaine Chiron a récemment lancé des essais de thérapie
génique
susceptible de traiter les personnes atteintes d'hémophilie A. Ces essais
visent à
vérifier les possibilités d'injection, par voie intraveineuse, de gènes codant
pour la
production directe par l'organisme d'un facteur sanguin précis : le
facteur VIII.
De même, la société Avigen a déposé un brevet relatif au traitement de l'anémie
par
l'injection, par l'intermédiaire du vecteur viral AAV, du gène codant pour l'EPO
(erythropoïétine).
La thérapie génique associée
Cette approche
thérapeutique semble très intéressante. C'est la combinaison de la thérapie
génique
et d'un traitement médical ou chirurgical classique. On peut citer quelques
exemples :
- Lors des greffes de moelle osseuse, on est amené à injecter au patient des
lymphocytes
du donneur de moelle ce qui présente l'inconvénient d'exposer le patient à un
risque de
maladie de greffon contre l'hôte qui peut être très grave.
L'une des parades possibles est de prélever des lymphocytes du donneur et d'y
transférer
un gène-suicide (enzyme TK du virus de l'herpès, comme dans le cas des
tumeurs
cancéreuses) avant de les injecter au patient greffé. Si ces lymphocytes
entraînent
effectivement une réaction sévère du greffon contre le receveur, on peut
administrer à
ce dernier une molécule comme le Ganciclovir. Les lymphocytes modifiés, rendus
sensibles
à cette substance seront détruits.
- Les cellules cancéreuses résiduelles peuvent également être
détruites :
Lors de traitements de cancers par chimiothérapie, l'on envisage souvent des
greffes
autologues de cellules sanguines (à l'origine des défenses immunitaires). Dans
ce cas,
on réinjecte au patient ses propres cellules, qu'elles aient ou non été
modifiées ; souvent il peut rester des cellules cancéreuses dans les
populations
cellulaires sanguines que le patient reçoit. Une équipe américaine (université
de
Yale) vient de mettre au point un système de thérapie génique par gène suicide
permettant de se débarrasser des cellules cancéreuses résiduelles.
- La thérapie génique peut également protéger les patients des effets
négatifs
de la chimiothérapie anticancéreuse.
Une des limitations de la chimiothérapie est l'excessive toxicité des molécules
utilisées s'exerçant notamment vis-à-vis des cellules sanguines et conduisant à
une
immunodépression sévère. Des stratégies de thérapie génique proposées reposent
sur
le transfert chez le malade de gènes protecteurs dans les cellules à l'origine
des
lignées sanguines et présentes dans la moelle osseuse. Dans ce type de
méthodes, le
transfert de gène est d'abord entrepris sur des cultures de cellules de moelle
osseuse du
malade (ex vivo), et les cellules modifiées sont ensuite ré-injectées au
patient.
Celui-ci peut alors recevoir sans risque un traitement chimiothérapique
intensif.
1.2.2.4. Le point sur la thérapie génique
Les maladies concernées par la thérapie génique
|
TÊTE |
Cécité : |
- thérapie génique pour neutraliser un gène déficient. |
|
|
Maladie de Parkinson : |
- ralentissement de la mort des neurones et stimulation de leur repousse. |
|
MUSCLES |
Myopathie de Duchenne (1 naissance/ 3000) : |
- injection du gène de l'utrophine pour rétablir la liaison muscle-cerveau. |
|
|
Dégénérescence musculaire due au vieillissement : |
- injection d'un facteur de croissance des muscles (IGF-1). |
|
SANG |
Diabète insulino-dépendant : |
- restitution d'une enzyme impliquée dans la synthèse du glycogène et de l'insuline. |
|
|
Hypercholestérolémie familiale : |
- mécanisme de fixation du cholestérol déficient. |
|
|
Anémie (déficit dans le transport d'oxygène) : |
- injection d'un facteur de croissance des muscles (IGF-1). |
|
|
Hémophilie A (1 naissance/5000) et B (1 naissance/50000) - défaut de coagulation du sang : |
- injection dans les muscles ou le foie du gène manquant. |
|
|
Intolérance au lactose (sucre présent dans le lait) : |
- introduction du gène codant pour la galactoridase. 50 % de la population mondiale touchée. |
|
POUMONS |
Mucoviscidose (1 naissance/2000, espérance de vie, environ 20 ans) - affection des poumons : |
- transfert du gène CFTR par voie orale. |
|
COLONNE VERTÉBRALE |
Sclérose latérale amyotrophique (paralysie progressive) : |
- on injecte un facteur de croissance des neurones ou un gène pour stopper la mort des neurones. |
|
CoeUR ET ARTÈRES |
Maladies cardiovasculaires : |
- revascularisation autour des veines obturées. |
|
SYSTÈME IMMUNITAIRE |
Maladie chronique granulomateuse (défaut de la lutte anti-infectieuse qui provoque infections sévères pluriviscérales) : |
- on introduit le gène codant pour l'enzyme NADPH oxydase. |
|
|
Maladies infectieuses : SIDA, paludisme, hépatite B, herpès, tuberculose, grippe, athérosclérose : |
- système de vaccination ADN. |
|
CANCERS |
Cerveau, ORL, prostate, reins, peau, sein, poumon, leucémie : |
- certains gènes injectés poussent les tumeurs à s'autodétruire, d'autres les asphyxient ou les empêchent de migrer, ou encore stimulent le système immunitaire contre elles. |
|
Source : L'Usine nouvelle - Hors série - Mars 1999. |
||
Les essais cliniques en cours 39( * )
|
PAYS |
PROTOCOLES |
PATIENTS |
||||
|
|
Nombre |
% |
Nombre |
% |
||
|
Amérique
|
277
|
75,5 |
2210
|
71,5 |
||
|
Europe
|
70
|
19,1 |
510
|
16,5 |
||
|
Asie |
10 |
2,7 |
29 |
0,9 |
||
|
Autres |
10 |
2,7 |
369 |
11,1 |
||
|
Total : |
367 |
100 |
3118 |
100 |
||
|
PHASE |
PROTOCOLES |
PATIENTS |
||||
|
I |
241 |
1463 |
||||
|
I / II |
92 |
857 |
||||
|
II |
32 |
520 |
||||
|
III |
2 |
249 |
||||
|
Total : |
367 |
3089 |
||||
|
|
PROTOCOLES |
PATIENTS |
||||
|
Cancer |
230 |
2099 |
||||
|
Cardiovasculaires |
11 |
39 |
||||
|
Maladies infectieuses |
29 |
405 |
||||
|
Maladies génétiques |
53 |
286 |
||||
|
Maladies auto-immunes |
5 |
28 |
||||
|
Autres |
39 |
232 |
||||
|
Total : |
367 |
3089 |
||||
Marché et industrie de la thérapie génique
- Alors
que, en 1997, le marché de la thérapie génique de 2010 était estimé à
3,2 milliards d'euros (selon la Business Communications Company), en 1998,
les
estimations étaient passées à 41 milliards d'euros vers 2010 (estimation
de la
Federal Trade Commission).
- Les grands groupes pharmaceutiques impliqués sont : Rhône-Poulenc
Rorer et
Hoechst Marion Roussel (Aventis), Schering-Plough, Novartis, Glaxo Wellcome et,
depuis
peu, Roche.
- La thérapie génique en France
40(
*
)
:
|
Les laboratoires : |
|
|
|
CNRS |
|
|
INSERM |
|
|
Généthon : association financée par le Généthon, entièrement consacrée à la recherche et à la production de vecteurs de thérapie génique |
|
Les entreprises : |
|
|
|
Gencell RPR avec un centre de production à Vitry |
|
|
Transgène avec un centre de production à Strasbourg |
|
|
Génopoïétic avec un centre de production à Lyon |
|
Les principaux centres de traitement : |
|
|
|
Hôpital Pitié-Salpêtrière |
|
|
Hôpital Necker |
|
|
Institut Gustave-Roussy |
|
|
Institut Curie |
|
|
Centre hospitalier de Nantes |
|
|
Centre Léon-Bérard de Lyon |
|
|
Centre hospitalier de Lyon-Sud |
|
|
Hôpital Debrousse, Lyon |
|
|
Établissement de transfusion sanguine de Besançon |
|
|
Institut Curie (Institut Calmette, à Marseille) |
|
|
CHU de Nantes |
LES TRÈS RÉCENTS PROGRÈS DE LA THÉRAPIE GÉNIQUE
1. La
réparation des gènes
41(
*
)
À côté de l'approche classique de la thérapie génique, qui consiste à introduire
dans l'organisme une " copie " correcte d'un gène
dysfonctionnel, une
nouvelle technique vient d'apparaître : la réparation
in situ
des
gènes
mutés. Elle serait efficace dans le cas de maladies provoquées par des mutations
génétiques ponctuelles, c'est-à-dire lorsqu'une seule base incorrecte est
responsable
de la production défectueuse d'une protéine. Parmi les pathologies notables
causées par
la mutation d'une seule base figure notamment l'anémie falciforme.
Les nucléotides employés pour la réparation génétique sont des
" chimères " c'est-à-dire des molécules constituées non
plus d'ADN
mais d'ARN-ADN.
Les résultats obtenus
in vivo
sont très encourageants et méritent d'être
confirmés.
2. L'électroporation
in vivo
pour l'injection d'ADN
nu :Améliorer le
transfert du gène grâce à des impulsions électriques
42(
*
)
La génétique et l'électricité n'ont au départ aucun point commun et n'avaient
jusqu'à présent pas mêlé leurs applications. Aujourd'hui, deux équipes de
chercheurs
français du CNRS dirigées respectivement par Luis MIR (Institut Gustave Roussy,
Villejuif) et Daniel SCHERMAN (unité mixte CNRS-Rhône-Poulenc Rorer Vector
Developpment)
rapportent que l'électricité pourrait jouer un rôle de stimulant dans le cadre
de
thérapies géniques en améliorant le fonctionnement des gènes injectés. Au cours
de
leurs expériences, les scientifiques sont parvenus à
" introduire
durablement un gène dans les muscles de souris en obtenant, sous l'action
d'impulsions
électriques, une efficacité au moins cent fois supérieure à la simple injection
d'ADN "
.
Un tel résultat est prometteur car cette technique appliquée aux muscles
pourrait, par
exemple, permettre de traiter des myopathies ou encore d'obtenir une sécrétion
d'hormones.
" Les impulsions électriques rendent perméables les
cellules,
elles ouvrent la porte des cellules pour y faire entrer le produit
choisi "
,
explique Luis MIR.
3. Suivre le chemin des gènes
43(
*
)
" Il est désormais possible de visualiser in vivo le cheminement de
brins d'ADN
grâce à la tomographie à émission de positons.
C'est une avancée méthodologique importante pour le développement de la thérapie
génique.
La fabrication d'une nouvelle molécule aux effets physiologiques prouvés ne
suffit pas
à la promouvoir au rang de médicament.
À cet effet, de multiples expérimentations sont nécessaires. Entre autres, il
faut
pouvoir suivre son trajet sinueux à l'intérieur du corps d'un animal, puis d'un
homme
-un des obstacles auquel se heurte le développement de la thérapie génique.
L'unité INSERM U 334 et le Service Hospitalier Frédéric Joliot de la
Direction
Sciences du Vivant du CEA a Orsay, ainsi que le Service de pharmacologie et
d'immunologie
(CEA/Saclay) viennent de relever le défi.
En utilisant une technique d'imagerie médicale, la tomographie à émission de
positons
(TEP), ils visualisent, dans un organisme vivant, le cheminement
d'oligonucléotides, ces
brins d'ADN ou d'ARN sur lesquels reposent les espoirs de la thérapie génique.
Utilisée en recherche fondamentale et en pharmacologie, la TEP permet la
localisation
corporelle d'une molécule marquée par un radio-isotope. On peut alors mettre en
évidence des fonctions, observer les dégâts causés par un accident vasculaire
grâce
à la mesure du débit sanguin cérébral ou encore localiser des tumeurs en
visualisant
la forte consommation de glucose par des cellules cancéreuses...
Dans leur étude, publiée dans Nature Medicine, les chercheurs se sont
intéressés aux
oligonucléotides, molécules qui agissent au sein des cellules. Selon leur
nature, les
oligonucléotides permettent à certaines cellules malades de synthétiser une
protéine
dont elles sont dépourvues, ou ils s'opposent à l'action d'une protéine
défectueuse,
soit en empêchant sa fabrication, soit en perturbant son fonctionnement.
Actuellement, il est possible de vérifier le chemin emprunté par ces molécules à
l'intérieur d'un organisme, mais uniquement chez des animaux de laboratoire et
par des
méthodes longues et laborieuses. La nouvelle technique permet de réduire le
temps
nécessaire à ces études de manière considérable : une fois les
oligonucléotides
radiomarqués, les chercheurs peuvent suivre leur trajet dans un organisme
vivant en
quelques heures seulement. Ils ont directement accès à une image de tous les
organes, ce
qui limite le recours à l'expérimentation animale. Non invasive et sensible,
cette
technique pourrait être applicable à l'homme. "
1.2.3. LES NOUVEAUX VACCINS
1.2.3.1. L'immunothérapie génique
Le principe de
l'immunothérapie utilisée contre les tumeurs est de stimuler le système
immunitaire
afin de s'opposer à la croissance des cellules cancéreuses.
Les tumeurs se développent généralement à partir de cellules dans lesquelles
s'accumulent des anomalies actuellement identifiées concernant des gènes codant
pour des
protéines impliqués dans la différenciation, la prolifération cellulaire ou le
contrôle du cycle.
Certaines de ces protéines sont des facteurs de croissance comme le
fibroblast growth
factor
(FGF), des récepteurs comme Her-2/neu, des tyrosine-kinases comme
abelson
(ABL), des facteurs de transcription comme myc ou des suppresseurs de tumeurs
comme la
protéine codée par le gène P 53.
La surexpression de protéines normales ou la production de protéines anormales
peuvent
conduire à une présentation antigénique de ces produits à la surface des
cellules
tumorales ; elles peuvent alors être reconnues par le système immunitaire.
" L'utilisation de cellules dendritiques
44(
*
)
,
préalablement incubées avec les protéines ou les peptides tumoraux, ou dans
lesquelles
auront été introduits les gènes codant les peptides est particulièrement
prometteuse.
Des cellules tumorales modifiées par les gènes codant la molécule B 7
ou des
cytokines comme le GM-CSF (
granolocyte macrophage colony stimulating
factor
) ou
l'IL 2 (interleukine 2) pourraient être également injectées aux
patients.
La GM-CSF sécrétée peut activer et recruter des cellules présentant l'antigène
tandis
que la présence de la molécule B 7 et de l'IL 2 peut stimuler les
réponses
lymphocytaires. Enfin, il serait possible de restaurer la présentation des
antigènes
tumoraux en injectant aux patients de l'ADN codant des antigènes tumoraux ou
encore les
molécules HLA
(human leucocyte antigen
) ".
45(
*
)
1.2.3.2. La vaccination génique
Il s'agit
d'une vaccination à base d'ADN " nu "
. Les vaccins à
ADN sont le
résultat d'une découverte fortuite. En 1988, une équipe de chercheurs de
l'Université
du Wisconsin en collaboration avec la société Vical travaillait sur la
pénétration de
l'ADN de plasmide dans les cellules, dans un but de thérapie génique. À leur
grande
surprise, de l'ADN " nu " (qui n'est inclus dans aucun
organisme)
simplement injecté en solution saline dans les cellules musculaires, s'est
montré
capable de s'exprimer, produisant les protéines correspondantes, mais sans
s'intégrer au
génome humain. C'est sur cette capacité que repose le principe de la
vaccination à ADN.
Elle consiste à introduire dans l'organisme animal ou humain une partie du
matériel
génétique de l'agent pathogène. Dans le cas du matériel génétique du virus
contre
lequel on recherche l'immunisation, on prélève la fraction d'ADN codant pour la
protéine susceptible de déclencher une réaction immunitaire protectrice
(antigène). On
l'introduit ensuite dans un plasmide (fragment d'ADN circulaire) que l'on fait
se
multiplier dans des bactéries. Après extraction et purification, celui-ci est
introduit
dans l'organisme où il est capable de pénétrer dans les cellules. L'ADN du
pathogène
s'exprime alors dans le noyau des cellules. Il y a production d'antigène ;
celui-ci
est présenté au système immunitaire et déclenche une réponse. L'antigène viral
provoque une double réponse immunitaire. D'une part, la production d'anticorps
capables,
lors d'une infection, de reconnaître spécifiquement cet antigène sur le
virus ;
d'autre part, l'apparition de lymphocyte T cytotoxiques (CTL) dont le rôle
est de
détruire les cellules infectées par le virus.
Cette technique peut théoriquement permettre de vacciner contre toutes les
maladies
infectieuses.
Les avantages de la vaccination à ADN sont multiples :
- la réponse immunitaire provoquée est de longue durée ;
- il n'y pas de risque d'infection par un agent adventice puisque le
vaccin est
composé d'ADN. Il n'y pas d'effets secondaires ;
- le vaccin peut être " multicible " : on peut
réaliser
des " cocktails " de vaccins où plusieurs gènes, codant
pour des
protéines de différents pathogènes seraient introduits en même temps, ce qui
rendrait
inutiles des injections multiples. Certains vaccins
" multicibles "
existent actuellement (DT polio, par exemple) mais d'autres sont
irréalisables à
cause d'incompatibilités entre les préparations ;
- le vaccin à ADN est chimiquement défini et thermiquement stable ce qui réduit
la
nécessité de maintenir la chaîne du froid ;
- sa préparation est standardisée : le procédé reste le même quelle que
soit la
maladie. Seul change le fragment d'ADN du pathogène à cloner dans le plasmide.
D'où une
économie d'échelle pour la fabrication et des coûts moindres.
Chez l'animal, les vaccins à ADN ont donné des résultats probants pour un grand
nombre
de maladies, notamment la grippe chez les primates, le paludisme, le VIH chez
la souris.
Toutefois, en l'état actuel des choses, plusieurs problèmes se posent :
- si le plasmide étranger s'intègre à l'ADN de la cellule hôte en certains
endroits, on ne peut écarter l'hypothèse qu'il active un oncogène, gène
déclencheur
de cancer ou, à l'inverse, inhibe l'action d'un gène suppresseur du cancer.
Même si ce
risque semble très théorique aux chercheurs, il doit être très rigoureusement
évalué ;
- les connaissances des mécanismes entrant en jeu lorsqu'on injecte l'ADN
doivent
être approfondies. En effet, si l'on a la preuve que le plasmide pénètre bien
dans le
noyau des cellules musculaires, puisque la protéine produite est retrouvée à
l'intérieur de ces mêmes cellules, on ne sait pas encore très bien comment le
système
immunitaire prend connaissance de sa présence.
Normalement, la réaction du système immunitaire est provoquée par la
" présentation " de l'antigène par des cellules
spécialisées.
Celles-ci incorporent les substances étrangères qui pénètrent dans l'organisme
et en
montrent des fragments à leur surface pour informer le reste du système. Cette
présentation nécessite l'intervention de molécules dites de classe I et II
du
complexe majeur d'histocompatibilité. Or, leur présence à la surface des
cellules
musculaires est loin d'être établie. Il se pourrait que la présentation de
l'antigène
soit réalisée par les cellules de Langerhans situées, entre autres, dans la
peau.
L'efficacité du " pistolet à gènes "
46(
*
)
qui, projette l'ADN vers le derme, est en faveur de cette hypothèse.
- Il faut également améliorer les formes d'administration des vaccins à
ADN par
voie nasale et orale. En effet, une bonne immunité au niveau des muqueuses est
indispensable pour se défendre, par exemple, contre le VIH. Or il n'est pas
évident
qu'une injection dans le muscle puisse déclencher une réponse au niveau de
l'estomac, de
l'intestin, des voies respiratoires, de l'appareil génital, etc.
Le vaccin contre la grippe prouve qu'il est possible d'avoir une bonne
protection contre
une maladie respiratoire, via, probablement des anticorps transportés par le
sang
jusqu'au site d'infection. Mais dans la majorité des cas, la réponse risque de
ne pas
être optimale : l'encapsulation de l'ADN, au moyen, par exemple de
liposomes,
permettant son administration par voie nasale ou orale et facilitant sa
pénétration au
niveau des muqueuses doit être perfectionnée. Il ne s'agirait plus alors d'ADN
vraiment
nu.
- Si les résultats obtenus sur les animaux sont probants, l'efficacité du
vaccin à
ADN chez l'homme n'est pas encore prouvée.
En 1998, des résultats ont été publiés pour cinq essais cliniques :
paludisme (US Navy/Vical/Pasteur Mérieux Connaught) ;
grippe (Merk) ;
HBV (Glaxo Welcome /PowderJect)
HIV (David Weiner / Apollon)
HIV (Britta Wahren).
Les chercheurs ont conclu à la bonne tolérance des vaccins à ADN et à une
réponse
immunitaire jugée " satisfaisante ".
Les premiers essais cliniques de phase II, pour la grippe et le paludisme
pourraient
débuter l'année prochaine.
Ils devraient permettre une meilleure évaluation de l'efficacité des vaccins à
ADN
ainsi que des doses à administrer à l'homme pour que l'immunisation soit
suffisante (si
cette quantité est trop importante, la vaccination à ADN risquerait en effet de
ne pas
être économiquement envisageable).
LE DERNIER ÉTAT DES RECHERCHES
Une équipe de
recherche regroupant notamment des chercheurs du National Marine Research
Center, du
Centre de Recherche Médical sur les Maladies Infectieuses de l'Armée de Terre
américaine, de la firme américaine Vical et de Pasteur Mérieux Connaught (groupe
Rhône-Poulenc), publie dans la revue Science du 16 octobre 98 les résultats des
essais
d'un nouveau vaccin à ADN nu contre le
paludisme
(ou encore
malaria
). Ces
essais ont été menés sur des sujets sains et portent sur l'innocuité et
l'immunogénicité, c'est-à-dire la réponse immune des lymphocytes T cytotoxiques
(CTL)
" Killer "
.
Le terme de vaccin à ADN nu fait référence à l'administration des plasmides
eux-mêmes. Selon l'article publié, la plupart des 20 sujets vaccinés avec ce
vaccin à
ADN contre le paludisme ont développé une réponse de variabilité du dosage des
CTL. En
se fondant sûr ces résultats prometteurs, les chercheurs étudient actuellement
l'action
préventive de ce vaccin.
Ces corecherches ont pour objectif le développement d'un vaccin à ADN avec pour
modèle
le paludisme, qui est une maladie infectieuse touchant de nombreux soldats de
l'Armée de
Terre américains. C'est la première fois que l'on publie les effets d'un vaccin
du
paludisme sur des sujets sains. De son côté, Vical entre dans les phases I
et II de
développement, notamment de " Allovectin 7 "
" Leuvectin " un vaccin à base de complexes ADN-lipides
adaptés aux
cellules
cancéreuses
ou encore de Vaxid, un vaccin à ADN à de type plasmide. Pour sa
part,
Pasteur Mérieux Connaught a acquis la licence sur la commercialisation de
vaccins à ADN
pour certaines
maladies infectieuses
, et qui sont développés par Vical.
Source : Vigie Médecine Pharmacie . N° 39 février 1999.
|
LES TESTS DES VACCINS À ADN 47( * ) Ce tableau répertorie certains des tests cliniques qui évaluent l'innocuité et l'efficacité immunitaire des vaccins à ADN. Tous les vaccins testés ont été bien tolérés, et les déterminations d'efficacité sont en cours. |
||
|
Objectif |
Protéines codées par les gènes |
Résultats |
|
- Prévention de l'hépatite B
|
- Antigène de surface de l'hépatite B |
- Réactions humorales et cellulaire |
|
- Prévention de l'herpès |
- Glycoprotéine de l'herpès |
- Analyses immunologiques en cours
|
|
- Prévention du SIDA |
- Protéines de l'enveloppe et de régulation, ou protéines de la capside et enzymes de réplication
|
- Réactions cellulaires (l'ensemble des gènes sera probablement testé dans un seul vaccin) |
|
- Prévention de la grippe
|
- Hémaglutinine |
- Analyses immunologiques en cours (l'essai est terminé)
|
|
- Prévention du paludisme
|
- Protéine d'une des formes du parasite |
- Réactions cellulaires |
|
- Thérapie du SIDA |
- Protéines de l'enveloppe et régulatrices, ou protéines TAT, NEF et régulatrices |
- Réactions humorales
dans le premier essai.
|
|
- Thérapie du SIDA |
- Protéines de l'enveloppe, régulatrices et de la capside, et enzymes impliquées dans la réplication du VIH
|
- Ce vaccin a été associé à une trithérapie, analyses immunologiques en cours |
|
- Thérapie des adénocarcinomes du sein et de l'intestin
|
- Antigène carcino-embryonnaire |
- Réactions cellulaires |
|
- Thérapie des lymphomes des lymphocytes B
|
- Immunoglobuline |
- Réactions humorales |
|
- Thérapie des lymphomes cutanés des lymphocytes T
|
- Récepteur de lymphocyte T |
- Analyses immunologiques en cours (l'essai est terminé) |
|
- Thérapie du cancer de la prostate
|
- Antigène spécifique de la membrane prostatique |
- Analyses immunologiques en cours |
1.2.3.3. L'utilisation de la connaissance du génome pour la découverte de nouveaux vaccins " traditionnels "
Les progrès
pouvant être réalisés dans la découverte de nouveaux vaccins sont liés à la
connaissance progressive du génome (c'est-à-dire de l'ensemble des gènes) des
bactéries et, bientôt, des parasites.
Cette connaissance permet d'identifier les composantes les plus pertinentes
pour un
vaccin, c'est-à-dire celles qui entraînent une réponse immunitaire.
L'identification des gènes d'une bactérie a pour corollaire la connaissance des
protéines codées par ces gènes. Or ces protéines constituent des
antigènes
48(
*
)
potentiels qu'il convient de
tester.
La connaissance du génome a donné aux chercheurs la possibilité de fabriquer les
multiples protéines d'une bactérie, un gène constituant en quelque sorte la
recette de
confection d'une protéine.
Les chercheurs produisent les protéines d'une bactérie qui, en qualité
d'antigènes
potentiels, sont considérés comme d'éventuels candidats vaccins.
Si l'une de ces protéines est un antigène d'intérêt, elle déclenchera, lors de
son
injection dans un organisme, une réponse immunitaire protectrice ; cet
organisme,
sera, à l'avenir, immunisé contre l'infection dont est responsable la bactérie.
On peut donner deux exemples très récents de l'utilisation de la connaissance
des
génomes des bactéries pour la mise au point de vaccins.
L'ulcère de l'estomac
En juillet 1997,
a été publiée la séquence complète du génome de
Helicobacter pylori
,
bactérie responsable des ulcères de l'estomac. Cette découverte a fourni des
informations globales sur les possibles facteurs de virulence, le métabolisme
de la
bactérie, l'organisation du génome. Elle a surtout fourni des outils de
recherche très
intéressants pour l'identification des protéines codées par les gènes de
Helicobacter
Pylori
, en particulier de celles permettant à la bactérie de survivre, se
multiplier
et s'implanter au niveau de la muqueuse gastrique. Elle a également permis la
mise en
place par les industriels (Astra et Pasteur-Mérieux-Connaught/OraVax) de
stratégies
d'envergure visant à identifier de façon systématique des antigènes protecteurs
et des
cibles thérapeutiques d'intérêt.
La recherche d'antigènes protecteurs est passée par l'identification des
protéines
spécifiques à
Helicobacter pylori
qui sont des antigènes potentiels.
Les gènes ont été amplifiés par PCR (
polymerase chain reaction
), clonés
et
introduits dans des souches bactériennes. Ces bactéries ont produit des
protéines qui
ont été purifiées et dont le pouvoir " protecteur " a été
testé
chez la souris
49(
*
)
.
Ayant travaillé sur l'implantation de
Helicobacter pylori
au niveau de
la muqueuse
gastrique, la société Astra a annoncé que les premiers tests de vaccin contre
l'ulcère
de l'estomac sur des volontaires commenceraient dans les mois à venir. Le
vaccin mis au
point stimulerait le système immunitaire pour qu'il crée des anticorps
empêchant les
bactéries
Helicobacter pylori
de se fixer dans la muqueuse de l'estomac.
Pasteur-Mérieux-Connaught/OraVax a déjà réalisé des essais de phase I/II.
La tuberculose
Des
scientifiques de l'Institut Pasteur ont récemment identifié un gène de
virulence du
bacille de la tuberculose. L'inactivation de ce gène atténue le pouvoir
pathogène du
bacille.
50(
*
)
.
Malgré les médicaments existants et la vaccination par le BCG, la tuberculose
continue
ses ravages. L'incidence de la maladie augmente à la fois dans les pays en
développement
et dans les pays industrialisés. Au cours des dix prochaines années, on estime
que 90
millions d'adultes seront touchés par la maladie. L'apparition de souches
résistantes
aux antibiotiques et l'association de
Mycobacterium tuberculosis
avec
le VIH
font de cette maladie en recrudescence un problème majeur de santé publique.
La tuberculose est due à des bactéries de la famille des mycobactéries :
Mycobacterium
tuberculosis, Mycobacterium bovis et Mycobacterium africanum.
La virulence
de ces
agents, c'est-à-dire leur pathogénicité, dépend de leur capacité à se multiplier
chez l'hôte.
À l'Institut Pasteur, l'unité de Génétique mycobactérienne dirigée par Brigitte
GICQUEL, cherche notamment à identifier les facteurs de virulence du bacille de
la
tuberculose. Si l'on inactive ces gènes, on pourra espérer obtenir un nouveau
vaccin
vivant atténué. Si le BCG permet de diminuer le nombre de nouveaux cas, s'il
empêche
les formes graves de la maladie chez les jeunes enfants, l'immunité conférée
par le
vaccin diminue progressivement après une dizaine d'années.
La connaissance récemment acquise du génome de
Mycobacterium
tuberculosis
a
permis l'identification d'un gène de virulence de ce bacille : il s'agit
du gène
erp
,
codant une protéine de la surface nécessaire à la multiplication du bacille
dans les
cellules hôtes. Des souches " mutantes " de
Mycobacterium
tuberculosis
et de la souche vaccinale
Mycobacterium bovis
BCG chez
lesquelles
le gène
erp
a été inactivé ont été construites. Les résultats montrent
que
l'inactivation du gène
erp
, en supprimant la production de la protéine de
surface, atténue considérablement la multiplication de
Mycobacterium
tuberculosis
et
de
Mycobacterium bovis
dans des macrophages en culture et chez la
souris.
La réintroduction de
erp
dans les souches mutantes restaure leur
capacité de
multiplication.
Ces résultats suggèrent que le gène
erp
pourrait être un bon candidat
pour
l'atténuation de la virulence de
Mycobacterium tuberculosis
et pour
l'élaboration
de nouveaux vaccins contre la tuberculose, en partie des vaccins vivants
atténués.
Ces travaux ouvrent une voie nouvelle pour l'étude des mécanismes de la
pathogénicité
des mycobactéries et pour la mise au point de nouveaux vaccins contre la
tuberculose qui
tue encore plus de 3 millions de personnes chaque année dans le monde
DE L'UTILITÉ DE TROUVER DE NOUVEAUX VACCINS... 51( * )
Liste non
exhaustive des pathogènes non encore couverts par une vaccination :
Chlamydia sp.
Coccidioides immitis
Cryptoccoccus neoformans
Cytomegalovirus
Dengue
Entamoeba histolytica
Enterotoxigenic Escherichia coli
Epstein-Barr virus (EBV)
Group A streptococcus
Haemophilis influenzae non typable
Hepatitis C virus (HCV)
Hepatitis D
Hepatitis E
Herpès simples virus types 1 et 2
Histoplasma capsulatum
Human Immune Deficiency virus HIV-1
Human Immune Deficiency virus HIV-2
Human papillomavirus
Legionella pneumophila
Leishmania sp.
Moraxella catarrhalis
Mycoplasma pneumoniae
Neisseria gonorrheae
Parainfluenza virus
Plasmodium spp.
Pseudomonas aeruginosa
Pseudomona cepacia
Respiratory syncytial virus
Rickettsia rickettsii
Schistosoma mansoni
Shigella
Toxoplasma gondii
Treponema pallidum
1.2.4. LA PHARMACOGÉNOMIQUE
" Right drug, right person, right time " (le bon médicament, à la bonne personne, au bon moment) 52( * )
1.2.4.1. Définition
La
pharmacogénomique est l'étude des mécanismes génétiques des variations
individuelles
de la réponse aux xénobiotiques et, plus particulièrement, aux médicaments. Ces
connaissances sont appliquées ou applicables à l'adaptation de certains
traitements à
chaque patient.
Sur les trois milliards de bases du génome humain, 99,9 % sont identiques
d'un
individu à l'autre. Les variations ou polymorphismes des 0,1 %,
pourcentage pourtant
infime, mais représentant trois millions de nucléotides, sont à la base des
différences entre les individus.
Parmi celles-ci figure la sensibilité aux
médicaments
. On entend par là que, pour une dose donnée, la réponse des
individus
est plus ou moins forte, excessive chez certains et faisant courir un risque de
toxicité,
insuffisante chez d'autres et pouvant entraîner l'inefficacité du traitement.
Parfois,
le changement est qualitatif :
- il n'y a aucune réponse (insensibilité) ;
- la réponse est différente de celle attendue.
On voit l'importance que cela peut revêtir en clinique et l'intérêt que
présente la
possibilité d'identifier les malades s'écartant des normes habituelles.
Le mécanisme d'interposition entre le gène et la réponse de l'organisme a deux
origines
possibles :
L'une pharmacodynamique, s'il s'agit d'une modification du récepteur auquel le
médicament vient se lier.
L'autre, pharmacocinétique, s'il s'agit d'une modification du métabolisme de
celui-ci
liée à une différence dans l'équipement enzymatique.
En dehors des atypies entraînant des variations dans l'activité d'une enzyme,
il faut
souligner la variabilité due à la réalité de notre unicité individuelle. Par
exemple,
les cytochromes hépatiques, enzymes oxydant les xénobiotiques et jouant un rôle
central
dans leur métabolisme, peuvent exister chacun sous plusieurs centaines de
variétés,
appelés iso-enzymes ;
Le terme " pharmacogénétique " est apparu dès la fin des
années
cinquante pour désigner les " modifications des réponses
pharmacologiques sous
l'influence de l'hérédité ". À cette époque, l'analyse génétique
n'était
possible que par l'analyse des expressions phénotypiques, en particulier des
différences
interindividuelles dans l'équipement enzymatique et ses conséquences sur le
métabolisme
des produits. Dans un certain nombre de cas, devenus classiques, tels la
sensibilité à
la succinyldicholine ou le métabolisme de l'isoniazide, l'identification du
phénotype
par des tests biologiques a pu conduire à des applications cliniques encore
utilisées
aujourd'hui.
La différence entre pharmacogénétique et pharmacogénomique est claire. La
pharmacogénomique s'adresse au gène lui-même et non plus seulement à son
expression.
Elle englobe la pharmacogénétique et la renouvelle en identifiant les
variations du
génome responsables des modifications des réponses de l'organisme. Lorsque les
liens
entre les mutations d'un ou plusieurs gènes et leurs traductions au niveau
d'une enzyme
ou d'un récepteur, ainsi que les conséquences cliniques de celles-ci, sont
établis,
l'analyse du génome, désormais rapide et sûre, permet d'éviter le recours à des
dosages et à des tests biologiques souvent longs, délicats, parfois imprécis et
toujours indirects.
1.2.4.2. Les applications de la pharmacogénomique
Des traitements adaptés aux patients
- La
maîtrise de la toxicité des traitements
. La pharmacogénomique a des
applications
immédiates dans le traitement des leucémies et des greffes d'organes. Aux
États-Unis,
la Mayo Clinic et la société Variagenics ont mis au point, au printemps 1998,
un test de
dépistage d'une infime anomalie génétique qui, par l'intermédiaire de la
production
d'une enzyme (la TPMT), freine le métabolisme de deux médicaments :
l'azathiopurine
et le 6-mercaptopurine. Les patients atteints de cette variation génétique
peuvent être
mortellement empoisonnés par ces médicaments. Or, les médicaments de la famille
des
thiopurines permettent d'atteindre un taux de rémission de 80 % dans le
cas des
leucémies lymphoblastiques. On ne peut donc renoncer à les utiliser. La
solution est de
faire passer un test génétique aux patients susceptibles d'être traités afin de
donner
à ceux qui présentent cette variation génétique des doses inférieures aux doses
standard.
Autre exemple, le laboratoire Abbott a signé avec le société Genset un contrat
de
coopération de recherche pour un traitement contre l'asthme. Il s'agit
d'identifier les
gènes responsables de désordres hépatiques observés chez certains patients
prenant du
Zyflo, afin de mettre au point un test de dépistage. Les sujets susceptibles
d'avoir ces
problèmes de toxicité hépatique seraient écartés de ce type de traitement dès le
départ alors qu'actuellement, il faut surveiller l'ensemble des patients
pendant toute la
durée du traitement.
-
La détermination de la résistance à certains médicaments
Les chercheurs des laboratoires ont déjà découvert de nombreuses variations
génétiques modifiant la production de cytochromes et entraînant une résistance
des
patients aux antidépresseurs et aux médicaments permettant de traiter les
psychoses ou
de réguler le rythme cardiaque.
À l'hôpital Bichat sont étudiées les mutations du génome du VIH pouvant induire
une
résistance aux médicaments antiviraux.
- L'évaluation des résultats positifs des traitements
À l'hôpital Broussais, les médecins établissent des corrélations entre les
variations
génétiques des patients et les bénéfices réels observés des traitements contre
le
cholestérol ou l'hypertension.
En 1998, des chercheurs canadiens et hollandais ont mis en évidence un
polymorphisme
permettant d'indiquer si un traitement à base de Pravastatin, du laboratoire
Bristol-Myers Squibb, bénéficiera réellement au patient.
Des scientifiques de la société Myriad Genetics Inc. ont trouvé une variation
génétique expliquant pourquoi le sel augmente la pression sanguine chez
certaines
personnes seulement et permettant donc d'évaluer l'efficacité d'un éventuel
régime
hyposodé.
- L'utilisation de la pharmacogénomique en chirurgie
On peut citer deux exemples :
- le dépistage génétique d'un polymorphisme fréquent dans la population
permet de
diminuer le risque thrombotique post opératoire ;
- les modalités d'intervention chirurgicale des sujets porteurs de cancers
colorectaux dépendent en partie de l'existence d'une mutation sur un gène de
susceptibilité.
En conclusion, en référence aux maladies cancéreuses, il est clair,
aujourd'hui, que
les altérations génétiques présentes dans les cellules cancéreuses
conditionnent leur
comportement face à la radiothérapie et la chimiothérapie. Demain, les
traitements
anticancéreux prendront en compte non seulement la constitution génétique du
patient
mais aussi celle de sa tumeur, conduisant à un véritable traitement
" sur
mesure " minimisant les risques et optimisant l'efficacité.
À terme, la pharmacogénomique permettra de prescrire les médicaments appropriés
aux
malades répondant bien et d'éviter l'usage de certains médicaments chez les
personnes
réfractaires ou répondant mal au traitement.
On peut espérer, en adaptant les traitement en fonction de certaines
caractéristiques
génétiques, améliorer leur efficacité : aujourd'hui, un grand nombre de
patients
répondent insuffisamment à des médicaments tels que les bêtabloquants,
l'interféron,
ou les inhibiteurs de l'enzyme de conversion de l'angiotensine.
La pharmacogénomique devrait permettre d'améliorer cette situation.
La recherche de médicaments
La
pharmacogénomique ne se contentera pas de modifier l'administration du
médicament en
fonction des réponses des patients ; en fait elle est appelée à jouer un
rôle
important à toutes les étapes de la vie du médicament.
- Le ciblage des essais
Aujourd'hui, 30 % environ des nouvelles molécules n'atteignent pas la
phase III
des essais cliniques
à cause d'effets secondaires indésirables
.
La découverte de ces effets à une phase avancée des essais cliniques génère un
énorme gâchis de temps et d'argent.
Le recours à la pharmacogénomique permettra de sélectionner pour les essais des
personnes présentant la plus grande variété de réponses à un médicament et de
réduire ainsi la taille de l'échantillon représentatif. On évitera ainsi les
mauvaises
surprises dues à des effets secondaires n'apparaissant que tardivement, au fur
et à
mesure que le nombre de patients impliqués dans les essais augmente.
L'estimation
précoce de l'efficacité et de l'innocuité d'un médicament est un élément
important
de réduction de la durée et des coûts de la recherche pharmaceutique.
On pourra en même temps évaluer le taux d'incidence de ces effets secondaires.
En raison
des variations génétiques individuelles, il est possible que le médicament
n'ait des
effets indésirables que pour un très faible pourcentage d'individus et soit
entièrement
efficace et sans danger pour le reste de la population.
LA PHARMACOGÉNOMIQUE DANS LE PROCESSUS DE DÉVELOPPEMENT DU PRODUIT PHAMARCEUTIQUE
|
DÉVELOPPEMENT |
|
COMMERCIALISATION |
||||||||||||
|
Phase I |
Phase III |
Phase IV |
||||||||||||
|
Choix d'une molécule " candidate " |
Études précliniques |
Dépôt de la demande d'autorisation d'essais cliniques |
Préparation du dossier de demande d'autorisation de mise sur le marché |
Autorisation de mise sur le marché |
Pharmacovigilance
|
Nouvelles indications thérapeutiques |
||||||||
|
|
Début du projet |
Plan de développement de la nouvelle molécule |
Début des essais cliniques |
Dépôt du dossier de demande d'AMM |
Lancement du médicament |
Développement de nouvelles formules galéniques |
||||||||
|
|
RECOURS À LA PHARMACOGÉNOMIQUE |
|||||||||||||
Source : Millennium Predictive Medicine. C. Van HUFFEL.
- La
récupération de molécules délaissées
Actuellement, sur dix molécules entamant la phase I des essais cliniques,
une seule
parvient sur le marché. La pharmacogénomique peut améliorer cette situation en
démontrant que certains candidats médicaments, s'ils sont inutiles ou dangereux
pour
certains patients, peuvent être utilisés pour d'autres ; en fonction des
polymorphismes, ils peuvent avoir des applications insoupçonnées.
Dans le cas de la maladie d'Alzheimer, des recherches menées au Canada ont
montré que le
gène Apolipoprotéine E était un moyen d'évaluation des médicaments
potentiels.
La mutation de ce gène est responsable, par exemple, pour plus de 60 % des
patients,
de l'échec de la tacrine, administrée aujourd'hui sans discrimination aux
personnes
souffrant de la maladie d'Alzheimer.
Mieux encore, ces travaux génétiques ont également permis à des chercheurs
lillois de
découvrir que ces malades, dont le gène Apolipoprotéine E muté, entraîne
une
résistance à la tacrine, réagissent bien, en revanche, à une molécule
expérimentale
(S 12024) que l'on s'apprêtait à " abandonner " pour
cause
d'inutilité...
- Les tests génétiques
La mise au point de ces tests est appelée à se développer pour optimiser et
réduire
les risques de molécules déjà connues et en trouver de nouvelles.
L'idée, à long terme, est de faire passer un test à un malade avant de lui
donner un
traitement afin que lui soient prescrits les médicaments les mieux adaptés à
son profil
génétique.
D'ores et déjà, la société américaine Affymetrix a mis au point, avec le centre
médical de l'Université de Georgetown, et commercialisé la première puce à ADN
spécifiquement destinée à une utilisation en pharmacogénomique. Elle est
capable de
détecter dix-huit altérations connues de deux gènes humains codant pour des
enzymes
impliqués dans le métabolisme de médicaments tels que des bêtabloquants
(anti-hypertenseurs) et certains antidépresseurs et anticonvulsifs.
1.2.4.3. Les différentes approches de la pharmacogénomique
La
pharmacogénomique génère deux options de stratégie de recherche-développement
pour
les firmes pharmaceutiques :
- soit elles optent pour des études menées, en interne, par leur propres
services de
recherche. Cette solution assez coûteuse a été choisie par Bristol-Myers Squibb,
Novartis, Glaxo et Pfizer.
- soit elles externalisent ces recherches et passent des contrats avec des
entreprises de
biotechnologie spécialisées dans la pharmacogénomique. Les plus importantes
sont les
suivantes :
|
COMPAGNIE |
STRATÉGIE |
|
VARIAGENICS
|
Recherche de
polymorphismes dans les gènes impliqués dans le cheminement
des médicaments dans l'organisme.
|
|
GENSET
|
Création d'une carte du
génome montrant la localisation de
60 000 polymorphismes.
|
|
INCYTE PHARMACEUTICALS
|
Recherche des
polymorphismes pour chaque gène isolé.
|
|
MILLENNIUM PHARMACEUTICALS
|
A créé la
" médecine prédictive ", auxiliaire de
l'utilisation des variations génétiques pour améliorer la recherche de
médicaments.
|
|
GENENTECH
|
Commercialise un médicament contre les cancers du sein qui est efficace seulement chez les 25 à 35 % de la population qui ont une variation génétique particulière. |
|
|
(53( * )) |
Les approches
les plus originales sont celles qu'ont choisi Variagenics, Millenium
Pharmaceuticals et
Genset.
- Variagenics s'intéresse aux variations génétiques impliquant des
différences
d'expression des protéines que rencontre un médicament lors de son cheminement
dans
l'organisme. Le niveau d'expression de ces protéines modifie l'efficacité ou la
toxicité d'un produit :
" lorsqu'on prend un médicament, il
touche 30
à 40 protéines différentes avant de quitter le
corps "
.
54(
*
)
Les chercheurs de Variagenics ont identifié plus de 6000 gènes impliqués
dans le
cheminement des médicaments et, en mai 1998, ils ont mis en place des essais
cliniques
pour étudier les différences génétiques entre les patients atteints d'un cancer
du
colon et soignés avec du 5-Fluorouracil.
- Chez Millenium Pharmaceuticals, dans la division de Médecine Prédictive,
les
chercheurs comparent les gènes activés dans plusieurs types de cellules
cancéreuses
afin d'identifier ceux qui correspondent aux formes agressives des différents
cancers.
Être capable de déterminer si un cancer croît lentement ou rapidement à des
conséquences énormes pour le traitement que doit suivre le patient. Millenium
recherche
donc à connaître les gènes dont la présence signe, par exemple, la certitude
d'une
métastase.
" Millenium a trouvé un gène, dans les cellules du mélanome, qui
agit
comme une boule de cristal. Lorsqu'il est là, le cancer ne forme pas de
métastases ; mais quand il est absent, le cancer devient
tueur "
1
.
- Genset, société française dont le siège est au Génopole d'Évry, a élaboré
une carte à haute résolution du génome humain. La démarche est une approche
génomique
globale qui permet de comparer les séquences d'ADN d'individus n'appartenant pas
forcément à la même famille (ce qui facilite l'opération) et présentant certains
traits cliniques (malades, non malades, répondeurs, non répondeurs) afin de
localiser
les polymorphismes à l'origine de ces traits.
Les différentes formes que peut prendre un gène s'appellent des allèles ;
la carte
établie par Genset s'appelle
" carte à haute résolution de
marqueurs
bi-alléliques "
. Pour la réaliser, les chercheurs ont placé des
jalons
(environ 60 000 " marqueurs ") sur les zones
sensibles de
l'ADN. En comparant des régions du génome de malades très réceptifs à un
traitement
à celles de patients réfractaires, ils veulent parvenir à localiser la variation
génétique concernée et à l'identifier.
1.2.4.4. Espoirs et limites de la pharmacogénomique
L'hypothèse de
base de la pharmacogénomique est d'obtenir par l'établissement préalable du
profil
génétique d'un individu, la possibilité de choisir les médicaments pouvant lui
être
profitables, d'éviter ceux qui pourraient lui être nuisibles et d'adapter la
posologie
à son cas particulier. Ceci suppose l'existence de tests rapides (pour pouvoir
être
effectués avant l'institution du traitement), fiables et peu coûteux. À la
limite, ce
profil pourrait être établi dès la naissance et renouvelé périodiquement pour
tenir
compte de l'apparition des nouveaux médicaments.
" Cette hypothèse est légitime dans la mesure où elle justifie les
efforts
et les investissements dans ce secteur. Il n'est cependant pas illégitime de la
tempérer
par quelques considérations.
Tout d'abord, les facteurs génétiques ne sont pas les seuls à influencer les
réponses
aux médicaments. Les facteurs acquis ne sont pas à négliger.
Ensuite, on doit s'interroger sur la force de la liaison entre le gène et la
réponse.
C'est le problème classique des relations génotype-phénotype et
phénotype-réponse.
L'intérêt d'un test en dépend étroitement. Ce lien est certainement très fort et
pertinent dans certains cas, beaucoup moins dans bien d'autres.
Il convient également de considérer l'impact clinique de la modification
génétique.
Seul celui-ci compte en pratique. Par exemple, une variation d'activité
enzymatique ou de
taux plasmatique du médicament de 50 % n'a d'intérêt que si elle entraîne
des
conséquences en termes d'efficacité ou de tolérance. Or, l'indifférence
thérapeutique
est la règle et non l'exception et les armoires des pharmacologues sont
encombrées de
publications de variations de métabolisme dépourvues de toute incidence
clinique.
Enfin, l'exemple de la pharmacogénétique montre que beaucoup de recherches ont
débouché sur peu d'applications bien qu'elle soit plus proche du phénotype et
de la
réponse que la pharmacogénomique (il est vrai que la pharmacogénomique peut
être plus
précise et plus simple à mettre en oeuvre).
On peut donc légitimement se demander si les applications de la
pharmacogénomique, loin
d'être ubiquitaires, ne se limiteront pas à un certain nombre de médicaments et
de
circonstances particulières, dont le critère de sélection sera la pertinence
clinique.
On retrouverait alors un instrument de diagnostic biologique, puissant certes,
dont
l'indication serait à discuter dans le cadre d'une stratégie thérapeutique. Ce
qui
évidemment, n'enlèverait rien à l'intérêt de la pharmacogénomique et à l'urgence
d'investir dans ce domaine. "
55(
*
)
1.2.5. 1.2.5. LE DIAGNOSTIC MOLÉCULAIRE
Le diagnostic
moléculaire fait appel aux technologies issues en particulier de la biologie
moléculaire
(étude des molécules qui constituent la cellule et des processus moléculaires
qui
régissent son fonctionnement). Il repose sur l'utilisation de tests ciblant
directement
le patrimoine génétique. Ceux-ci sont notamment réalisés grâce à des puces à
ADN.
La dépense globale du diagnostic médical, est chiffrée à 25 milliards de
dollars
par an ; 10 à 20 % de l'ensemble des analyses seront vraisemblablement
réalisés sur des biopuces en 2005.
Le diagnostic moléculaire couvre deux champs distincts : les biopuces
permettent de
détecter les maladies infectieuses et les maladies génétiques.
1.2.5.1. Le diagnostic des maladies infectieuses
Les pathologies
visées par les diagnostics sur biopuces sont celles qui nécessitent une grande
rapidité
d'intervention et pour lesquelles les méthodes traditionnelles de diagnostic
sont
limités. En effet, le diagnostic moléculaire permet des tests plus rapides,
sensibles et
spécifiques. En évitant certaines étapes préliminaires telle que la culture, il
permet
d'obtenir un résultat en quelques heures là où plusieurs jours étaient
nécessaires.
Le champ d'application :
- les maladies sexuellement transmissibles telles que les infections à
Chlamydiae
et
N. Gonorrheae
;
- l'hépatite C : la société CIS Bio International a mis au point une
biopuce
capable de détecter la présence du virus de l'hépatite C et d'en préciser
le type
(I, II ou III).
- la tuberculose : chez Bio-Mérieux, le programme de recherche sur les
puces à ADN
est orienté vers une puce capable d'identifier en une seule étape toutes les
espèces de
mycobactéries connues (dont le bacille de la tuberculose), pathogènes ou non
pathogènes. Elle permettra d'identifier précisément l'espèce présente dans un
prélèvement pulmonaire et renseignera, en cas de
Mycobacterium
tuberculosis
, sur
la présence d'éventuelles mutations conférant une résistance au médicament
antituberculeux classique : la rifampicine. Cette puce Mycobactéries compte
aujourd'hui plus de 40 000 oligonucléotides.
- Dans le domaine, fort préoccupant lui aussi, des infections nosocomiales
la
société Bio-Mérieux Vitek étudie des biopuces regroupant des sondes spécifiques
des
ARN bactériens. Celles-ci pourraient permettre d'identifier les souches
bactériennes
dans les hôpitaux de déterminer rapidement les germes résistants aux
antibiotiques et
de prendre notamment des mesures prophylactiques.
La société Vysis (Napperville, Illinois) élabore un outil de diagnostic
miniaturisé
basé sur l'hybridation génomique comparative.
" Cette puce,
intitulée
Genosensor CGH d'environ 6 cm2 sera constituée de 625 sondes
représentatives
du génome humain et permettra la détermination de plusieurs centaines
d'aberrations
génétiques. "
56(
*
)
Il conviendra
ensuite de
préciser les conséquences de ces mutations.
- Les puces VIH différent selon les sociétés qui les ont mises au point.
La puce mise au point par Affymetrix, commercialisée début 1996 permet de
rechercher,
parmi une portion restreinte du millier de paires de bases du génome du VIH-1,
les
mutations conférant au virus une résistance vis-à-vis des inhibiteurs de la
transcriptase inverse et des antiprotéases. Elle donne un résultat en cinq
heures
seulement.
Une biopuce, objet de recherche chez Bio-Mérieux, rendra possible un test
quantitatif de
mesure de la charge virale, particulièrement utile aujourd'hui avec le
développement des
trithérapies.
- Le diagnostic du cancer peut également être réalisé par des biopuces,
notamment
par l'étude des gènes K-Ras et P 53.
CIS-Bio International et le Centre de lutte contre le cancer de Montpellier ont
conçu,
selon le procédé Micam, une biopuce capable d'identifier des mutations dans les
oncogènes
57(
*
)
, notamment dans le
gène K-Ras.
Affymetrix a, pour sa part, développé avec la firme Oncormed, spécialiste des
tests
génétiques de cancérologie, une puce capable de détecter les mutations du gène
P 53. Ce gène suppresseur de tueur est en effet muté dans près de la
moitié des
cancers. Commercialisée depuis juillet 1997 cette puce n'est pas seulement un
outil de
diagnostic, elle permet également de suivre l'impact, sur la tumeur, des
traitements de
radiothérapie ou de chimiothérapie.
1.2.5.2. La connaissance des prédispositions génétiques
Actuellement,
dans certains cas, les puces à ADN permettent de déceler les mutations
génétiques
indiquant qu'un individu a de fortes probabilités d'être atteint de telle ou
telle
maladie.
Plusieurs types de puces permettent, avec une fiabilité comparable aux
techniques
classiques de biologie moléculaire mais de façon plus rapide, plus simple et à
terme
moins coûteuse, de détecter des prédispositions génétiques. On peut
citer :
- les mutations du gène CFTR chez les patients qui risquent d'avoir une
mucoviscidose ;
- les mutations du gène de la bêta-globine chez les enfants menacés de
bêta-thalassémie (anémie héréditaire causée par un défaut dans la synthèse de
l'hémoglobuline) ;
- les mutations du gène BRCA 1 chez les femmes risquant d'être
atteintes d'un
cancer du sein. Dans ce dernier cas on peut même avoir recours, si le gène ne
présente
que de minuscules atteintes (microdélétions) à des puces spécialisées qui ne
sondent
que les positions connues pour porter ces mutations.
1.2.6. LES PROTÉINES THÉRAPEUTIQUES
1.2.6.1. Des protéines animales aux protéines recombinantes
Dans un
premier temps, un certain nombre de protéines naturelles étaient extraites de
fluides
biologiques ou organes animaux et humains dans un but thérapeutique. C'était le
cas du
facteur VIII, de l'urokinase, de l'insuline, de la calcitonine, de
l'hormone de
croissance...
Toutefois, cette production par extraction présentait des inconvénients :
- le risque de contamination lorsque les protéines venaient de sujets
malades non
détectés ;
- l'hétérogénéité des protéines d'extraction animales pouvant provoquer des
réactions immunitaires chez les patients ;
- la disponibilité limitée de matière première (l'hypophyse humaine par
exemple).
Puis la connaissance des gènes et les procédés biotechnologiques ont
ouvert, au
début des années quatre-vingt, une nouvelle voie pour la production des
protéines
thérapeutiques ; le procédé se déroule en quatre phases :
- détection et isolation du gène humain codant pour la protéine
recherchée ;
- intégration de ce gène dans une cellule-hôte (une bactérie ou une
levure) ;
- culture des cellules qui expriment la protéine ;
- extraction et purification de la protéine.
|
Les protéines ainsi produites couvrent un large champ thérapeutique : |
||
|
Produit |
Indication thérapeutique |
Première date d'autorisation |
|
- Abaximab |
Antiplaquettaire |
1994 |
|
- Aldeslenkine - IL 2 |
Carcinome du rein |
1992 |
|
- Altéplase t - PA |
Infarctus du myocarde aigu |
1995 |
|
- DNAse |
Mucoviscidose |
1993 |
|
- Erythropoïetine (EPO) |
Anémies |
1988 |
|
- Facteurs de
croissance
|
Cancer |
1991 |
|
- Facteur VIII |
Hémophilie A |
1992 |
|
- Filgrastim fG- CSF |
Neutropénie associée à la
chimiothérapie
|
1991 |
|
- Glucocérébrosidase |
Maladie de Gaucher |
1994 |
|
- Hormone de
croissance
|
Insuffisance en hormone de croissance chez l'enfant |
1985 |
|
- Insuline |
Diabète sucré insulino-dépendant |
1982 |
|
- Interférons |
Leucémie, Sarcome de Kaposi, Hépatites C et B, Mélanome malin, Condylomes acuminés |
1986 |
|
- Interférons |
Sclérose en plaques,
|
1986 |
|
- Muromonab CD3 |
Rejet de greffe aigu |
1993 |
|
- Sagramostim |
Transplantation de moelle, leucémie myéloïde |
1991 |
|
- Satumonab pendetide |
Détection et suivi du cancer colorectal et de l'ovaire |
1996 |
|
- Vaccin hépatite B |
Hépatite B |
1986 |
1.2.6.2. Les protéines issues d'animaux transgéniques
Un nouveau mode
de production de protéine émerge actuellement. Il repose sur l'insertion de
certains
gènes, codant pour la production de protéines thérapeutiques, dans des cellules
animales au stade embryonnaire.
Les animaux choisis sont producteurs de lait (chèvres, vaches, truies ou
lapines :
dans ce lait est produite la protéine).
Trois grands laboratoires mettent au point cette technique :
- chez Genzyme Transgenic des chèvres produisent de
l'antithrombine III. Cet
anticoagulant est en phase trois d'essais cliniques et sera utilisé à
l'occasion des
pontages cardio-pulmonaires.
Le recours aux chèvres convient pour des protéines à produire en grande quantité
telles que l'albumine humaine.
- Pharming a recours à des lapines pour produire des protéines dont les
quantités
nécessaires sont moins importantes : l'alpha-glucosidase, par exemple,
utilisée
dans le traitement de la maladie de Pompe (déficit enzymatique qui peut être
mortel).
- PPL Therapeutics fait usage de cinq types d'animaux (lapin, mouton,
souris, vache
et porc) ; cette société dispose d'un troupeau de 600 brebis
laitières. Elle
produit de la sérumalbumine humaine.
Une plus petite entreprise, Gala Design, au Canada, utilise une technique de
thérapie
génique pour introduire le gène désiré directement dans le pis d'une vache.
2. DEUXIÈME PARTIE : DE NOUVEAUX CHOIX À FAIRE
Toutes les
profondes modifications scientifiques et technologiques qui viennent d'être
exposées ont
pour conséquence l'absolue nécessité d'espérer de nouveaux choix dans les
domaines de
la recherche, de l'industrie et de la société.
Il s'agit d'une priorité : si la France ne s'adapte pas très rapidement à
ces
nouvelles technologies, elle sera totalement distancée dans la
" course
biologique " qui caractérisera le XXIe siècle.
Ce serait d'autant plus regrettable que, grâce aux travaux de grands chercheurs
tels que
Jean DAUSSET, Daniel COHEN ou Jean WEISENBACH, notre pays, au début des années
quatre-vingt-dix, ne manquait pas d'atouts. La situation s'est gravement
dégradée depuis
cette époque. Il est urgent de faire de nouveaux choix.
2.1. POUR LA RECHERCHE
2.1.1. LES STRUCTURES DE LA RECHERCHE
2.1.1.1. Les nouvelles stratégies de recherche dans l'industrie pharmaceutique et leurs conséquences
2.1.1.1.1. L'externalisation croissante de la recherche
Le
développement de nouveaux médicaments atteint des coûts de plus en plus
élevés :
3 milliards de francs et douze ans de mise au point pour une nouvelle
molécule
thérapeutique.
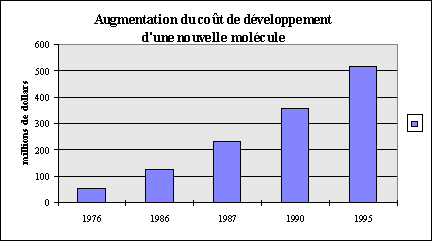
(58( * ))
Les grands
groupes pharmaceutiques, compte tenu du contexte de concurrence, doivent donc
améliorer
significativement la productivité de leur recherche-développement et déposer des
brevets pour de nouvelles molécules.
De nombreux analystes du secteur pharmaceutique estiment que la productivité de
la
recherche-développement des firmes pharmaceutiques n'est pas suffisante. Les dix
dernières années ont été marquées, dans toute l'industrie pharmaceutique par une
chute du nombre de lancements de nouveaux médicaments : 62 lancements
en 1987,
37 en 1996 et 47 en 1997
59(
*
)
. Les stratégies de
recherche-développement des grands groupes ne semblent pas permettre :
d'assurer un " retour " financier acceptable, à leurs
yeux, sur
les dépenses de recherche-développement ;
d'introduire un nombre suffisant de molécules sur le marché pour élargir
l'éventail des offres de soins et, parallèlement, pallier la diminution des
revenus des
produits anciens dont les brevets arrivent à expiration. Ce dernier problème va
devenir
crucial dans les années à venir, ainsi que le montre le tableau
suivant :
|
EXPIRATION DES BREVETS DES MÉDICAMENTS LES PLUS PRESCRITS DANS LE MONDE 60( * ) |
|||||||||||||||
|
Ventes mondiales en 1996 (en milliards de dollars) |
|
|
|
|
|
|
|
|
|
|
|
|
|
||
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
$12 |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
$10 |
|
$ 9,6 |
|
|
|
|
|
|
|
|
|
$ 10,3 |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
Cozaar |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
BuSpar |
|
|
|
|
|
|
|
|
|
Other |
|
|
|
|
$8 |
|
Sporanox |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
Cardura |
|
|
|
|
|
|
|
|
|
Zofran |
|
|
|
|
|
|
Ceftin |
|
$ 6,2 |
|
|
|
|
|
|
|
Zithromax |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
$6 |
|
Pepcid |
|
Transderm |
|
|
|
|
|
|
|
Rocephin |
|
|
|
|
|
|
|
|
Estraderm |
|
$ 4,7 |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
Accutane |
|
|
|
|
|
|
|
Pravachol |
|
|
|
|
|
|
Vasotec |
|
Prinivil |
|
Optiray |
|
|
|
|
|
|
|
|
|
|
$4 |
|
|
|
|
|
Accupril |
|
|
|
|
|
Biaxin |
|
|
|
|
|
|
|
|
Zestril |
|
Relafen |
|
|
|
$ 2,9 |
|
|
|
|
|
|
|
|
|
|
Mevacor |
|
Intron |
|
$ 2,2 |
|
|
|
Zoloft |
|
|
|
|
|
|
|
|
|
|
Axid |
|
|
|
Paraplatin |
|
|
|
$ 1,6 |
|
|
$2 |
|
|
|
|
|
Primaxin |
|
Lotensin |
|
Engerix |
|
|
|
|
|
|
|
|
Prilosec |
|
|
|
Nolvadex |
|
J&J |
|
|
|
|
|
Kytril |
|
|
|
|
|
|
Prozac |
|
|
|
|
|
Diflucan |
|
Zocor |
|
Lamisil |
|
|
|
|
|
|
|
|
Augmentin |
|
Cipro |
|
|
|
|
|
|
|
|
$0 |
|
|
|
|
|
|
|
|
|
Claritin |
|
|
|
Imitrex |
|
|
|
|
2000 |
|
2001 |
|
2002 |
|
2003 |
|
2004 |
|
2005 |
|
2006 |
|
|
ANNÉE D'EXPIRATION DU BREVET |
|||||||||||||||
Les groupes pharmaceutiques actuels ne peuvent mener à bien ces recherches. L'industrie pharmaceutique est faiblement concentrée : malgré les récents rapprochements, aucun groupe ne représente plus de 5 % du marché mondial et ne peut ni prendre le risque scientifique ni supporter la charge financière de la recherche-développement de très nombreuses molécules.
|
LES TRENTE PREMIERS GROUPES PHARMACEUTIQUES MONDIAUX 61( * ) |
||
|
Rang |
Groupes |
Parts du
|
|
1
|
Novartis
|
4,24 %
|
|
N.B. : Compte tenu des fusions intervenues en 1999, le groupe Rhône-Poulenc Rorer-Hoechst Marion Roussel (Aventis) aura une part de marché égale à 4,30 %, le groupe Astra-Zeneca 4,25 % et le groupe Sanofi-Synthélabo 1,48 %. |
||
Les groupes sont amenés à externaliser leur recherche : on estime à près de 30 % du budget de recherche-développement des dix plus grands groupes mondiaux la part externalisées en 2000, contre 4% en 1994.
2.1.1.1.2. Les nouveaux partenariats
Cette externalisation se concrétise par des accords entre les groupes et les entreprises de biotechnologie :
|
ACCORDS
PHARMA-BIOTECH DANS LA NOUVELLE APPROCHE DE MISE AU
POINT DE MÉDICAMENTS
|
||||||||
|
Sociétés |
Entreprises de biotechnologie par secteur de recherche |
|||||||
|
pharmaceutiques |
Génomique |
Chimie
|
Tests
|
Chimio-
|
Autres (thérapie
|
|||
|
Novartis
|
HGS
|
Pharmacopeia |
Evotec |
|
Idun Pharma
|
|||
|
Merck & Co
|
Merck Genome
|
Argonaut |
Cellomics |
|
Axys
|
|||
|
Glaxo Wellcome
|
Incyte
|
Affymax
|
|
MDL Info.Syst.
|
Ligand
|
|||
|
Pfizer
|
Incyte
|
Oxford
|
|
Tripos |
Ligand |
|||
|
Bristol Myers Squibb
|
Millennium
|
Trega
|
Aurora |
Tripos |
Cadus
|
|||
|
Johnson & Johnson
|
Incyte
|
ArQule |
|
|
Allelix
|
|||
|
American Home Products
(Wyeth Ayerst)
|
Millennium
|
ArQule |
SIGA
|
3-Dimensional
|
Ligand |
|||
|
Roche |
Incyte
|
Alanex
|
Clontech
|
Oxford molecular |
Signal |
|||
|
Eli Lilly |
Incyte
|
Sphinx |
Aurora
|
|
Allelix
|
|||
|
SmithKline Beecham
|
HGS
|
|
Evotec |
Orchid Biocomputer |
Axys
|
|||
|
Source : " Biotechnologies et Santé " . Publication Eurasanté. Lille 1999. |
||||||||
|
GENCELL
(RHÔNE-POULENC RORER) : COLLABORATIONS AVEC
|
||||||||
|
|
|
Domaine de recherche |
||||||
|
France |
Génopoïetic |
Gène suicide, puis plasmavirus |
||||||
|
États-Unis |
Applied Immune
Science*
|
Thérapie
cellulaire
|
||||||
|
* Acquis par R-PR ( 62( * ) ) |
||||||||
|
NOVARTIS : PRINCIPALES COLLABORATIONS DE RECHERCHE
|
||||||||
|
|
|
Domaine de recherche |
||||||
|
Europe |
Evotec |
Nano-thérapie |
||||||
|
Japon |
Yoshitomi
Pharmaceuticals
|
Immunosuppression |
||||||
|
États-Unis |
Alexion
|
Thérapie
génique
|
||||||
|
* Filiale à 53 % de Novartis (1) |
||||||||
En 1997,
240 contrats représentant 4,35 milliards de dollars ont été signés
entre les
entreprises pharmaceutiques et les petites et moyennes entreprises de
biotechnologies
mondiales contre 180, pour 2,84 milliards de dollars, en 1996
63(
*
)
Ce type de contrat devient progressivement familier aux sociétés de
biotechnologies
européennes. À titre d'exemple, voici quelques-uns des principaux contrats
obtenus par
des firmes européennes ces derniers mois :
|
|
Genset et Pharmacia&Upjohn (pharmacogénomique) ; |
|
|
Cerep et Exonhit (3 programmes dont un concernant le développement d'un outil de prédiction de toxicité et un autre sur l'identification de cibles dans le domaine des maladies neurodégénératives) ; |
|
|
Gencell et Endocyte (accord prévoyant des droits exclusifs sur la propriété intellectuelle détenue par Endocyte concernant l'utilisation de récepteurs de l'acide folique pour l'administration de produits de thérapie génique en oncologie) ; |
|
|
Gencell et Oxford Biomedica (accords de collaboration et de licence sur 2 ans et portant sur un minimum de 1 million de dollars) ; |
|
|
Gencell et LXR Biotechnology lnc (accord de .recherche sur le rôle cytoprotecteur du gène Sarp-1) ; |
|
|
Exonhit Therapeutics et Sosei Co Ltd (accord de partenariat franco-japonais) ; |
|
|
Biovector Therapeutics et Bayer (accord de R&D visant à mettre au point un vaccin ADN contre le SIDA) ; |
|
|
Biovector therapeutics et Chiron (accords de développement thérapeutique contre l'hémophilie). 64( * ) |
Cette
externalisation donne également naissance à des partenariats entre les groupes
pharmaceutiques et les centres de la recherche publique.
À titre d'exemple, on peut citer les accords conclus par Rhône-Poulenc Rorer.
En 1998,
près de 20 % du budget de recherche a été externalisé grâce à des
partenariats
dont un quart représentent des collaborations avec des organismes publics et
semi-publics
français (CNRS, INSERM, universités, Institut Pasteur...). Le médicament
anticancéreux
Taxotere est le résultat d'une collaboration entre Rhône-Poulenc Rorer et le
CNRS. Trois
unités mixtes de recherche (UMR) regroupent des chercheurs du CNRS et de
Rhône-Poulenc
Rorer.
Dorénavant, la valorisation des recherches effectuées dans le secteur public
passera par
des accords avec les entreprises privées, associant les chercheurs publics et
privés.
Cette tendance existe déjà. En France, le montant des contrats entre les
laboratoires de
la recherche publique et des commanditaires privés ou publics était de
500 millions
de francs en 1983. Il s'est élevé à 3,4 milliards de francs en 1995. Le
nombre de
contrats en cours entre le CNRS et des entreprises était de 350 en 1982 et de
3200 en
1996.
65(
*
)
Il est indispensable que ces partenariats public-privé se renforcent en se
structurant
dans deux directions :
- la collaboration des chercheurs publics avec les grands groupes
pharmaceutiques ;
- la collaboration des chercheurs publics avec les entreprises de
biotechnologie, qui
ont elles-mêmes noué des liens avec les groupes.
Cette orientation trouve son illustration dans les bilans de l'Institut Pasteur
pour 1997
et 1998.
"
En 1997, des partenaires nouveaux se sont manifestés et des
contacts se
sont développés notamment dans le secteur des biotechnologies. Il apparaît en
effet de
plus en plus utile de rechercher l'appui de petites sociétés innovantes,
proches du
milieu de la recherche, qui constituent des intermédiaires privilégiés entre les
chercheurs et la grande industrie. "
66(
*
)
" La connaissance du génome des micro-organismes pathogènes offre en
effet de
vastes possibilités de recherche de nouveaux agents thérapeutiques. Dans ce
domaine,
l'Institut Pasteur a noué des collaborations aussi bien avec des sociétés de
dimension
internationale, comme Hoechst Marion Roussel, qu'avec de petites sociétés
émergentes du
secteur des biotechnologies.
Au total, ces contrats de Recherche et Développement ont apporté un concours
industriel
de près de 17 millions de francs, finançant notamment 38 personnes
dont
27 jeunes chercheurs. Ce résultat est apprécié comme un succès de la
politique
d'intensification et de diversification des liens avec
l'industrie. "
67(
*
)
L'INSERM (Institut national de la santé et de la recherche médicale) manifeste
un souci
analogue :
" L'INSERM soutient des programmes transversaux parmi lesquels la
physiopathologie, soit tout ce qui concoure à la compréhension des maladies et
la
thérapeutique, avec la découverte de molécules nouvelles par le biais des
biotechnologies ou de la chimie du médicament. Dans ce domaine, il est
nécessaire de
bâtir des plates-formes où recherche académique et recherche industrielle se
trouvent
mêlées. À ce titre, l'INSERM prévoit de dynamiser sa politique de brevets et de
favoriser la création d'entreprises. "
68(
*
)
2.1.1.2. La nécessaire valorisation de la recherche publique
" Notre pays dispose d'un potentiel scientifique et technologique de premier plan mais le couplage de ces découvertes et de ces connaissances avec les activités industrielles s'effectue moins facilement qu'aux États-Unis et au Japon " 69( * ) .
2.1.1.2.1. Le " transfert des chercheurs "...
Une interdiction levée :
En 1998, de
nombreux rapports officiels, dont celui d'Henri Guillaume et celui de la Cour
des Comptes
ont mis en lumière un certain nombre d'incohérences et de conflits d'intérêt
faisant
obstacle à la création d'entreprises par les chercheurs des organismes publics.
Ceux des
EPST (établissements publics à caractère scientifique et technique) n'avaient
pas le
droit de prendre une participation dans une société privée (loi du
13 juillet
1983). La Cour des Comptes notait en particulier
" les risques de
dérive
auxquelles pouvaient conduire les aménagements apportés à l'interdiction, pour
un
fonctionnaire de recherche, de participer au capital d'une entreprise liée par
contrat
avec son établissement "
: les chercheurs devaient se mettre
" hors la loi ", avec ou sans l'accord de leur
administration, pour
valoriser leur savoir dans le monde socio-économique.
" Le statut du chercheur ne lui permet pas de créer une
entreprise. Du coup,
la valorisation industrielle de la recherche s'opère dans la clandestinité à
travers un
système hypocrite favorisant l'assentiment tacite illégal de la hiérarchie au
détriment d'une réelle stratégie "
70(
*
)
.
C'est pourquoi le projet de loi présenté par le Ministre de l'Éducation
nationale, de
la Recherche et de la Technologie a eu notamment pour objectif de mettre fin à
cette
situation très nuisible au transfert de l'innovation à partir de la puissance
publique.
Il permettra aux chercheurs et aux enseignants chercheurs de créer des
entreprises :
les chercheurs pourront s'engager dans la création d'une entreprise. Ils seront
autorisés à participer en tant qu'associé, administrateur ou dirigeant à cette
entreprise nouvelle, pendant une période à l'issue de laquelle ils pourront
opter entre
le retour dans le service public ou l'appartenance à l'entreprise. Durant cette
période,
et pour une durée maximale de six ans, ils seront détachés ou mis à
disposition. Ils
conserveront par conséquent leur statut de fonctionnaire. Cette entreprise
pourra
entretenir des liens contractuels avec le laboratoire d'origine du chercheur,
ce qui
facilitera le transfert de technologie. Le chercheur ne sera pas contraint à
une rupture
brutale avec son laboratoire d'origine.
Il est donc indispensable, cette loi ayant été finalement votée, le
30 juin 1999,
à minuit, que toutes les mesures d'application soient prises très rapidement
afin de
mettre fin à la situation difficile de certains chercheurs et de promouvoir la
valorisation de la recherche publique
Des incitations à créer
Chaque année,
tandis que les chercheurs américains créent 400 à 500 entreprises, leurs
homologues français n'en lancent que le dixième. Afin d'améliorer cette
situation, il
ne suffit pas de lever les obstacles juridiques. Il convient également de créer
un cadre
fiscal incitatif.
Dans le cadre des mesures financières, on pourrait envisager de créer un prix
annuel,
dont le montant ne serait pas symbolique, destiné à récompenser l'équipe du
laboratoire qui a travaillé avec un chercheur dès lors que celui-ci crée une
entreprise
innovante dans la spécialité du laboratoire.
Enfin, s'il est important d'inciter les chercheurs publics à se lancer dans la
création
d'entreprises, il est également essentiel d'encourager tous ceux qui n'ont pas
l'esprit
d'un chef d'entreprise, ce qui se conçoit parfaitement, à exercer des fonctions
de
consultant auprès des entreprises innovantes. Ces consultations doivent se
situer dans un
contexte de transparence et sans interférence avec les décisions publiques.
Si même cette solution suscite peu d'enthousiasme, il faut au minimum faire,
dans tous
les organismes de recherche, comme au Commissariat à l'Énergie atomique :
inciter
les chercheurs qui ne veulent pas s'engager dans l'aventure industrielle à
réfléchir
néanmoins aux débouchés éventuels de leurs résultats en terme de produit ou de
service, de façon à pouvoir en confier le développement à un créateur
d'entreprise.
On passe ainsi du " transfert des chercheurs " au transfert
des
recherches.
2.1.1.2.2. ... ou le " transfert de la recherche "
Il repose sur une amélioration des structures de valorisation et de transfert technologique.
Se garder de saupoudrer le transfert technologique
Dans la période
d'après-guerre, le développement technologique français reposait sur le
transfert
d'innovations scientifiques vers les grosses structures industrielles,
essentiellement
sous forme de cessions de licences d'exploitations de brevets dans le cadre de
grands
programmes d'État (aéronautique, défense, nucléaire...)
Aujourd'hui la valorisation s'est diversifiée : elle recouvre des activités
multiples (prise de brevet, création d'entreprises innovantes, contrats de
recherche pour
l'industrie, activités de consultant) et, de plus, elle se tourne de plus en
plus vers
les petits et moyennes entreprises. Ceci est particulièrement vrai dans le
domaine des
biotechnologies où la souplesse et la capacité d'adaptation d'une petite
entreprise est
un atout considérable compte tenu de l'évolution très rapide des techniques.
Les outils de valorisation se sont également diversifiés pour atteindre tous les
éléments du nouveau tissu industriel.
Toutes sortes de structures assurent l'interface entre le monde de la recherche
et celui
de l'industrie. Leurs tâches sont multiples : prospecter les
laboratoires ;
inciter les entreprises à innover et à embaucher des scientifiques ;
offrir des
services particuliers (détachement de spécialistes, contrats de sous-traitance,
mise en
place d'équipes mixtes de recherche) et des aides financières.
Cette multiplication des outils de valorisation a un effet négatif. Plusieurs
centaines
de structures de transfert technologique ont des compétences et des missions
qui se
recoupent souvent.
" Elles bénéficient d'un financement plus ou moins important de
l'État
-notamment via l'ANVAR, qui gère les aides à l'innovation depuis 1979- et des
collectivités territoriales. Certains, comme les 52 Centres relais
innovation (CRI)
créés par appel d'offre en 1995, reçoivent même des subsides européens. Ces
structures peuvent être, à des degrés divers, adossées à des organismes de
recherche
ou des universités, et avoir des activités plus ou moins spécialisées.
Certaines sont
fédérées en réseaux, mais cela ne suffit pas à assurer leur homogénéité.
C'est par exemple le cas des centres régionaux d'innovation et de transfert de
technologies (CRITT), créés en 1982 sous l'impulsion du ministère de l'Éducation
nationale, de la Recherche et de la Technologie (MENRT) pour structurer le
potentiel de
recherche des régions sont un exemple intéressants. Il en existe aujourd'hui
environ
120, de statuts (associations, groupements d'intérêt public [GIP]...) et
missions très
hétérogènes. La moitié d'entre eux sont spécialisés dans des domaines
technologiques
et possèdent parfois des équipements de recherche et développement
importants ;
l'autre moitié se résume à de simples " points d'appui
technologiques ". Plus généralistes, ces centres ont un rôle de
sensibilisation
et d'orientation des PME/PMI. Les pouvoirs publics ont également créé, en 1990,
un
réseau de diffusion technologique (RDT), animé par l'ANVAR et auquel participe
le CEA
(Commissariat à l'énergie atomique), qui est aujourd'hui implanté dans
21 régions. Il permet de coordonner différentes structures régionales
publiques et
parapubliques de transfert. Parallèlement, de nouveaux réseaux se constituent à
l'initiative des collectivités locales ou d'acteurs privés : réseau de
développement industriel (RDI), réseaux de développeurs, etc.
Par ailleurs, en 1996, l'ANVAR a agréé 48 sociétés de recherche sous
contrat
(SRC) et assimilées, elles aussi d'initiative privée, qui font de la R&D
orientée
à la demande de l'industrie et ont donc un rôle de relais entre la recherche et
les
entreprises. Quant aux technopôles, comme Sophia-Antipolis ou Rennes-Atalante,
ce sont
des acteurs régionaux importants de la valorisation. Enfin, toutes ces
interfaces
s'ajoutent à d'autres plus traditionnelles, comme les 18 centres techniques
industriels (CTI), créés en 1948 par arrêté ministériel sur proposition des
organisations professionnelles, et répondant en particulier aux besoins de
formation
professionnelle, de prestations d'essais, de mesure et de contrôle, et de
protection de
l'environnement d'un certain nombre de branches professionnelles du secteur
industriel et
manufacturier "
71(
*
)
.
Henri GUILLAUME, dans son rapport sur la technologie et l'innovation, avait mis
en
lumière ce foisonnement et conclu que la multiplicité des structures de
transfert et de
diffusion technologiques nuisait à leur efficacité globale, les entreprises
identifiant
mal l'interlocuteur utile.
C'est pourquoi il est indispensable de rationaliser rapidement les structures de
valorisation de la recherche.
Simplifier les relations contractuelles entre les organismes de recherche et les entreprises
Les structures
de valorisation précédemment décrites s'adressent essentiellement aux
industriels et
existent indépendamment des organismes publics de recherche.
Il existe des structures particulières de coopération entre les établissements
publics
de recherche et d'enseignement supérieurs et les entreprises. Elles résultent
essentiellement de la loi du 15 juillet 1982 qui avait prévu plusieurs
modalités de
coopération telles que les groupements d'intérêt public les unités mixtes de
recherche, la création de filiales et les prises de participation.
Malheureusement, la rigidité de ces structures explique leur faible succès.
En particulier, les groupements d'intérêt public (GIP), conçus pour établir des
coopérations durables entre des partenaires des secteurs publics et privés sont
lourds
à mettre en oeuvre. Leur création nécessite par exemple un arrêté
interministériel
d'approbation requérant la signature de plusieurs ministres : les délais
qu'impliquent une telle procédure sont incompatibles avec les impératifs de
rapidité
économique. De fait, seuls une dizaine de GIP ont été créés.
Il faudrait multiplier ces types de coopération pour assurer de véritables
échanges
entre les chercheurs publics et privés. Là encore, la loi sur l'innovation
allège les
formalités administratives pour ces structures de collaboration (notamment en
remplaçant
l'arrêté interministériel par un régime d'autorisation tacite) ; elle
facilite
également la gestion des contrats à durée déterminée entre les établissements
d'enseignement supérieur et de recherche et les entreprises, novation
importante, compte
tenu du succès croissant de ces collaborations ponctuelles et souples (le CNRS
a passé
350 contrats de recherche avec les entreprises en 1982 et 3200 en
1996...).
Favoriser les réseaux
Il est
indispensable de faciliter les relations entre la communauté scientifique
académique et
le milieu industriel. Dans le domaine de la biotechnologie, la
recherche-développement
constitue une part importante de l'activité des entreprises. Les échanges avec
les
laboratoires de recherche fondamentale sont fructueux pour les deux parties.
Les grands groupes créent des consortiums réunissant universités, laboratoires
publics
et entreprises industrielles. Leur but est de poursuivre une recherche
scientifique
commune ou de mener des actions de recherche-développement servant de base à la
découverte de produits nouveaux.
Ainsi, le groupe Pfizer a constitué un réseau, baptisé Pfizergen, de douze
sociétés
spécialisées pour la plupart dans les recherches sur le génome.
Dans le domaine de la thérapie génique, par l'intermédiaire de Gencell, le
groupe
Rhône-Poulenc Rorer a construit un réseau composé de plus de 18 partenaires
publics et
privés internationaux.
Quant au groupe Hoechst Marion Roussel, il a créé en 1997, un GIP pour
favoriser une
collaboration entre chercheurs du public et du privé sous l'égide du Ministère
de
l'éducation nationale, de la recherche et de la technologie. Ce GIP finance,
pour un
montant de 220 millions de francs sur trois ans, des laboratoires
d'organismes
publics (CNRS, INSERM, CEA, Institut Pasteur et Universités). L'objet de ces
recherches
est la connaissance des fonctions des gènes et la mise au point de biopuces. À
l'heure
actuelle 62 programmes scientifiques ont été sélectionnés.
Le Ministère de l'Éducation nationale de la Recherche et de la Technologie met
en place
un réseau des divers partenaires intéressés par le génome humain. Ce réseau,
similaire à celui de Génoplante, a pour objectif de permettre à la France de
structurer
sa contribution au décryptage du génome humain et d'aller au-delà du séquençage
du
chromosome 14.
2.1.2. LES ORIENTATIONS DE LA RECHERCHE
Le 1 er juin 1999, les priorités de la recherche française ont été fixées par le deuxième Comité interministériel de la recherche scientifique et technologique (CIRST). Elles vont dans un sens favorable aux sciences du vivant : le relevé des conclusions du CIRST, précise que " Dès 1999, quatre domaines d'actions feront l'objet d'un développement immédiat : la génomique et la " post-génomique ", les technologies appliquées à la médecine, les sciences du cerveau et de la cognition, la lutte contre les maladies infectieuses " .
|
RÉCAPITULATIF DES ACTIONS PRIORITAIRES LANCÉES EN 1999
|
|||
|
|
Les actions concertées incitatives (ACI) : elles permettent l'élaboration de programmes de recherche destinés notamment à favoriser l'émergence de disciplines nouvelles et la formation de spécialistes dans ces domaines, à encourager des partenariats public/privé et à assurer un soutien à certaines politiques publiques. |
||
|
|
Les réseaux de recherches technologiques (RT) : ils associent des acteurs de la recherche publique et des industriels, à partir de l'identification des besoins, sur des projets porteurs de croissance et de création d'emploi. |
||
|
|
Sciences du vivant (395
MF) :
|
80 MF
|
|
|
|
Sciences de
l'information et de la communication (175 MF) :
|
80 MF
|
|
|
|
Sciences humaines et
sociales (85 MF) :
|
20 MF
|
|
|
|
Sciences de la planète
et de l'environnement (45 MF) :
|
10 MF
|
|
|
|
Énergie (20
MF) :
|
20 MF |
|
|
|
Génie civil,
architecture, urbanisme et transports (75 MF) :
|
65 MF
|
|
|
|
Aéronautique (10 MF)
|
|
|
|
Source : AFP Sciences n° 1189. 3 juin 1999. |
|||
De plus, le
Ministre de l'Éducation nationale, de la Recherche et de la Technologie a
confirmé, en
septembre 1999, que la biologie constituait une priorité de la recherche
française et
que : " la génomique [...] recevra plus de 2 milliards de
francs sur
trois ans "
72(
*
)
.
Mais les organismes publics de recherche doivent participer encore plus qu'ils
ne le font
au développement des sciences de la vie. L'enveloppe consacrée à celles-ci par
le CNRS
est de 300 millions de francs, dont 15 millions de francs pour le
programme
Génome lors de sa création, puis 20 millions de francs pour l'année
suivante. Pour
1999, au CNRS, le budget des sciences de la vie est, certes, en progrès de
10 % ; mais il faudrait que cette tendance s'amplifie. Le Professeur
Pierre
CHAMBON conseille de répartir autrement ce budget pour prendre en compte
l'évolution de
la science et le développement considérable de certains secteurs, tout
particulièrement
celui de la biologie. Il rappelle qu'aux États-Unis, en dix ans, le budget de
la biologie
et de la biomédecine a plus que doublé en dollars constants
73(
*
)
.
2.1.2.1. Affiner la connaissance de l'ADN et de ses " produits ", les protéines
2.1.2.1.1. Les " non-gènes " du génome
L'ADN est composé d'environ trois milliards de paires de bases (A , G, C, T) mais moins de 10 % seulement de ces bases constituent les séquences nommées gènes, que les cellules utilisent pour synthétiser des protéines.
La régulation génétique
Certaines des
séquences non porteuses de gènes gouvernent des fonctions essentielles, telles
l'activation ou l'inhibition de gènes. En effet, toutes les protéines codées
par les
gènes ne sont pas synthétisées uniformément et en quantité similaire quels que
soient
l'instant ou le lieu de l'organisme considéré. Certaines protéines ne sont
synthétisées que dans certains types de cellules comme les cellules musculaires
ou les
cellules nerveuses. D'autres ne sont produites qu'à certains moment du cycle
cellulaire.
Ceci s'explique par la présence, sur la chaîne d'ADN de sites qui sont soit
promoteurs
soit répresseurs et qui régulent l'activité du gène c'est-à-dire la production
de
protéines.
Il est essentiel que les chercheurs se penchent sur l'étude de ces sites car il
pourrait
être plus aisé d'agir sur eux plutôt que sur les gènes pour moduler
l'expression des
protéines et donc soigner les maladies.
L'intérêt de cette approche est tel que des firmes s'y intéressent déjà. La
société
Genset a déjà procédé au clonage de séquences de régulation. La société Genelabs
en Californie travaille sur le développement de petites molécules
pharmaceutiques
pouvant agir au niveau de ces séquences régulatrices en activant ou désactivant
la
séquence codante : ces molécules se lieraient directement à des séquences
spécifiques de l'ADN en s'y incrustant. Elle a déjà sélectionné environ
700 séquences spécifiques d'ADN qui peuvent donner lieu à la découverte de
nouvelles molécules pharmaceutiques.
Les microsatellites
Certaines régions de l'ADN non-codant sont composées de microsatellites, courtes séquences répétitives d'une combinaison particulière des quatre bases de l'ADN (par exemple : ATTCAGATTCAGATTCAG). Les généticiens leur ont récemment découvert un rôle important et de nombreuses fonctions : leur nature répétitive les rend particulièrement sujets à des augmentations ou des réductions de longueur qui ont parfois de lourdes conséquences pour l'organisme. Chez certaines bactéries pathogènes, une variation du nombre de répétitions favorise l'apparition de nouvelles propriétés et la survie en cas de modifications de l'environnement. Chez l'être humain aussi, certains microsatellites ont vraisemblablement des effets importants ; leur nombre peut atteindre 100 000 dans le génome. Actuellement les seules fonctions connues des microsatellites humains sont négatives (ils sont notamment à l'origine de maladies neurologiques). En attendant de trouver des moyens d'action sur ces microsatellites, les chercheurs devraient étudier attentivement les possibilités de les utiliser dans le domaine du diagnostic précoce.
DÉPISTER LE CANCER
" Les
microsatellites amélioreront bientôt le dépistage précoce du cancer. On détecte
aujourd'hui jusqu'à une seule cellule cancéreuse parmi 10 000 cellules
normales en recherchant des mutations de gènes, tels les gènes p53 et ras
(certaines
formes de ces gènes prédisposent au cancer). Toutefois, ces mutations ne se
produisent
pas dans tous les cancers, ni même dans tous les cancers d'un même type.
Les microsatellites fournissent une autre méthode de dépistage précoce du
cancer, car
la fréquence d'augmentation ou de réduction des microsatellites est bien plus
importante
dans certaines cellules cancéreuses que dans les cellules normales. De telles
modifications, qui portent souvent sur de nombreux microsatellites différents,
sont
facilement délectables. Cette méthode permet aujourd'hui de déceler une cellule
cancéreuse parmi environ 500 cellules normales.
Les modifications des microsatellites dans les cellules cancéreuses furent
découvertes
en 1993 par Manuel PERUCHO, de l'Institut de recherches biologiques de La
Jolla, en
Californie. Étudiant la forme héréditaire d'un cancer du côlon, il observa que
la
longueur des microsatellites de cellules cancéreuses était différente de celles
des
cellules normales du même patient. On a ensuite montré qu'une des anomalies qui
provoquait ces modifications se situe dans un gène codant une enzyme
responsable de la
correction de la longueur des microsatellites. La perte du gène fonctionnel
augmenterait
vraisemblablement la probabilité de rencontrer des erreurs non corrigées.
Enfin, Richard BOLAND, de l'Université de San Diego, et d'autres généticiens ont
inséré un chromosome humain porteur d'un gène normal de réparation de l'ADN
dans des
cellules cancéreuses cultivées en laboratoire. Ils ont observé que le gène
introduit
ralentissait la fréquence de mutation des microsatellites des cellules
cancéreuses.
Toutefois, les souris génétiquement modifiées nommées
"knock-out "
ne possèdent pas le gène codant l'une des principales protéines de réparation;
elles
ne vivent que peu de temps et présentent de nombreux types de cancers. Pourtant
les
mutations de leurs microsatellites ne sont plus fréquentes dans aucune de leurs
cellules
cancéreuses. L'instabilité des microsatellites semble être plus un symptôme que
la
cause du cancer. Ces changements ne formeraient qu'une partie des nombreuses
modifications
génétiques qui se produisent en cascade dans tout le génome d'une cellule, lors
du
processus de cancérisation.
Néanmoins, cette association est suffisamment fréquente pour que l'instabilité
des
microsatellites fournisse aux médecins un nouvel outil de dépistage. Des essais
cliniques concluants de dépistage précoce, à l'aide de microsatellites, ont été
d'abord effectués pour des cancers colorectaux et pour des cancers de la
vessie. Ils sont
aujourd'hui utilisés pour de nombreux autres types de cancers. Cependant, ces
tests ne
sont encore utilisés que dans des recherches. On espère qu'ils serviront aussi à
déterminer le type de cancer dont souffrent les malades. "
(74( * ))
2.1.2.1.2. La protéomique
L'intérêt
principal de l'acquisition de nouvelles connaissances sur les génomes est de
permettre de
mieux cerner la formation des protéines, leur fonction et leur structure. Cette
étude
des protéines, la protéomique, doit être réalisée au fur et à mesure des
progrès de
la génomique.
" La recherche académique doit prendre le relais de l'industrie.
Cette
décennie a connu des progrès scientifiques inimaginables, mais il y a sûrement
trop
d'entreprises qui s'occupent de cartographier le génome humain et pas assez qui
s'intéressent au fonctionnement de la cellule. "
75(
*
)
Mieux comprendre l'épissage
Épissage, un
terme peut-être inapproprié, est la traduction du mot anglais
" splicing "
. Il désigne un processus génétique un peu
complexe. Au niveau du gène, l'information génétique est fragmentée en segments
d'ADN
appelés exons, que l'on retrouve au niveau de l'ARN messager, séparés par des
segments
d'ADN sans correspondance dans l'ARN messager, les introns. Au début de la
transcription
de l'ADN, l'ARN prémessager subit une série de modifications qui conduisent à
la mise
bout à bout des exons et à l'élimination des introns. Ce processus est appelé
épissage ; il n'est pas figé et n'aboutit pas toujours au même résultat.
Suivant
la façon dont les exons se réorganisent (certains sont parfois éliminés), les
ARN
messagers sont différents, alors qu'ils sont issus de la même séquence d'ADN.
Un gène
peut coder une protéine différente de celle à laquelle l'information génétique
de
base devrait donner naissance. Des dysfonctionnements de l'épissage peuvent
avoir des
conséquences pathologiques.
Cette connaissance qualitative et non plus quantitative des gènes doit être
approfondie,
car elle présente de véritables opportunités thérapeutiques. En France, la
société
privée ExonHit Therapeutics travaille sur ce sujet en collaboration avec
l'Institut de
Génétique, Biologie moléculaire et cellulaire (Strasbourg), l'Hôpital Paul
Strauss
(Strasbourg) l'hôpital Cochin-Port-Royal (Paris) et le Centre de Pathologies
infectieuses
et immunologiques (Tours).
Connaître les fonctions, les interactions et les structures des protéines
La connaissance
des séquences génétiques permettra, à terme, d'établir un classement
fonctionnel des
protéines (les protéines régulatrices, celles qui sont douées de propriétés
enzymatiques, celles qui transportent les ions, etc.).
Toutefois, cette approche classique a ses limites. Les chercheurs regroupent
les gènes et
les protéines correspondantes en classes fonctionnelles en se fondant sur les
similitudes
observées dans les séquences de leur ADN. Cependant l'on constate aujourd'hui
que des
gènes de séquences apparemment différentes peuvent coder pour des protéines
ayant des
structures tridimensionnelles et des fonctions très similaires. De plus
l'approche
génomique, dans la majorité des cas, permet d'établir la structure primaire,
" linéaire ", des protéines mais pas de prédire, à partir
d'une
seule séquence, la structure bidimensionnelle d'une protéine. Or, la
connaissance de
cette structure dans l'espace est indispensable pour comprendre la fonction de
la
protéine au niveau atomique et étudier, par exemple les serrures physiologiques
où
pourrait entrer les clés que sont les molécules thérapeutiques.
Ce constat amène à émettre quelques recommandations en matière de
recherche :
- Il conviendrait de tenir compte de la nouvelle stratégie élaborée par les
chercheurs américains ; ils ont constaté la quasi-impossibilité d'établir
les
structures tridimensionnelles des protéines correspondant à l'ensemble des
gènes dont
on a déjà déchiffré les séquences. Ils ont alors décidé
76(
*
)
de déterminer les structures d'un échantillonnage des protéines qui soient
représentatives de tous les types de structures trouvées dans la nature, de
manière à
disposer de références générales pour l'ensemble des protéines existants. La
connaissance de ces structures " chefs de file ",
améliorerait les
programmes informatiques permettant de prédire la structure et la fonction des
autres
protéines à partir des séquences de leurs gènes.
- Il faut investir dans des programmes de bio-informatique permettant des
comparaisons fines entre les structures des diverses protéines, sachant que ces
comparaisons peuvent donner une idée des fonctions de ces protéines.
- Il faut également, au niveau de la recherche fondamentale étudier la
voie de la
cristallographie à haut débit. Les biopuces permettent d'assigner une fonction
biochimique aux protéines ; celles-ci peuvent être synthétisées, purifiées
puis
cristallisées. La cristallographie à haut débit permet ensuite d'obtenir les
structures
des protéines en trois dimensions.
- Il convient de s'intéresser aux interactions des protéines pour
connaître les
fonctions de ces dernières. On peut citer en ce domaine la stratégie développée
par la
société française Hybrigenics, créée en 1997 grâce à une participation
financière
de l'Institut Pasteur. Elle vise à établir le réseau des interactions entre les
protéines grâce à des techniques d'hybridation et de criblage : à partir
d'une
protéine " appât ", elle crible rapidement l'ensemble des
protéines
" proies ". Elle se base pour cela sur des méthodes
laboratoires,
dites du " double-hybride " et du
" triple-hybride ".
- Il est indispensable de développer la recherche en biologie structurale.
Notre
pays souffre en ce domaine d'un retard grave. Or, la génomique et la
bio-informatique ne
donneront les résultats thérapeutiques attendus qu'en restant liées en
permanence aux
données de la biologie structurale, notamment à la conformation spatiale des
protéines.
2.1.2.2. Les biopuces en France
Il faut
confirmer l'orientation de la recherche française vers les biopuces, compte
tenu de leurs
multiples applications, de la nécessité d'améliorer leurs performances et de
l'attitude
du principal producteur, Affymetrix. Cette firme ne fabrique pas des puces à la
demande
mais collabore à des projets scientifiques seulement lorsqu'ils lui paraissent
intéressants. À cette politique de restriction des thèmes de recherche,
Affymetrix
ajoute une protection jalouse de certains brevets, notamment celui qui protège
sa
capacité à mettre plus de 400 séquences d'ADN par centimètre carré de
surface.
Pour contourner ce brevet, certaines sociétés développent des biopuces
utilisant des
surfaces constituées de microcuvettes. Pour Ron LONG, un dirigeant du groupe
anglo-suédois Amersham Pharmacia, évoquant ce domaine précis,
" la
protection de la propriété intellectuelle va être un véritable champ de
mines "
.
Outre la nécessité de s'affranchir de la tutelles d'Affymetrix, celle de
perfectionner
les biopuces doit guider les chercheurs et les firmes française. Ces
perfectionnements
concernent plusieurs domaines.
De la synthèse ex situ à la synthèse in situ
-
L'amélioration de la synthèse
ex situ
des sondes d'ADN destinées à être
fixées sur les biopuces est l'objet de recherches menées, dans le cadre du
programme
génome du CNRS par une équipe strasbourgeoise et par un groupe réunissant des
chercheurs du laboratoire de biologie de l'École supérieure de Physique et de
Chimie
industrielles de la ville de Paris, de l'École normale supérieure et de
l'Institut
Curie.
- La synthèse
in situ
des sondes, qui représente la solution d'avenir est
actuellement étudiée dans le cadre du projet ROSA intégré au programme Génome
du CNRS
et piloté par l'École centrale de Lyon. D'après Francis GALIBERT, responsable du
programme Génome,
" le but de ce projet est de développer une
méthode de
fabrication de puces avec des milliers d'oligonucléotides, conçues selon la
demande, en
utilisant la chimie classique, qui permet de synthétiser des oligonucléotides
de grande
longueur. Le
problème est alors essentiellement un problème de fluidique,
c'est-à-dire d'adressage de réactifs et de chimie de surface "
.
Cette approche est également celle de la petite firme américaine
Protogene : tout
en menant leurs recherches de façon autonome, les équipes françaises doivent
impérativement maintenir les contrats de collaboration avec cette société.
Après avoir, il y a quelques années, développé avec la société CIS Bio
International
un procédé original (MICAM, brevet 1993) de fixation des sondes par électrolyse
permettant la fabrication de puces de très haute résolution, le CEA a également
décidé récemment d'investir dans la synthèse
in situ
. Les chercheurs
grenoblois
du LETI-CEA, dans le cadre du programme AMIGO, s'attachent à faire évoluer la
technologie des biopuces dans trois domaines.
Dans l'approche issue de la microfluidique, une première évolution
concerne les
façons de déposer les gouttes, c'est-à-dire la dispense du liquide par des
procédés
issus des techniques de jet d'encre. Le LETI-CEA utilise des dispenseurs de type
piézo-électrique.
La seconde évolution concerne la préparation du substrat : la
surface de la
biopuce n'est plus plate mais constituée d'un substrat structuré offrant une
succession
de cuvettes gravées dans du silicium et préparées pour recevoir les
microgouttes.
La troisième évolution consiste à utiliser les robots de dispense non
plus pour
déposer des sondes précédemment synthétisées, mais des réactifs chimiques
permettant
de construire les sondes oligonucléiques directement sur le substrat,
in
situ
.
Quatre tête de dépôt sont utilisées, distribuant sélectivement les bases A, T,
C ou
G.
La détection des hybrides
Elle permet de
repérer avec quelles sondes se sont appariés les brins d'ADN mis au contact de
la
biopuce, c'est-à-dire ceux qui ont la séquence complémentaire. D'une manière
générale, la détection des hybrides se fait grâce au marquage préalable des
sondes à
l'aide de produits fluorescents.
Les progrès à réaliser dans ce domaine consistent à supprimer l'étape du
marquage et
de procéder à une lecture directe de l'hybridation simplifiant ainsi la
réalisation du
diagnostic et abaissant son coût.
- Dans le cadre du programme ROSA, l'École centrale de Lyon a développé un
concept
nouveau dans lequel l'hybridation est détectée par mesure des variations des
charges
électriques provoquées par la formation d'hybrides.
" En utilisant comme récepteur de simples brins d'acides
nucléiques, et en
faisant des mesures systématiques à toutes les étapes de l'élaboration de la
structure
EDS, (Électrolyte/Diélectrique/Semi-conducteur) nous avons montré que
l'hybridation de
brins d'ADN induit un effet de champ dans la structure semi-conductrice
sous-jacente. Le
potentiel de bandes plates du semi-conducteur est ainsi lié à la répartition des
charges de surface, qui varie avec l'hybridation.
S'appuyant sur ces résultats, l'élaboration d'un biocapteur détectant
directement
l'hybridation d'ADN est donc envisageable en utilisant un biorécepteur
constitué de
brins simples d'ADN, et un transducteur capable de détecter des variations de
charges
électriques par effet de champ, à savoir par exemple, une structure FET (Fiel
Effect
Transistor ou transistor à effet de champ).
Un prototype de biocapteur d'acides nucléiques appelé GenFET permettant la
détection
directe et
in situ
de séquences d'ADN a été
élaboré "
77(
*
)
.
- Quant aux chercheurs du Département de recherche fondamentale sur la
matière
condensée du CEA, ils ont choisi d'utiliser, pour détecter les hybrides, les
variations
de poids de la biopuce. En effet, lorsqu'il s'hybride, l'ADN testé ajoute sa
masse sur la
puce. Mais il n'existe pas de balance suffisamment précise à l'échelle de la
puce ! Les systèmes les plus précis du monde, les microbalances à quartz,
peuvent
peser environ 1 nanogramme, soit un milliardième de gramme, l'équivalent de
100 millions de brins d'ADN. Les chercheurs ont donc élaboré un détecteur
comportant 100 millions de brins identiques où pourront s'hybrider les
séquences
d'ADN testées, qui joue en même temps le rôle de balance.
Il leur reste maintenant à miniaturiser l'ensemble afin que plusieurs
détecteurs de ce
type puissent être mis sur le même support et permettent de tester plusieurs
séquences
d'ADN différentes.
Cette technique n'en est qu'à ses débuts et doit être perfectionnée. Outre son
intérêt intrinsèque, elle suggère deux réflexions.
- la recherche bénéficie toujours des dialogues et des collaborations
entre des
chercheurs d'horizons différents (en l'occurrence, recherche fondamentale sur
la matière
condensée et biologie...) ;
- il est indispensable d'encourager et de soutenir financièrement les
chercheurs
français essayant de trouver des technologies innovantes et originales de
fabrication et
de " lecture " des biopuces.
2.2. POUR L'INDUSTRIE
2.2.1. LE TISSU INDUSTRIEL
2.2.1.1. L'aide aux start-up : capital-risque et bio-incubateurs
L'Europe a pris
beaucoup de retard dans le secteur de la biotechnologie.
En 1998, les États-Unis comptaient plus de 1 300 entreprises de
biotechnologie
(ou " biotech ") représentant 153 000 emplois et
plus de
13 milliards de dollars de recettes annuelles, alors qu'en Europe il
n'existe que
1 200 " biotech ", représentant
46 000 emplois et
un chiffre d'affaires de 3,7 milliards d'euros.
On estime que, dans un proche avenir, les activités liées à la génétique au sens
large du terme représenteront un marché mondial de 110 à 120 milliards de
dollars
et que près de 20 % des nouvelles molécules pharmaceutiques seront issues
des
biotechnologies.
Il est donc indispensable de renforcer la position de l'Europe et, tout
particulièrement,
celle de la France.
Le rapport annuel du cabinet de conseil Ernst & Young sur les
biotechnologies faisait
état, pour l'année 1997 d'un recul de la France, qui se situait, dans le
secteur des
sciences de la vie, au troisième rang européen derrière la Grande-Bretagne et
l'Allemagne.
|
PRINCIPALES SOCIÉTÉS DE BIOTECHNOLOGIE FRANÇAISES |
|||
|
Société |
Ville |
Domaine |
|
|
|
Ap Cells |
Paris |
Immunothérapie |
|
|
Atlangene Applications |
Nantes |
Diagnostics moléculaires |
|
|
BioAlliance Pharma SA |
Paris |
Pénétration et ciblage intracellulaire des médicaments |
|
|
Bioprotein Technologies |
Paris |
Protéines recombinantes |
|
|
Biospace Instruments |
Paris |
Instrumentation biologique
|
|
|
Biotech Inflection Point |
Paris |
R&D Pharma Biotech |
|
|
Biovector Therapeutics |
Labege |
Délivrance de principes actifs |
|
|
Bio X Tal |
Roubaix |
Biologie structurale |
|
|
Cayla |
Toulouse |
Thérapie génique |
|
|
Cerep |
Paris |
Chimie combinatoire,
|
|
|
D-Genos |
Angers |
Diagnostic microbiologique |
|
|
Flamel Technologies |
Vénissieux |
Libération contrôlée de principes actifs |
|
|
Genefit |
Loos-Lille |
Génomique fonctionnelle |
|
|
Genopoïetic |
Paris |
Thérapie génique |
|
|
Genoway |
Saint-Cloud |
Conception de modèles de recherche |
|
|
Genset SA |
Paris |
Informations génomiques pour la pharmacie |
|
|
Hemox Therapeutics |
Paris |
Thérapie cellulaire |
|
|
Hybrigenics |
Paris |
Génomique fonctionnelle - protéomique |
|
|
IDM |
Paris |
Thérapie cellulaire pour le cancer |
|
|
Imtix-Sangstat |
Lyon |
Protection immunologique des greffes d'organes |
|
|
Meristem Therapeutics |
Clermont-Ferrand |
Protéines recombinantes à usage thérapeutique à partir de plantes |
|
|
Neurotech SA |
Évry |
Thérapie génique cellulaire |
|
|
Nicox SA |
Valbonne Sophia-Antipolis |
R&D pharmaceutique |
|
|
Proteus SA |
Nîmes |
Découverte de nouvelles enzymes industrielles |
|
|
Qiagen SA |
Courtaboeuf |
Technologies innovantes séparation / purification acides nucléiques |
|
|
Regentech |
Paris |
Cicatrisation et régérération cellulaire |
|
|
Syntem |
Nîmes |
Découverte et optimisation de nouvelles molécules bio-actives |
|
|
Transgène |
Strasbourg |
Thérapie génique |
|
|
Tripos Sarl |
Antony |
Informatique moléculaire (78( * )) |
En 1998, on
comptait en France un peu moins de 100 " biotech ", la
majorité
de ces sociétés étant des microstructures employant moins de 10 personnes
et dont
la pérennité est loin d'être assurée.
Depuis un an la situation se transforme rapidement et pourrait permettre
l'indispensable
rebond de la France.
Le soutien financier : le capital-risque
Le
capital-risque est le meilleur moyen de financer des start-up de biotechnologie
qui ne
peuvent générer de profit à très court terme mais doivent, en revanche,
consacrer de
fortes sommes à leur budget de recherche.
En France, dans le domaine du capital-risque, 1998 a été une année de rupture.
Les fonds disponibles dans les sociétés de capital-risque ont connu une
augmentation
substantielle : 4 milliards de francs de capital-risque ont été levés
en 1998
contre 1,5 milliards en 1997 ; il existe maintenant une vingtaine
d'opérateurs
de taille nationale contre la moitié l'année précédente . Près de
100 sociétés sont cotées au Nouveau Marché, contre 38 au début de 1998.
Par ailleurs, la puissance publique a manifesté sa volonté d'aider les
entreprises
technologiques en débloquant au premier trimestre 1999 une enveloppe budgétaire
de
200 millions de francs dont la moitié est destinée à constituer des
" fonds de capital-amorçage ". Ceux-ci sont destinés à être
investis exclusivement dans les petites et moyennes entreprises notamment dans
les
" biotech ".
Enfin, l'État a constitué un Fonds public pour le capital-risque d'un montant de
600 millions de francs prélevé sur l'ouverture du capital de France
Télécom,
auquel se sont ajoutés 300 millions de francs apportés par la Banque
européenne
d'investissement (BEI). Le Fonds public pour le capital-risque est géré par la
Caisse
des dépôts et placé sous la tutelle d'un comité d'engagement et d'orientation,
composé de représentants de l'administration et de trois chefs d'entreprise,
présidé
par Henri GUILLAUME, auteur du rapport " Technologie et
innovation ".
Outre l'impulsion en faveur des jeunes entreprises, l'intérêt de ce fonds est
l'originalité de son fonctionnement. Il n'intervient pas directement dans les
entreprises
mais aide à la constitution de Fonds commun de placements à risques (FCPR) qui
prennent
eux-mêmes des participations dans les PME.
Ainsi, l'argent public s'associe à
l'argent privé pour réaliser un véritable effet de levier
. L'objectif est
de faire
passer le nombre de structures de capital-risque actives en France de huit à
près de
vingt-cinq. Ces Fonds communs de placement à risques doivent avoir un capital
d'au moins
100 millions de francs ; leurs capitaux doivent être majoritairement
privés ; ils doivent s'engager à financer à hauteur d'au moins 50 %
des
entreprises françaises, innovantes, créées il y a moins de sept ans.
Le Fonds public pour le capital-risque et le Fonds BEI peuvent apporter 15 à
30 %
des capitaux, pour des montants compris entre 15 et 90 millions de francs.
Trois catégories de FCPR ont jusqu'à présent bénéficié des fonds publics pour le
capital-risque :
- des fonds nationaux d'une taille de 300 à 800 MF pouvant financer
des projets
lourds avec des interventions pouvant atteindre 30 à 50 MF par
affaire ;
- des fonds régionaux, d'une taille plus réduite, de 120 à 200 MF, qui
financeront des PME régionales avec des montants compris entre 1 et
10 MF ;
- des fonds créés par des personnes physiques, spécialistes d'un secteur,
pouvant
investir dans les phases d'amorçage ou de création (
" business
angels "
).
Les fonds nationaux ayant bénéficié de ces mesures sont : Sofinnova, Banexi
Ventures, Auriga Ventures, Siparex Ventures, Galileo Ventures, Natexis Ventech.
Le fonds Auriga Ventures, doté de 410 millions de francs, a exprimé
l'intention
d'investir 50 % de ses actifs dans le domaine des sciences de la vie, pour
une forte
majorité en France mais aussi dans le reste de l'Europe. Il a déjà décidé
d'investir
14,5 millions de francs dans la société Hybrigenics, essaimage de
l'Institut
Pasteur, dans le cadre d'un deuxième tour international de 60 millions de
francs. En
terme sectoriel, Auriga Ventures privilégie les sociétés orientées vers la
post-génomique, c'est-à-dire la protéomique.
Le soutien logistique : les bio-incubateurs
L'aide
financière ne suffit pas pour induire un véritable démarrage des jeunes
entreprises de
biotechnologie. Les start-up connaissent en effet au moment de leur naissance
des
problèmes matériels divers : immobiliers, juridiques, commerciaux ou de
gestion.
Les incubateurs sont des structures d'accueil qui aident les créateurs
d'entreprises
à :
- s'installer matériellement, avec des locaux et du matériel de
qualité ;
- réaliser des études de faisabilité de leur projet ;
- s'assurer de la brevetabilité de la technologie envisagée
- négocier éventuellement avec les organismes ou groupes détenteurs des
brevets ;
- mener des études de marché ;
- établir un
" business plan "
;
- trouver des collaborateurs capables d'assurer la gestion.
Les Britanniques ont mis en oeuvre un tel soutien logistique depuis plusieurs
années.
BIO INCUBATEURS : L'EXEMPLE BRITANNIQUE 79( * )
" Pour
faciliter le transfert de technologie du laboratoire vers l'industrie, les
Britanniques
ont adopté le système des bio-incubateurs. Le concept est bien plus qu'une
opération
immobilière visant à offrir aux start-up des locaux munis d'équipement dernier
cri.
Au sein de l'incubateur, les jeunes pousses ont aussi à leur disposition toute
une gamme
de services d'accompagnement. Bien souvent les chercheurs qui s'improvisent
entrepreneurs
n'ont pas les compétences dans des domaines aussi variés que la propriété
intellectuelle, le droit, le marketing, la fabrication ou encore la finance.
" Notre étude de marché aux États-Unis et en Europe a révélé qu'il
existe
plus de 1 000 bio-incubateurs. Mais, seuls ceux qui avaient une équipe
complète d'accompagnement ayant une expérience commerciale
réussissent ",
selon David BEST, du Bioscience Innovation Centre, à Cambridge.
La plupart à l'image de l'incubateur d'Oxford-BiotechNet-, sont financés par une
combinaison de fonds publics et privés. À Manchester, une partie du financement
est
issue du capital-risque ou de fonds d'amorçage. En invitant aussi des
compagnies plus
avancées dans leur développement, le directeur, Mark FERGUSSON, augmente les
chances de
retour d'investissement dans un délai acceptable. Enfin, pour les sociétés qui
peuvent
rester dans les locaux de leur université d'origine, il y a les bio-incubateurs
virtuels.
L'un d'eux, le Company Maker, d'Imperial College, se contente ainsi d'apporter
un
savoir-faire au niveau du management et du financement. "
En Allemagne, une pépinière d'entreprises accueille les start-up sur le campus
de Berlin
Buch. Avec un loyer inférieur à 30 francs le mètre carré, des équipements
ultramodernes répartis sur 6 000 m
2
et une connexion à un
réseau
de transmission de données à grande vitesse, cet incubateur a déjà attiré plus
d'une
vingtaine d'entreprises.
En France, les biopôles se dotent aussi d'incubateurs. Au Génopôle d'Évry, par
exemple
le bâtiment d'accueil des start-up prévoit de leur offrir
" une
aide
juridique à la commercialisation et à la communication
d'entreprise "
.
C'est à ces incubateurs chargés d'accompagner les entreprises en phase de
création
qu'est consacrée l'autre moitié de l'enveloppe budgétaire prévue pour 1999, soit
environ 100 millions de francs. Une vingtaine d'incubateurs devraient être
mis en
place dans l'Hexagone, au sein d'universités ou d'organismes de recherche
publics. Chacun
de ces lieux d'accueil pourrait héberger entre 15 et 20 start-up pendant
une durée
limitée.
2.2.1.2. Les biopôles
Le concept de biopôle et son développement en Europe
Les biopôles
sont des lieux de contact. Généralement créés autour d'un centre universitaire
ou de
recherche, ils réunissent autour des scientifiques une nébuleuse de sociétés qui
couvrent tous les domaines des biotechnologies : entreprises de service,
grands
groupes pharmaceutiques, cabinets de consultation, audit, capital-risqueurs,
spécialistes
de la propriété intellectuelle... Le but de ces contacts multiples est de faire
naître
une synergie entre les différents acteurs, industriels, entrepreneurs ou
chercheurs.
D'après Michel RENAUD, vice-président de l'Université d'Auvergne et fondateur du
biopôle de Clermont-Limagne :
" Pour créer une entreprise, il faut un " porteur de
projet ". On pense souvent aux chercheurs eux-mêmes, mais est-il
vraiment
souhaitable d'amputer les laboratoires de leurs meilleurs éléments ? Pour
moi, les
bons porteurs de projet ce sont les jeunes diplômés en général. Les
" ex-post docs " notamment connaissent bien leur
discipline, ils ont
pour beaucoup une ouverture internationale et ils sont, plus souvent qu'on ne
le pense,
ouverts à l'esprit d'entreprise. Ce sont ces personnes-là qu'il faut chercher,
former et
encourager. [...] Mais il ne suffit pas de penser en termes de créations
d'entreprise : pour que la source ne se tarisse pas, la recherche doit être
développée. [...] Enfin, toujours dans l'idée d'une chaîne continue et d'une
fertilisation croisée, il faut un lieu qui accueille les entreprises, mais pas
seulement
les PME de haute technologie. Le Biopôle Clermont-Limagne a une logique de
site, presque
de filière industrielle : à terme, il devra réunir toutes les entreprises
nécessaires pour traiter un problème biotechnologique sous différents angles, y
compris
les services. "
80(
*
)
Ces propos illustrent la multiplicité des acteurs susceptibles de collaborer
pour qu'un
biopôle réalise la nécessaire synergie entre le monde de la science et celui des
entreprises. On peut ajouter que le rôle d'un biopôle est aussi, en favorisant
la
proximité de tous les acteurs, qu'ils soient scientifiques, industriels
confirmés ou
créateurs de start-up, de permettre le transfert de ce que l'on appelle
l'information
" non codifiée ", le savoir tacite (
" tacit
knowledge "
).
Si le nombre d'entreprises de biotechnologie en Europe reste insuffisant, son
augmentation
rapide (45 % en 1998) est sans conteste liée à la multiplication des
biopôles en
Europe.
De nombreux centres nationaux sont nés :
- En Grande-Bretagne : Cambridge, Oxford, Edimbourg ;
- En Allemagne : Munich, Rhénanie (Cologne, Düsseldorf, Wuppertal,
Aix-la-Chapelle, Jülich), Triangle Rhin-Neckar (Heidelberg, Ludwigshafen,
Mannheim),
Berlin-Brandebourg, Iéna ;
- En Irlande : Dublin ;
- Aux Pays-Bas : Leiden-Wageningen ;
- En Belgique : bio-Flandres et
Louvain-La-Neuve/Bruxelles-Sud/Charleroi ;
- En Finlande : Turku ;
- En Italie : Milan.
Au niveau de la Communauté européenne, on peut noter :
- que les biopôles bénéficient d'aides financières (ainsi, au biopôle de
Clermont-Ferrand, le Fonds européen de développement économique régional a
financé
pour moitié les coûts de création de cinq nouveaux laboratoires) ;
- que le rassemblement des start-up au sein des biopôles permet une
meilleure
coordination de leurs activités, notamment pour répondre à des appels d'offres
européens.
En France, les quatre principaux biopôles sont le Génopôle d'Évry,
Clermont-Ferrand,
Lille Eurasanté et Montpellier.
|
GÉNOPÔLE 81( * ) |
|
|
|
Dominante : génomique |
|
|
Principales bases de recherche : Généthon, Centre national de séquençage, Centre national de génotypage |
|
|
Nombre de PMI innovantes : 8 |
|
|
Leaders : Genset, Département génomique de Rhône-Poulenc Rorer, Neurotech, Visible Genetics/ACT Gene, Cybergène/ESGS, Novagali |
|
|
Atouts de développement : volonté publique de faire du Génopôle le pôle génomique français ; neuf installations de start-up prévues ; concentration scientifique et économique de la région parisienne |
|
CLERMONT-FERRAND 1 |
|
|
|
Dominante : filière agro-alimentaire |
|
|
Principales bases de recherche : premier centre de province de l'INRA, laboratoires INSERM et CNRS, Centre de recherche en nutrition humaine, Université d'Auvergne |
|
|
Nombre de PMI innovantes : 15 |
|
|
Leaders : GreenTec, Genolife, Meristem Therapeutics |
|
|
Atouts de développement : présence de Limagrain ; stratégie du pôle ; mise en réseau des centres de Clermont-Limagne, de Vichy (cosmétique-parapharmacie) et Aurillac (environnement-agro-alimentaire) |
|
LILLE EURASANTÉ 82( * ) |
|
|
|
Dominante : santé |
|
|
Principales bases de recherche : Institut Pasteur Lille, CHRU, Institut de chimie pharmaceutique |
|
|
Nombre de PMI innovantes : 10 |
|
|
Leaders : Cerep, PIL-Biovector Therapeutics |
|
|
Atouts de développement : dynamisme et originalité des projets (Genfit) |
|
MONTPELLIER 1 |
|
|
|
Dominante : expertise dans la génomie du riz et dans la recherche sur le cancer |
|
|
Principales bases de recherche : Génotrop de l'Orstom-IRD, Laboratoire du Cirad, Laboratoire de biologie moléculaire et cellulaire du CNRS, Laboratoire des biotechnologies de l'environnement, Université de Montpellier I |
|
|
Nombre de PMI innovantes : 6 |
|
|
Leaders : DNA, Vitropic, Mycos |
|
|
Atouts de développement : dynamisme ; plusieurs projets d'incubateurs ; ouverture prochaine d'une pépinière dans les biotechnologies |
Sélection et coopération des biopôles
S'il est
indispensable de favoriser la création de biopôles, il convient de le faire
dans les
meilleures conditions possibles, en pratiquant la sélection et en organisant la
coopération.
- La sélection :
Elle est indispensable pour que les crédits
de soutien
ne soient pas disséminés, donc inefficaces, et pour que la
" labellisation " des biopôles corresponde à une véritable
excellence, reconnue à l'extérieur.
En France, elle sera effectuée en automne 1999 dans le cadre d'une des actions
concertées incitatives (ACI) créées par le Conseil interministériel sur la
recherche
et la technologie. Plusieurs sites parisiens et huit villes de provinces
concourent à
l'appel d'offres (Bordeaux, Lille, Strasbourg, Lyon, Grenoble, Marseille,
Montpellier et
Toulouse).
Afin de réaliser cette sélection dans de bonnes conditions, il serait
souhaitable de
prendre en considération les méthodes utilisées par les Allemands dans le cadre
du
programme Bioregio : en 1995, le Ministère fédéral allemand pour
l'enseignement,
la recherche, la science et la technologie a lancé un concours appelé Bioregio
afin
d'accélérer le développement industriel des biotechnologies.
Le ministère entendait ainsi encourager la coordination de toutes les sources de
financement de recherche-développement et la création d'entreprises en synergie
régionale dans ce domaine. Les régions devaient formuler leur stratégie et les
objectifs de leurs recherches tout en faisant état du potentiel biotechnologique
régional. Trois dossiers (sur dix-sept) ont été sélectionnés par le jury
constitué
de membres issus de l'industrie, de la recherche scientifique et des
organisations
syndicales : celui de la Rhénanie, du triangle Rhin-Neckar et de Munich (à
cela
s'est ajouté un prix exceptionnel obtenu par la biorégion Iéna) : les trois
régions " gagnantes " se sont vu accorder, chacune,
50 millions
de DM sur cinq ans, ces moyens devant leur permettre de mobiliser le plus
possible de
financements privés.
La participation de l'État français, pour l'année 1999 est moins importante
mais elle
s'inscrit dans la durée. Afin d'être vraiment efficace l'action des pouvoirs
publics
doit se situer dans une optique précise :
- Cette action concertée incitative doit être l'occasion de dresser un
inventaire aussi exhaustif que possible des atouts de chaque région dans le
domaine de la
génomique (infrastructure, organismes de recherche, possibilités de financement
privé,
opportunités industrielles, incubateurs, réseaux informatiques...).
Cet inventaire sera en effet indispensable aux régions comme aux décideurs
nationaux,
aux groupes industriels et financiers, et aux organismes internationaux qui ne
peuvent
eux-mêmes recenser toutes les possibilités offertes par les régions dans un
domaine
précis mais ont besoin de ces éléments pour leur confier la réalisation de
programmes
(c'est le cas de la région de Montpellier et d'un programme de la Banque
mondiale).
- Le recours à des experts internationaux pour l'évaluation des dossiers
présentés par les régions doit être systématique afin, bien sûr de
profiter
de leur expérience et de leur impartialité mais aussi de faire en sorte que la
labellisation " biopôle " des régions sélectionnées soit
reconnue
internationalement.
- La coopération
au niveau national : elle est indispensable pour que les biopôles
sélectionnés aient un effet d'entraînement sur les autres. Elle passe bien
entendu par
l'utilisation de réseaux informatiques mais également par l'accueil, au sein des
biopôles, des start-up n'ayant pas trouvé d'incubateur local.
au niveau européen : elle constitue le meilleur moyen de parvenir à
des
biopôles de taille suffisante pour se mesurer aux sites américains. Elle existe
déjà
dans la péninsule scandinave : la Medicon Valley regroupe le Danemark et
la Suède,
et constitue le troisième centre de recherche en biotechnologies en Europe. Une
autre
initiative particulièrement intéressante est la Biovalley qui regroupe
l'Alsace, le
Bade-Wurtemberg et le Nord-Ouest de la Suisse. L'objectif de la BioValley est
d'atteindre
la taille critique d'un pôle de niveau mondial.
BIOVALLEY
L'espace central
du Rhin Supérieur -Alsace, Bade-Wurtemberg, nord-ouest de la Suisse- réunit
l'ensemble
des composantes favorables au développement d'un pôle majeur des
biotechnologies en
Europe : excellence des centres de recherche, présence de grands groupes
industriels, secteur en forte croissance.
BioValley, dont l'initiative a été lancée officiellement en 1996, recense déjà
plus
de 300 partenaires potentiels : entreprises, institutions, organismes,
tri-nationaux de développement économique, organismes financiers, universités,
centres
de recherche, centres de transfert de technologies.
Le réseau BioValley se mobilise
pour atteindre la taille critique d'un pôle
de niveau
mondial
dans le domaine des biotechnologies en :
favorisant les alliances entre partenaires européens ;
encourageant les alliances entre partenaires européens ;
facilitant l'accès des jeunes entreprises au capital-risque ;
identifiant les travaux de recherche académique susceptibles d'être
valorisés ;
accélérant le transfert de technologies.
Actuellement, le concept BioValley devient réalité grâce à la diversité des
services
et des prestations proposées :
Un réseau trinational " Vie et Santé "
avec des
partenaires dans les trois pays impliqués ;
BioValley Promotion Team
pour la coordination des activités au
sein du
réseau ;
Tables rondes :
rencontres mensuelles à Bâle, Fribourg et en
Alsace ;
BioValley
" Easy Access System "
:
accès
simple et facile aux contacts du réseau, que ce soit de l'intérieur ou de
l'extérieur
de la BioValley ;
Fonds d'expertise BioValley :
aide au financement pour les
chercheurs
souhaitant créer leur propre entreprise et aide à l'élaboration d'un business
plan afin
de faciliter les contacts avec les financiers ;
Guide BioValley :
annuaire de tous les partenaires de la
BioValley ;
BioValley Technology Watch :
la BioValley
"
Promotion
Team "
en collaboration avec d'autres institutions mettent en
place une
veille technologique en biotechnologie ;
Site Internet :
http ://www.biovalley.com :
présentation de
BioValley, partenaires de BioValley, forum et
newsgroups
, base de
données ;
Point de rencontre BioValley :
événements, séminaires,
rencontres et
conférences ;
Foires et congrès :
participation de la BioValley du Rhin
Supérieur
aux foires, salons et congrès ;
BioValley Bridges :
conférences multidisciplinaires et
rapports pour
l'harmonisation des normes et législations entre les régions de la BioValley,
en se
basant sur les directives européennes ;
Formation continue :
rencontres et séminaires de formation
proposés
par les organismes de formation continue et initiale de la BioValley ;
BioValley Universities Partnership Programm :
programme
d'accompagnement
des universités, afin de favoriser la création d'entreprises et la collaboration
universités-entreprises.
(83( * ))
2.2.2. LES BREVETS
2.2.2.1. Aspect juridique : le champ de la brevetabilité
Le texte de base pour la Communauté européenne : la directive de juillet 1998
La directive
98/44 du Parlement européen et du Conseil, du 6 juillet 1998
84(
*
)
,
relative à la protection juridique des inventions biotechnologiques a été
adoptée
après dix années de tergiversations. Le nombre de considérants qui l'accompagne
(cinquante-six considérants pour dix-huit articles) prouve combien les termes
de cette
directive ont été pesés et ont fait l'objet de compromis.
Elle a cependant le mérite d'être très explicite.
Elle rappelle la distinction fondamentale entre les
découvertes
, par
essence non
brevetables et les
inventions
qui, seules, le sont. Elle confirme la non
brevetabilité du corps humains aux différents stades de sa constitution et de
son
développement. De même, la simple découverte d'un élément du corps humain dans
son
environnement naturel,
" y compris la séquence ou la séquence
partielle
d'un gène "
ne peuvent constituer des inventions brevetables.
En revanche, un élément isolé du corps humain, dès lors qu'il est le résultat de
procédés techniques visant à l'identifier, le purifier, le caractériser et le
multiplier est brevetable
" même si la structure de cet élément est
identique à celle d'un élément naturel "
85(
*
)
.
Par exemple, le travail d'identification d'un gène nécessitant l'intervention
de la
technologie humaine ne constitue pas une simple découverte et peut être breveté
s'il
remplit, par ailleurs, les conditions classiques de brevetabilité :
l'application
industrielle d'une séquence génétique partielle ou complète doit
obligatoirement être
concrètement exposée dans la demande de brevet. Une simple séquence d'ADN sans
indication de fonction ne peut être brevetée.
Certains considérants relatifs à l'article 5 apportent des éléments précis
d'information :
" (20) considérant, en conséquence, qu'il est nécessaire
d'indiquer qu'une
invention qui porte sur un élément isolé du corps humain ou autrement produit
par un
procédé technique, et qui est susceptible d'application industrielle, n'est pas
exclue
de la brevetabilité, même si la structure de cet élément est identique à celle
d'un
élément naturel, étant entendu que les droits conférés par le brevet ne
s'étendent
pas au corps humain et à ses éléments dans leur environnement naturel ;
(21) considérant qu'un tel élément isolé du corps autrement produit n'est pas
exclu de
la brevetabilité puisqu'il est, par exemple, le résultat de procédés techniques
l'ayant identifié, purifié, caractérisé et multiplié en dehors du corps humain,
techniques que seul l'être humain est capable de mettre en oeuvre et que la
nature est
incapable d'accomplir par elle-même ;
(22) considérant que le débat sur la brevetabilité de séquences ou de séquences
partielles de gènes donne lieu à des controverses ; que, aux termes de la
présente
directive, l'octroi d'un brevet à des inventions portant sur de telles
séquences ou
séquences partielles doit être soumis aux mêmes critères de brevetabilité que
pour
tous les autres domaines technologiques, nouveauté, activité inventive et
application
industrielle ; que l'application industrielle d'une séquence ou d'une
séquence
partielle doit être exposée de façon concrète dans la demande de brevet telle
que
déposée;
(23) considérant qu'une simple séquence d'ADN sans indication d'une fonction ne
contient
aucun enseignement technique; qu'elle ne saurait, par conséquent, constituer une
invention brevetable ;
(24) considérant que, pour que le critère d'application industrielle soit
respecté, il
est nécessaire, dans le cas où une séquence ou une séquence partielle d'un gène
est
utilisée pour la production d'une protéine ou d'une protéine partielle, de
préciser
quelle protéine ou protéine partielle est produite ou quelle fonction elle
assure. "
La jurisprudence
- La
jurisprudence européenne a montré les difficultés qui pouvaient surgir
lorsqu'on tente
d'appliquer les règles classiques de brevetabilité aux sciences du vivant,
notamment
pour la définition de " l'invention " et de la
" découverte ".
En 1991, le Howard Florey Institute avait obtenu, de la part de l'Office
européen des
brevets (OEB), un brevet portant sur un fragment d'ADN codant pour une protéine
humaine,
la Relaxine H2. Cette protéine, sécrétée par les femmes enceintes pour atténuer
les
contractions, a un intérêt thérapeutique évident.
Le brevet accordé fit l'objet d'une procédure d'opposition qui permit à la
division
d'opposition de l'OEB de rendre une décision importante le 8 décembre
1994 :
elle a jugé que l'invention de la Relaxine H2 remplissait les conditions de
nouveauté et d'activité inventive, considérant qu'il s'agissait d'une substance
naturelle isolée pour la première fois et dont l'existence était inconnue
auparavant ; elle a estimé que cette substance ne pouvait s'assimiler à une
découverte dans la mesure où un procédé permettant de l'obtenir et de la
caractériser
convenablement par sa structure avait été mis au point, soulignant ainsi
l'importance,
pour la brevetabilité de l'intervention humaine permettant de révéler une chose
jusque-là ignorée dans la nature
86(
*
)
. Cette
décision
n'avait pas, alors, fait l'unanimité.
La clarté de la directive européenne, précisant qu'une simple séquence d'ADN
sans
fonction n'est pas brevetable, devrait permettre aux diverses instances
juridiques des
États membres d'harmoniser leur jurisprudence.
- La jurisprudence américaine est beaucoup moins claire.
La notion d'utilité nécessaire à l'obtention d'un brevet laisse à penser
que
l'on ne peut pas breveter un gène dont l'utilisation pratique n'est pas
précisée. Sur
cette idée se fondait le rejet, par l'Office américain des brevets, des demandes
formulées en 1991 et 1992 par Craig VENTER, l'un des responsables du programme
américain
de séquençage du génome humain. Celui-ci avait déposé, pour le compte des
Instituts
nationaux de la santé (NIH), une demande de brevet sur plusieurs centaines de
gènes
humains. Dans un premier temps, l'Office américain des brevets a rejeté cette
demande
pour " défaut d'utilité " de ces gènes ; mais, les NIH
ayant
décidé, devant l'émoi de la communauté scientifique, de ne pas aller au terme
de la
procédure, la jurisprudence américaine est restée imprécise.
La confusion a même été accentuée par une récente décision de l'Office
américain des brevets, qui a accordé à la société Incyte le premier brevet sur
des
EST. Les EST (
Expressed Sequence Tags
) sont de courtes séquences d'ADN
utilisées
pour " étiqueter " les gènes et permettre de décoder de
longues
séquences d'ADN.
Les EST n'ont aucune fonction biologique précise. L'Office américain a
cependant admis
qu'un EST pouvait être utile si les applications potentielles étaient
suffisamment
décrites : identification de gènes ayant eux-mêmes une fonction connue,
cartographie chromosomique.
Dans ces conditions, le problème est moins celui de la brevetabilité que celui
de la
portée du brevet. En effet, la crainte principale des chercheurs est que le
" propriétaire " d'un EST n'ait également des droits sur le
gène
complet séquencé grâce à l'outil que constitue l'EST.
Certes, accorder des droits sur un gène de fonction inconnue, en s'appuyant sur
des
droits reconnus sur un sous-ensemble du gène, ne serait pas conforme au droit
américain
des brevets
87(
*
)
.
Mais cette décision de l'Office américain des brevets va donner lieu à des
batailles
juridiques pouvant avoir des solutions différentes selon les instances
saisies.
2.2.2.2. Aspect économique : l'impact des brevets
L'initiative
prise le 15 avril 1999 par les plus grands groupes pharmaceutiques
mondiaux change la
donne économique en matière de brevets sur le génome.
Sous l'égide de Novartis, les groupes AstraZeneca, Bayer, Bristol-Myers Squibb,
Hoffmann-La Roche, Glaxo-Wellcome, Hoechst Marion Roussel, Pfizer, Searle et
SmithKline
Beecham se sont associés à cinq grands laboratoires publics directement
impliqués dans
la recherche sur le génome humain : Whitehead Institute (MIT) for genome
research,
Sanger Center (Wellcome Trust), Cold Spring Harbor Laboratory, Stanford Human
Genome
Center, Washington University School of Medicine.
Le but du SNP Consortium ainsi constitué est l'établissement d'une carte de
marqueurs
génétiques du polymorphisme. Les SNP (
Single Nucleotide Polymorphism
)
sont des
sites du génome présentant des variations d'un seul nucléotide. La connaissance
des
SNP, qui sont répartis de façon homogène sur le génome (1 pour 1000 bases
environ) devrait accélérer la localisation des gènes associés aux pathologies.
Le SNP Consortium a pour objectif d'identifier, parmi les 3 milliards de
paires de
nucléotides formant le génome humain, environ 300 000 SNP. Les
chercheurs
pourront ensuite comparer les cartes de SNP obtenues chez des personnes non
malades avec
celles de groupes de personnes souffrant de telle ou telle affection.
Cette comparaison permettra de réaliser les génotypages nécessaires au
développement
de nouvelles pistes diagnostiques et thérapeutiques.
Or, les responsables du consortium n'envisagent pas, semble-t-il, de garder
pour eux ces
précieuses informations ni de les faire breveter. Ils ont indiqué, en effet,
que les
informations issues de leurs recherches seraient immédiatement rendues
publiques et mises
à la disposition de l'ensemble de la communauté scientifique internationale.
Mais leur
démarche n'est pas exclusivement philanthropique, malgré certaines
déclarations :
" Notre
mission consiste à rendre largement accessible un important outil de recherche
qui fera
progresser notre compréhension des processus des maladies et, par extension, le
champ de
la médecine humaine "
88(
*
)
...
Leur but est surtout de partager le risque financier et de réduire la
duplication des
efforts de recherche qui résulteraient d'une situation où chaque grand groupe
pharmaceutique chercherait à établir pour son propre compte des cartes de SNP.
Enfin, leur démarche vise à ne pas dépendre entièrement de plus petites
sociétés,
propriétaires de l'information génétique. En effet, les principales sociétés de
génomique (Celera, Affymetrix, Millenium Pharmaceuticals, Incyte aux États-Unis
et
Genset en France) consacrent des sommes très importantes à l'établissement de
cartes
des SNP afin de faire breveter au plus vite leurs découvertes et de vendre ces
informations aux grands laboratoires pharmaceutiques.
Dès lors que les cartes de SNP seront du domaine public, l'impact des brevets
va se
déplacer pour aller en aval : les sociétés de génomique ne pourront plus
vendre
des données brutes. C'est ce qu'expose le président directeur général de Genset,
Pascal BRANDYS :
" Il est certain que les SNP seront un jour
accessibles
à tous ; mais nous n'avons pas attendu pour nous engager dans les étapes
suivantes.
Nous développons des technologies de génotypage et l'analyse biostatistique
pour relier
génotypes et phénotypes "
.
Au niveau français, il serait utile de mettre en place, à l'Institut
national de la
propriété industrielle, sous la tutelle du secrétariat d'État à l'Industrie, une
cellule de réflexion à laquelle participeraient des responsables des
entreprises de
génomique. Cette cellule aurait pour mission de donner rapidement aux décideurs
politiques, aux chercheurs, responsables des biopôles et aux industriels de
tous niveaux
impliqués dans la recherche génomique, des informations sur :
- les conséquences de la création du SNP consortium dans le domaine des
brevets ;
- les possibilités de réorientation de la stratégie en matière de
brevetabilité
de l'information génétique.
2.3. POUR LA SOCIÉTÉ
2.3.1. LA FORMATION PROFESSIONNELLE
2.3.1.1. Les biologistes
Voici quelques
recommandations pour adapter la formation des biologistes aux nouveaux
besoins :
- Favoriser l'éclosion de " vocations " pour la
biologie :
il conviendrait, dans cette optique, d'offrir à tous les
étudiants
de médecine et de pharmacie qui ne sont pas reçus au concours de fin de première
année, à cause du
numerus clausus
, la possibilité de passer directement
en
deuxième année du DEUG de biologie, à condition d'avoir obtenu soit une moyenne
de 10
dans toutes les matières enseignées en première année de médecine ou de
pharmacie,
soit une moyenne de 12 en biologie. Des passerelles de ce type existent
aujourd'hui
mais :
elles sont inégalement mises en oeuvre en fonction des différentes
facultés ;
elle ne tiennent pas compte de façon spécifique de la note en
biologie ;
elles ne font pas toujours l'objet d'une information bien diffusée chez
les
étudiants ;
elles offrent un nombre limité de places générant un
" gâchis " intellectuel considérable. Il serait très
profitable, en
effet, aux facultés de biologie, de recruter ces étudiants qui ont suivi une
formation
scientifique au cours de leur première année de médecine ou de pharmacie.
- Encourager l'enseignement de la biologie dans les universités situées
près
des biopôles
. Ainsi, l'Université d'Évry a créé un DEUG de sciences de la
vie,
une licence de biologie moléculaire et physiologie, ainsi qu'une maîtrise de
biologie
moléculaire (mention génétique humaine) ; elle envisage aussi d'orienter
vers la
biologie certaines matières déjà enseignées comme l'informatique ou les
mathématiques. Ce type d'initiative favorise considérablement la synergie entre
les
universités et les biopôles.
- Instituer une option " informatique " dans tous
les cursus
de biologie
afin de faciliter le dialogue entre biologistes et
informaticiens et,
éventuellement, de susciter des vocations de bio-informaticiens chez certains
biologistes.
- Créer des écoles doctorales de biologie qui proposent des
spécialisation en
GÉNOMIQUE, BIOTECHNOLOGIE, BIOLOGIE STRUCTURALE, RECHERCHE THÉRAPEUTIQUE et
BIO-INFORMATIQUE
. Les écoles doctorales sont les structures administratives
chargées, au sein des universités et des grandes écoles, de l'organisation des
DEA et
des thèses. Depuis deux ou trois ans certaines écoles doctorales en biologie,
outre
leurs fonction administrative, proposent à leurs étudiants des enseignements les
préparant aux métiers des biotechnologies. Mais ces initiatives ne sont pas
systématiques et dépendent des directeurs de ces écoles. Sur une vingtaine
d'écoles
doctorales françaises, un quart ne propose aucun enseignement de ce type, alors
qu'un
autre quart offre un cursus très intéressant à ses étudiants.
|
LES ÉCOLES DOCTORALES LES PLUS DYNAMIQUES EN 1997-1998 |
|||
|
Écoles |
Nombre d'étudiants |
Organisation des enseignements |
Les " plus " |
|
École doctorale " sciences de la vie et de la santé " de Toulouse |
280 |
- 25 modules.
|
Biostatistiques, législation européenne |
|
École doctorale " biochimie et biologie moléculaire " de Paris-VII |
400 |
- 17 modules.
|
Protection et valorisation des recherches, cours d'économie à l'université de Paris-Dauphine |
|
École doctorale " biologie et santé " de Vandoeuvre-les-Nancy |
400 |
- Accompagnement sur 3 ans du thésard |
Initiation à la démarche qualité |
|
Collège d'études doctorales " sciences et santé " d'Amiens |
115 |
- 11
modules.
|
Gestion de projets, création d'entreprises en collaboration avec l'UTC Compiègne |
(89( * ))
Il est
indispensable de systématiser et d'officialiser la mission d'enseignement des
écoles
doctorales. Il faut également en créer un certain nombre, pour les matières
précitées, dans les universités situées près des biopôles. Elles devraient
dispenser
une double formation et permettre aux étudiants de 3
e
cycle
d'étudier,
parallèlement à leur spécialité, des matières destinées à développer leur
" culture entrepreneuriale " : veille technologique et
intelligence économique, propriété industrielle, réglementation, communication,
capital-risque, connaissance des marchés, gestion des ressources humaines...).
Cette double formation doit pouvoir être reconnue et il convient donc de
modifier les
titres universitaires décernés en fin de cursus pour que les étudiants puissent
se
prévaloir de cette double compétence.
2.3.1.2. Les médecins et les pharmaciens
- À
l'issue de la première année
,
il faut
, ainsi que cela a été exposé
ci-dessus,
favoriser la reconversion en biologie
.
-
Dès le second cycle, les étudiants doivent aborder la génomique
.
Cela a
semblé particulièrement important à la Commission " Génétique et
Pharmaciens " instituée au Conseil national de l'Ordre des
Pharmaciens.
" La Commission estime qu'il est important d'organiser, au niveau
de la
formation commune de base des pharmaciens, un enseignement de sensibilisation à
la
génétique.
Actuellement, ces formations sont très disparates, que ce soit en nombre
d'heures de
cours ou selon les thèmes abordés.
L'enseignement est majoritairement réalisé par des professeurs de différentes
disciplines.
Il semble souhaitable de fixer à la fois un minimum d'heures de formation, de
définir un
contenu plus précis des formations et d'en donner la responsabilité à une ou
deux
disciplines biologiques définies. "
Quant aux médecins, il est indispensable de leur faire connaître l'essor des
tests
génétiques, leur réalisation sur des biopuces et de les sensibiliser aux
problèmes que
posera la médecine prédictive à tous les praticiens, quelle que soit leur
spécialité.
-
Au niveau du troisième cycle
, comme le préconise la Commission
" génétique et pharmaciens ",
il faudrait organiser
une
formation spécialisée de génétique
permettant aux pharmaciens biologistes
d'accéder à une formation diplômante de génétique
.
Une autre de ses propositions est également intéressante :
" La commission souhaite pouvoir
organiser un rapprochement
entre le DES
de génétique
, lequel n'est actuellement ouvert qu'aux médecins,
et le
DES de
biologie médicale
. Ainsi pourraient être associés aux 4 semestres
d'enseignement polyvalent du DES de biologie médicale, 4 semestres
spécialisés en
génétique biologique, enseignement partagé avec les étudiants du DES de
génétique.
Cet enseignement nouveau permettrait la reconnaissance, pour les pharmaciens et
médecins
biologistes, de compétences en génétique jusqu'à présent
inexistantes. "
Par ailleurs, il convient de revitaliser la recherche sur le médicament en
France et
d'assurer un enseignement de haut niveau, favorisant l'application des
découvertes en
génomique au domaine des médicaments. Le Royaume-Uni, les Pays-Bas, la
Belgique, la
Suisse, l'Italie et l'Allemagne l'ont déjà fait. En France, il est nécessaire
de
créer
un troisième cycle de recherche sur le médicament, ouvert aux pharmaciens, aux
médecins
et aux biologistes
.
-
Dans le domaine de la formation continue, il conviendrait de proposer
des
" sessions de génomique " aux médecins et pharmaciens
.
Les pharmaciens biologistes devront bénéficier de cours renforçant leurs
compétences
dans les méthodes relevant de l'analyse des acides nucléiques (biologie
moléculaire).
Quant aux 22 000 pharmaciens d'officine qui auront éventuellement à
dispenser
des produits d'une nature nouvelle, il devront compléter leur formation soit
par des
cours, soit par la consultation d'une documentation très précise. La façon dont
ils se
sont adaptés à la dispensation des antirétroviraux augure bien de l'avenir.
2.3.1.3. Les bio-informaticiens
- Le besoin urgent de bio-informaticiens
La formation
massive de bio-informaticiens est l'enjeu majeur des années à venir
. Elle
est l'un
des moyens que la France doit impérativement utiliser pour retrouver la place
qu'elle
occupait dans le secteur de la génomique au début des années 1990. L'urgence
s'évalue
à la lumière de l'initiative prise par les National Institutes of Health (NIH)
américains pour accélérer considérablement le séquençage du génome humain dont
la
première ébauche sera communiquée au printemps 2000.
Une énorme quantité d'informations brutes, sans aucune annotation, sera mise
à la
disposition de l'ensemble de la communauté internationale. Il faut se garder
d'une
excessive naïveté : ces informations ne pourront être valorisées
scientifiquement
(articles) et économiquement (brevets) qu'après avoir été triées, comparées et
analysées. Ces tâches ne peuvent être accomplies que grâce à la
bio-informatique
.
C'est pourquoi les États-Unis peuvent faire preuve de générosité en offrant ces
données brutes à la communauté internationale ; ils sont très avancés dans
le
secteur bio-informatique et vont accroître leurs capacités : en juin 1999,
un
conseil consultatif fédéral a recommandé aux NIH d'investir lourdement dans
l'informatique et de créer une vingtaine de centres de formation
90(
*
)
.
Au niveau européen a été fondé le European Bioinformatics Institute (EBI),
situé à
Cambridge et placé sous l'égide du Laboratoire européen de biologie moléculaire
d'Heidelberg.
Au Royaume-Uni et en Allemagne d'intenses efforts ont été faits depuis 1993.
La France a pris conscience du problème plus récemment. En 1995, a été créé un
Groupement d'Intérêt Scientifique " Informatique appliquée à l'étude
des
biomolécules et des génomes " (INFOBIOGEN), à l'initiative du
ministère de
l'enseignement supérieur et de la recherche, et de l'Association française
contre les
myopathies.
OBJECTIFS
Les missions de INFOBIOGEN sont :
1.
de développer des activités de transfert et de service visant
à :
assurer la production, la maintenance, la normalisation, la mise en ligne des
logiciels et
banques de données thématiques produits par l'ensemble des équipes françaises
travaillant dans le domaine des génomes et de la structure des molécules ;
permettre à l'ensemble de la communauté scientifique française un accès
interactif aux
bases de données, en particulier en développant la connexion des utilisateurs
aux
réseaux nationaux et internationaux et en facilitant l'accès à des données
biologiques
actualisées (accès aux séquences quotidiennes de l'EMBL et des autres grandes
bases de
données internationales) ;
fournir une assistance informatique aux équipes françaises dans leurs projets de
cartographie, d'analyse de séquences et des macromolécules biologiques ;
mettre à leur disposition des programmes d'analyse et de recherche rapide de
similitudes
dans les séquences de biomolécules et de génomes.
2.
d'exercer des actions de formation
;
3.
de coordonner le réseau national des activités de bio-informatique et
de
participer à la concertation et à la coordination nationale et
internationale ;
4.
De favoriser la valorisation des logiciels et des banques de données
biologiques produites en France ;
5.
d'avoir une activité de recherche propre comportant en particulier la
création
de logiciels d'analyse des génomes et de biomolécules, la recherche sur les
modalités
de mise en relation de bases de données et le développement d'outils
d'acquisition de
données, de stockage et d'analyse ;
6.
de proposer des méthodes d'expertise et d'évaluation dans le domaine
de la
bio-informatique.
PARTENAIRES DU GIS INFOBIOGEN
Quatre EPST :
le CNRS (Centre national de la recherche scientifique) ,
l'INSERM (Institut national de la santé et de la recherche médicale) ,
l'INRA (Institut national de la recherche agronomique) ,
l'INRIA (Institut national de la recherche informatique et automatique).
Quatre universités parisiennes :
Université de Paris V ;
Université de Paris VI ;
Université de Paris VII ;
Université de Paris XI.
Généthon avec le soutien de l'AFM (Association française contre les
myopathies)
le MENRT (Ministère de l'éducation nationale, de la recherche et de la
technologie)
(91( * ))
Le GIS
INFOBIOGEN a été doté de 15 millions de francs en 1999 au titre des actions
prioritaires pour les sciences du vivant définies par le Comité
interministériel de la
recherche scientifique et technologique (CIRST).
Cette impulsion doit être complétée par des actions tendant à faire
connaître aux
informaticiens français l'intérêt de la bio-informatique et à leur
offrir, dans
un premier temps des formations courtes en biologie
. INFOBIOGEN pourrait,
par
l'intermédiaire d'Internet, envoyer des messages aux écoles, IUT et facultés qui
enseignent l'informatique ainsi qu'aux agences de l'emploi (afin, notamment, de
réorienter après janvier 2000 les informaticiens dont la tâche aura été de
gérer le bogue de l'an 2000). Cette activité d'information doit être
accompagnée d'un
effort du système éducatif pour mettre en place des sessions courtes
d'initiation à la
biologie.
Compte tenu de la demande et de l'urgence, ces formations pourraient être
partiellement
financées par les industries pharmaceutiques et biotechnologiques.
- Les actions à plus long terme
La formation
d'experts en traitement de l'information génomique doit devenir l'une des
priorités de
l'enseignement supérieur, cycle long et filières professionnalisées confondus.
Deux
types d'action peuvent être parallèlement menés :
permettre aux scientifiques d'acquérir des compétences informatiques
de bon
niveau
. Ainsi, l'Institut de formation supérieure en informatique et
communication de
l'Université de Rennes propose un DESS de compétence complémentaire en
informatique qui
accueille des scientifiques de tous horizons ;
proposer un module de formation " biologie
moléculaire "
dans tous les enseignements d'informatique
: IUT, universités, écoles
d'ingénieur, afin que les informaticiens soient en mesure de dialoguer avec les
biologistes pour mieux adapter leurs programmes aux recherches menées.
L'essentiel est en effet de faciliter les contacts entre deux mondes très
différents
mais qui sont devenus complémentaires : le domaine du vivant et celui de
l'ordinateur.
2.3.1.4. La formation des citoyens
Tous les
Français doivent pouvoir acquérir un minimum de connaissances sur la génétique
et ses
diverses applications, tant médicales qu'agro-alimentaires.
une " école de l'ADN " :
des initiatives
de ce type
ont été prise outre-Atlantique il y a plus de dix ans. La plus célèbre, le
" DNA Learning Center " de Cold Spring Harbor (New-York) a
eu plus de
150 000 visiteurs en dix ans et accueille 5 000 stagiaires
par an.
À Nîmes, une école de l'ADN a été créée tout récemment, dans les murs du Muséum
d'Histoire naturelle, à l'initiative de Philippe BERTA, professeur de biologie
(détaché
de l'INSERM) à l'université de Montpellier-Nîmes. Elle s'est dans un premier
temps
consacrée à un public de jeunes de l'enseignement secondaire ou supérieur en
proposant,
dans quatre ateliers de niveaux différents, de découvrir l'ADN dans le cadre du
laboratoire, avec des expériences génétiques.
Cette école de l'ADN a connu un tel succès qu'elle envisage, outre sa fonction
d'information, de développer des activités de formation permanente (pour des
juristes,
des policiers, des industriels de l'agro-alimentaire) et de devenir un lieu de
débat
(bioéthique, aliments transgéniques...).
Une seconde école de l'ADN pourrait, par exemple, être créée au sein de la
Cité
des Sciences et de l'Industrie, dans le cadre du programme " les
défis du
vivant " qu'elle va lancer au printemps 2001
.
Ne serait-il pas pertinent de créer
un parc à thème, ludique et
pédagogique
,
GÉNOSCOPIA
?...
2.3.2. LA MÉDECINE PRÉDICTIVE
Depuis une
dizaine d'années l'accélération de la connaissance des gènes impliqués dans les
maladies héréditaires a permis la mise au point de nombreux tests génétiques.
L'apparition de la technologie des biopuces va amplifier ce phénomène en
favorisant la
diffusion massive de tests bientôt peu coûteux et d'utilisation facile.
Les tests génétiques ont pour caractéristique de réaliser, notamment chez des
sujets
à risque, un diagnostic avant toute apparition de symptôme : on parle de
tests
prédictifs puisqu'ils permettent de prédire une maladie potentielle.
Leur multiplication risque de poser de sérieux problèmes et devra nécessairement
s'accompagner de précautions, à prendre dans trois domaines principaux.
2.3.2.1. Précautions pour l'analyse des résultats
Les réponses
fournies par les tests génétiques ne peuvent
, compte tenu notamment de la
complexité des gènes, de leur interaction et de l'influence sur eux de
l'environnement,
être
catégoriques
.
Certaines mutations génétiques décelées par des tests ont des conséquences
médicales très claires. Ainsi, la polypose adénomateuse familiale, responsable
de
1 % des cancers colorectaux, est systématiquement corrélée à une
altération du
gène APC, gène suppresseur des tumeurs, situé sur le bras long du
chromosome 5.
Cette mutation provoque toujours la formation de polypes intestinaux,
l'évolution de l'un
de ces polypes au moins vers le cancer étant inéluctable.
En revanche, de nombreux autres diagnostics sont moins clairs. En effet,
les
techniques de détection de mutation sont fiables en elles-mêmes si elles sont
pratiquées correctement. Mais, souvent, elles ne permettent pas d'examiner la
totalité
du gène : certaines mutations peuvent alors échapper à l'investigation.
Dans le
cas des formes héréditaires de prédisposition au cancer du sein (5 à 8 %
des
cancers), les deux gènes impliqués (BRCA 1 sur le chromosome 17 et
BRCA 2
sur le chromosome 13) sont très longs, et ne peuvent être, en l'état
actuel de la
science, totalement explorés. De plus l'analyse ne porte que sur la région
codante de
cette portion d'ADN et non sur les régions régulatrices du gène, dont les
lésions
éventuelles ne peuvent être détectées.
Le résultat d'un test portant sur les gènes BRCA 1 et BRCA 2 peut donc
être :
- soit négatif : aucune mutation n'est décelée mais elles ne sont pas
toutes
décelables...
- soit positif : une mutation est décelée mais elle ne signe qu'une
prédisposition
à la maladie, celle-ci pouvant ne jamais apparaître...
De nombreux autres tests ne permettent de diagnostiquer qu'une prédisposition à
la
maladie : c'est le cas, par exemple, du syndrome de Huntington ou du
syndrome de
Lynch (HNPCC :
hereditary non polyposis colon cancer
) qui concerne
2 à
5 % des cancers colorectaux et que les mutations des gènes MSH 2,
MSH 6,
MLH 1, PMS 1 ou PMS 2 peuvent éventuellement provoquer.
Enfin, il faut toujours garder à l'esprit que les gènes interagissent avec
l'environnement, qui peut faire s'exprimer ou non les prédispositions
génétiques. On
sait notamment combien le mode de vie compte dans des maladies telles que le
cancer.
2.3.2.2. Évaluation de l'utilité thérapeutique des tests
L'utilité
thérapeutique du diagnostic est avérée lorsqu'existe un traitement médical ou
chirurgical.
Cet intérêt apparaît clairement pour certaines maladies monogéniques pouvant
bénéficier d'un traitement médical simple (l'hémochromatose familiale ou la
fièvre
méditerranéenne familiale).
Dans d'autres cas une solution chirurgicale est envisageable : pour la
polypose
adénomateuse familiale dont l'évolution cancéreuse est, ainsi qu'on l'a vu,
inéluctable, une colectomie totale est préconisée avec un bon pronostic vital.
De même, le syndrome de néoplasie endocrinienne multiple, détecté grâce à une
mutations du gène RET sur le chromosome 10 et qui se traduit par des
cancers de la
thyroïde et des parathyroïdes, peut être évité par un acte chirurgical
préventif.
La recherche de mutations génétiques a également un intérêt incontestable
dans
le domaine de la prévention :
- elle évite certains dépistages astreignants et pénibles : les
enfants des
familles à risque chez qui aucune mutation du gène APC n'a été détectée ne sont
pas
soumis à des coloscopies répétées ;
- elle permet de mettre en place, chez les individus dont les mutations
génétiques
signent la prédisposition à une maladie, des examens préventifs fréquents qui
permettent de déceler une tumeur dès le début du processus de cancérisation et
d'accroître ainsi considérablement l'efficacité d'un acte chirurgical. On peut
citer le
cas du cancer du sein ou de la prostate.
Toutefois, en dehors des cas précités de polypose adénomateuse familiale ou de
néoplasie endocrinienne, il faut se garder de tout acte chirurgical préventif
non
justifié par la découverte réelle d'une tumeur à l'occasion d'un examen de
dépistage : aux États-Unis, un test commercial de détection des mutations
du gène
BRCA 1, réalisé sur simple prise de sang, est proposé par Affymetrix.
Nombreuses sont les femmes américaines qui ont passé ce test, mais, hormis la
connaissance de leur risque important de contracter ce cancer, il n'est pas
possible de
prévoir, si elles le développeront (et quand), s'il affectera le sein ou
l'ovaire, sa
gravité et sa curabilité, etc.
Ceci entraîne chez les familles considérées à risque des comportements
psychologiques
de détresse, de confusion et même une exérèse des ovaires et une mastectomie
double,
pratiquées " à titre préventif " chez des femmes en bonne
santé,
qui n'auraient peut-être jamais, malgré la présence du gène, développé de
cancer.
En revanche, dans bien d'autres cas, les tests génétiques n'ont qu'une
fonction
d'information, car aucune solution, préventive ou curative, médicale ou
chirurgicale
n'existe. Leur rôle est alors de lever le doute ou d'informer sur les risques de
transmission du handicap aux enfants.
C'est le cas de certains cancers : le syndrome de Li et Fraumeni, par
exemple, lié
à une mutation rare du gène P 53 et qui induit un risque élevé de
développement
de plusieurs tumeurs de localisation variées.
C'est également le cas de quelques handicaps monogéniques rares (certaines
surdités ou
cécités) et de maladies monogéniques relativement fréquentes : myopathies,
chorée de Huntington
92(
*
)
; dans le cas de
cette
dernière, les tests prédictifs montrent toutes leurs limites : lorsqu'une
personne
est porteuse de la mutation génétique de la chorée de Huntington, elle n'a
aucune
certitude de développer la maladie mais elle a celle, en revanche, de ne
pouvoir disposer
d'aucun traitement si la pathologie apparaît... Dans de tels cas, un test
génétique
peut avoir des conséquences psychologiques, personnelles ou familiales
catastrophiques.
Il est indispensable de veiller à ce que ces tests ne soient pas disponibles
dans
n'importe quelles conditions sur le marché et d'organiser une prise en charge
médicale
et psychologique
.
La Fédération mondiale de neurologie et l'Association Internationale Huntington
ont
émis des recommandations précises : le test ne peut être réalisé que chez
un
adulte à haut risque (frère, soeur, enfant d'un malade et éventuellement
petit-enfant,
neveu ou nièce). La demande doit être faite en toute liberté et en-dehors de
toute
pression. La personne à risque doit avoir reçu toutes les informations
nécessaires et,
avant de prendre la décision définitive de passer le test, consulter des
neurologues,
généticiens et psychologues. En cas de résultat défavorable un suivi
médico-psychologique peut être mis en place si la personne le souhaite.
2.3.2.3. Préserver le droit de ne pas savoir et celui de ne pas faire connaître
" Le
droit à " l'intimité génétique " -la
genetic
privacy
- va
être l'une des plus grosses revendications du prochain siècle ; dans
l'agenda
politique et social de la plupart des pays, elle occupera la place qui fut
celle de la
question des droits de l'homme et des droits civiques au siècle dernier. À
mesure que le
nombre de victimes de diverses formes de discrimination génétique augmentera,
les gens
vont s'organiser afin que cette information génétique soit sous leur propre
contrôle,
et non exploitée par n'importe quelle institution. "
93(
*
)
La principale précaution à prendre en médecine prédictive est de faire en sorte
qu'aucune pression ne soit exercée sur les personnes à risque pour qu'elles
réalisent
des tests génétiques, et que la diffusion des résultats des tests soit
contrôlée : elle peut avoir des répercussions sur la famille, le travail
et dans
le domaine des assurances.
Il faut bien peser les
conséquences des tests génétiques dans une
famille
.
Si le test d'un membre d'une famille à risque ne révèle pas de prédisposition à
une
maladie héréditaire, les problèmes ne sont pas tous résolus pour autant.
Certaines
études montrent que ceux qui ne sont pas porteurs d'un gène muté rencontrent
parmi les
leurs des difficultés d'un autre ordre : d'une part, la
" maladie " demeure dans la famille et, d'autre part, le
hasard
génétique qui épargne les uns et affecte les autres est mal vécu.
Si le test révèle une prédisposition à une maladie de nombreux problèmes se
posent :
" Quelles sont les conséquences pour les individus de la
connaissance qu'ils
portent un mauvais gène ? Cette connaissance est-elle de nature à rendre
le regard
accusateur sur ses parents ou sur son lignage ? À écraser le désir
d'enfant sous
un sentiment de culpabilité ou sous un désir de normativité ? Enfin, au
sein du
couple, quel peut être le potentiel destructeur de la responsabilité d'un des
parents
dans la transmission, à un enfant gravement atteint, d'un gène d'une maladie
dominante ?
Voilà quelques questions dont on peut faire
l'économie. "
94(
*
)
Enfin, l'information génétique n'est pas personnelle : une personne
indiquant à
ses proches qu'elle est porteuse d'une mutation génétique peut faire naître le
doute
dans l'esprit de ses enfants aussi bien que de ses parents. Une telle
information peut
donc avoir des répercussions graves sur les membres de la famille, à titre
personnel
mais aussi, éventuellement, à titre professionnel et dans le domaine des
assurances si
des règles de protection ne sont pas clairement définies.
On peut craindre qu'à moyen terme, lorsque l'usage des tests génétiques
sera plus
répandu, les entreprises ne pratiquent une
sélection à
l'embauche
,
éliminant de certains postes les personnes atteintes d'une prédisposition
génétique
défavorable.
Cette pratique discriminatoire peut être présentée dans un premier temps comme
un
élément de médecine préventive. En France, l'Institut national de la recherche
et de
la sécurité pour la prévention des accidents du travail et des maladies
professionnelles a consacré un rapport à " la médecine prédictive
appliquée
au travail ". Ces travaux ont été approuvés tant par le Comité
consultatif
national d'éthique que par le Conseil national du patronat français et se
situent dans
un cadre préventif précis : déterminer les bases biologiques et génétiques
de
prédisposition à certaines affections, pour éviter aux personnes qui en seraient
porteuses d'être exposées à un risque supplémentaire dans un environnement
professionnel éventuellement pathogène.
Il est impossible que les employeurs n'aient pas la tentation d'utiliser ces
tests dans
d'autres buts. Dans un souci de rentabilité, ils chercheront à améliorer
l'adéquation
entre l'employé et l'emploi et surtout lutter contre une perte de productivité
liée à
une susceptibilité anormale d'un employé à telle ou telle maladie.
Une telle dérive, socialement inadmissible, se produira inévitablement si la
loi ne fixe
pas des interdits.
Aux États-Unis, la discrimination génétique est déjà très répandue. Jeremy
RIFKIN,
dans son récent ouvrage " Le siècle biotech : le commerce des
gènes dans
le meilleur des mondes "
95(
*
)
évoque
deux
études ; la première, réalisée par L. GELLER à Harward en 1996 montre
que
la discrimination génétique est utilisée par les employeurs, les compagnies
d'assurance, les écoles et les agences d'adoption. Dans l'autre enquête, il
était
demandé à des directeurs de ressources humaines et des chefs d'entreprise s'ils
utiliseraient les tests génétiques quand ceux-ci seraient moins coûteux et plus
répandus ; beaucoup ont répondu par l'affirmative, précisant que cela leur
permettrait de mieux planifier les embauches et l'avancement...
Dans le
domaine des assurances
, les problèmes de sélections
génétiques
sont inégaux selon les pays considérés et les risques couverts.
Aux États-Unis la situation est très hétérogène. Près de la moitié des États ont
voté des lois interdisant la discrimination génétique sur le lieu de travail,
pour
l'assurance maladie ou d'autres types d'assurances. Mais, selon les États, ces
interdictions sont plus ou moins étendues et ne couvrent pas les mêmes champs.
Leur
portée est donc relative. Le Congrès américain dispose de plusieurs projets de
loi sur
la discrimination génétique mais ils n'aboutissent pas car ils se heurtent
notamment aux
intérêts des assureurs qui ont clairement indiqué :
" Nous
nous
battons pour que les assurances aient accès à toute l'information médicale
existante,
sans distinction entre le dossier médical classique et l'information
génétique "
96(
*
)
Au Japon, l'Association japonaise de l'Assurance-Vie a présenté en 1997 un
rapport sur
la nécessité de communiquer aux compagnies d'assurance-vie lors de la signature
d'un
contrat les informations génétiques de l'intéressé, si celui-ci en a
connaissance,
concernant les risques d'une maladie spécifique. Ce rapport n'a pas force
contraignante
mais reflète l'attitude des compagnies face à l'utilisation des données
génétiques.
En Europe, le Royaume-Uni occupe une place particulière. Le Gouvernement a
décidé
d'accepter les règles du jeu fixées par l'ABI (Association of British
Insurers), dans le
secteur de l'assurance-vie. Celles-ci se résument ainsi :
- quelle que soit la couverture du contrat, l'assureur ne peut obliger son
client à
se soumettre à des tests génétiques ;
- un client qui a effectué des tests génétiques a pour obligation de
communiquer
les résultats à son assureur avant la signature de tout nouveau contrat ;
- jusqu'à 100 000 livres de couverture, soit approximativement
un million
de francs, l'assureur ne peut pas utiliser les résultats ;
- au-delà de ce montant, l'assureur est libre d'en tenir compte ou
non : en cas
de prédisposition à une maladie grave, il peut augmenter la prime d'assurance
ou refuser
d'assurer son client.
Les autres pays d'Europe se classent en non-interventionnistes (Allemagne,
Italie, Espagne
et Portugal) et prohibitionnistes (Autriche, Norvège, Suède, Belgique, Pays-Bas
et
France).
En France, la Fédération française des sociétés d'assurance (FFSA) a décidé en
mars
1999, à l'unanimité de sa commission exécutive, de renouveler pour une période
de cinq
ans l'engagement pris en 1994. Les assureurs se sont donc engagés
" à ne
pas tenir compte des résultats de l'étude génétique des caractéristiques d'un
candidat à l'assurance, même si ceux-ci leur sont apportés par l'assuré
lui-même "
et à ne poser
" aucune question relative
aux tests
génétiques et à leurs résultats dans les questionnaires de
risques "
.
Ils ne demanderont pas à leurs clients de se soumettre à des tests génétiques
avant de
souscrire une assurance-vie et ne réclameront pas les résultats de tests
éventuellement
réalisés auparavant.
Pendant ce moratoire,
il est indispensable que des règles précises soient
fixées par
le législateur français, particulièrement à l'occasion de la révision de la loi
sur
la bioéthique de N° 94-654 du 29 juillet 1994
.
Il faut par ailleurs distinguer les conséquences d'une discrimination génétique
par les
assurances en fonction des risques couverts.
- Dans le domaine de
l'assurance-vie
, des assureurs peuvent avoir
la tentation
d'utiliser des informations génétiques.
Ils se protégeraient ainsi du risque " d'antisélection " ou
" sélection adverse " : si un client est protégé par le
" droit au mensonge " l'évaluation du risque par l'assureur
est
faussée. En effet, si quelqu'un, au vu des résultats d'un test génétique,
découvre
qu'il court un risque de décès prématuré, il peut, en cachant ces résultats à
l'assureur, contracter une assurance d'un montant considérable, faussant ainsi
les
calculs de l'assureur.
Cet argument est discutable car dans de nombreux cas, les prédispositions
génétiques à
une maladie ne conduisent pas, heureusement, au développement de la pathologie.
Dans ce
cas si la majorité de ceux qui ont une prédisposition prouvée prend une
assurance-vie
surdimensionnée, les assurances ne courent qu'un seul risque : celui de
voir leur
bénéfice s'accroître...
En tout état de cause, dans le domaine de l'assurance-vie, il est indispensable
d'aboutir
rapidement à la rédaction de directives européennes garantissant la
non-discrimination
génétique tout en plaçant l'ensemble des assureurs européens dans la même
situation
afin de ne pas fausser les règles de la concurrence.
C'est dans cet esprit que se situe la convention de bioéthique européenne qui
vise, à
terme, à protéger les citoyens des quarante et un États membres du Conseil de
l'Europe
contre " les applications abusives des progrès biologiques et
médicaux ". Cette convention, élaborée en 1994, entrera en vigueur le
1
er
décembre
1999 car elle vient d'être ratifiée par un cinquième pays, le Danemark, en août
1999
(la lenteur de sa ratification souligne la difficulté qu'éprouvent les pays
européens
à prendre des positions de principe communes sur les résultats des tests
génétiques
personnels et l'usage scientifique de l'embryon humain).
En ce qui concerne la génétique, cette convention interdit toute forme de
discrimination
à l'encontre d'une personne en raison de son patrimoine héréditaire et
n'autorise les
tests prédictifs de maladie génétique qu'à de strictes fins médicales.
- Dans le domaine de l'
assurance maladie
, la situation est très
différente
si l'on se place dans un système privé ou public.
Aux États-Unis la discrimination génétique pourrait avoir des conséquences
graves si
les assurances privées refusent de couvrir le risque maladie et invalidité des
personnes
dont les tests génétiques sont négatifs, laissant sans assurance ceux qui en
ont le
plus besoin.
Dans une telle situation, on peut même craindre que nombre de personnes
renoncent à
passer des tests génétiques, même s'ils sont recommandés par leur médecin,
simplement
pour ne pas risquer de perdre leur contrat d'assurance ou de payer une très
forte
surprime.
Dans les pays où la solidarité sociale est organisée par l'État, le problème est
beaucoup moins grave.
En Grande-Bretagne, la Nuffield Foundation sur la bioéthique a rappelé, en
1993, qu'il
n'y avait pas lieu de s'inquiéter tant que l'assurance santé dépendait
principalement
du secteur public.
En France, le système de sécurité sociale constitue la meilleure garantie
contre
l'exclusion de ceux qui ont des prédispositions génétiques inquiétantes. En
effet,
seule une assurance maladie universelle, non discriminante par essence,
permettra de
conserver une réelle solidarité
.
CONCLUSION ET RECOMMANDATIONS CONCLUSION ET RECOMMANDATIONS
Le sujet de ce
rapport, génomique et informatique, sous des titres et des approches comme des
présentations différentes, a fait récemment l'objet de deux publications
importantes ; l'une est due au Conseil économique et social sous le
titre
:
" La France au défi des biotechnologies : quels enjeux pour
l'avenir "
adoptée le 7 juillet 1999. L'autre a pour
origine
l'Académie des Sciences avec comme titre
" Développement et
applications
de la génomique. L'après-génome "
, présenté à la presse le
8 septembre 1999.
Il me semble que ces trois rapports se complètent plus qu'ils ne se répètent,
soit par
la volonté du Conseil économique et social d'embrasser plusieurs domaines
marqués par
les biotechnologies, de l'humain à l'agriculture en passant par l'alimentation,
avec une
attention particulière aux aspects " éthiques " de ce défi,
soit
par l'ampleur et le haut niveau scientifique de celui de l'Académie des
Sciences auxquels
les notes personnelles de participants prestigieux donnent une valeur inédite
par leur
pertinence.
Tenter d'en faire un résumé serait déplacé dans ce rapport demandé par l'Office
parlementaire d'évaluation des choix scientifiques et technologiques. En
présenter
quelques-unes des conclusions sans développer le cheminement intellectuel comme
scientifique ou technique les altéreraient trop.
Le rapport que m'a confié l'Office permet de poursuivre le but principal de
cette
délégation : mettre à la disposition des parlementaires les éléments de
connaissance et de compréhension d'évolutions prévisibles d'une société, dans
laquelle ils ont cette responsabilité spécifique qu'ils ne peuvent transférer à
nul
autre, contribuer à ce que la loi soit dite.
Les recommandations que je me permets de suggérer sont, me semble-t-il, une
façon
efficace de fournir des arguments, des éléments de réflexion et de proposition,
au
législateur.
LES RECOMMANDATIONS
|
POUR LA RECHERCHE |
||
|
I. Au niveau des structures |
||
|
|
Renforcer les partenariats public-privé en les structurant dans deux directions : |
|
|
|
- |
La collaboration des chercheurs publics avec les grands groupes pharmaceutiques ; |
|
|
- |
La collaboration des chercheurs publics avec les petites entreprises de biotechnologies (qui ont souvent elles-mêmes noué des liens avec les groupes) ; |
|
|
Le partenariat public-privé trouve tout son sens dans le processus de découverte de nouveaux médicaments : compte tenu de l'investissement (3 milliards de francs) et du temps (12 ans) nécessaire pour la mise au point d'une nouvelle molécule thérapeutique, les grands groupes pharmaceutiques , dont aucun ne représente plus de 5 % du marché mondial, procèdent actuellement à une externalisation de leur recherche (près d' un tiers du budget de recherche-développement en moyenne, contre 4 % en 1994) et ont besoin de nombreux partenaires. |
|
|
|
Valoriser la
recherche publique, inciter les chercheurs à créer
des entreprises ou à exercer des fonctions de consultant auprès des
entreprises
innovantes, ces consultations devant se situer un contexte de transparence.
Il convient,
au minimum, d'inciter les chercheurs à réfléchir aux débouchés éventuels de
leurs
résultats en terme de produits.
Utile dans de nombreux secteurs
scientifiques, cette
valorisation de la recherche publique est indispensable dans le domaine de
la génomique
.
|
|
|
|
Favoriser les réseaux composés de partenaires publics et privés (nationaux et internationaux), sur le modèle notamment du réseau Génoplante . |
|
|
|
|
|
|
II. Au niveau des orientations |
||
|
|
Affiner la
connaissance de l'ADN (zones non codantes, épissage,
régulation génétique, microsatellites...) et des protéines (la
protéomique
) ;
développer la recherche en
biologie structurale
;
|
|
|
|
Favoriser la recherche et le développement des biopuces en France dans deux directions : la synthèse in situ et la détection des hybrides. |
|
|
|
|
|
|
POUR L'INDUSTRIE |
||
|
I. Le tissu industriel |
||
|
|
Aider les start-up de biotechnologie en intensifiant les efforts réalisés depuis 1998 dans le domaine du capital-risque et en multipliant les bio-incubateurs ; |
|
|
|
Favoriser le développement des biopôles, organiser la sélection des meilleurs et leur coopération . Dresser un inventaire exhaustif des atouts de chaque région dans le domaine de la génomique en recourant systématiquement à des experts internationaux afin que la labellisation " biopôle " soit reconnue à l'étranger. |
|
|
|
|
|
|
II. Les brevets |
||
|
|
Mettre en place à l'Institut national de la propriété industrielle une cellule de réflexion, à laquelle participeraient des responsables des entreprises de génomique, afin d' étudier les conséquences de la création du SNP 97( * ) Consortium en avril 1999 et de réorienter éventuellement la stratégie de brevetabilité de l'information génétique . |
|
|
|
|
|
|
POUR LA SOCIÉTÉ |
||
|
I. La formation professionnelle |
||
|
|
Les biologistes : |
|
|
|
- |
développer l'enseignement de la biologie du XXIe siècle ; |
|
|
- |
Encourager l'enseignement de cette discipline dans les universités situées près des biopôles ; instituer une option " informatique " dans tous les cursus de biologie ; créer des écoles doctorales proposant des spécialisation en génomique, biotechnologie et bio-informatique. |
|
|
|
|
|
|
Les médecins et les pharmaciens : |
|
|
|
- |
Enseigner la génomique dès le deuxième cycle ; rapprocher le DES de génétique (médecins) et le DES de biologie médicale (pharmaciens) en instituant des enseignements communs ; |
|
|
- |
Créer un troisième cycle de recherche sur le médicament, ouvert aux pharmaciens, aux médecins et aux biologistes ; |
|
|
- |
Proposer des " sessions de génomique " aux médecins et pharmaciens dans le cadre de la formation continue. |
|
|
|
|
|
|
Les bio-informaticiens : |
|
|
|
- |
Essayer de
combler le
besoin urgent de bio-informaticiens
en
faisant connaître aux informaticiens français l'intérêt de la
bio-informatique et en
leur offrant dans un premier temps des
formations courtes en
biologie
; à
plus long terme, permettre aux scientifiques d'acquérir des compétences
informatiques de
bon niveau et de proposer un modèle de formation " biologie
moléculaire " dans les enseignements d'informatique afin de
faciliter le
dialogue entre le domaine du vivant et celui de l'ordinateur.
|
|
|
|
|
|
|
Les citoyens |
|
|
|
|
Créer une seconde école de l'ADN , par exemple à la Cité des Sciences et de l'Industrie ; |
|
|
|
Créer un parc à thème ludique et pédagogique, " GÉNOSCOPIA "... ? |
|
|
|
|
|
|
|
|
|
II. Dans le domaine de la médecine prédictive |
||
|
|
Veiller à ce que les tests de diagnostic génétique ne soient pas disponibles dans n'importe quelles conditions et organiser une prise en charge médicale et psychologique des individus ; |
|
|
|
Préserver le
droit de ne pas savoir et celui de ne pas faire
connaître
:
|
|
|
|
|
|
* *
*
Les progrès de
la biologie modifient et enrichissent notre compréhension des mécanismes de la
vie ainsi
que les modes de production d'un grand nombre de domaines : agriculture,
élevage,
agro-alimentaire, environnement et santé. Les domaines de la santé humaine et
de la
médecine sont ceux qui bénéficient le plus rapidement et profondément des
apports
scientifiques et technologiques nouveaux.
La France doit prendre en compte cet état de fait et mobiliser ses moyens
pour que sa
contribution à l'enrichissement de la connaissance soit majeure et pour
bénéficier
aussi, à travers ses industries, du fruit de ces progrès technologiques.
L'origine des progrès dans les sciences de la vie et de la santé humaine tient
à la
maîtrise des connaissances de la génétique et de ses outils que sont la
génomique, la
protéomique, la bio-informatique. Dans le domaine de la recherche médicale,
celui de la
recherche des thérapeutiques médicamenteuses tire un plein avantage de la
maîtrise de
ces outils. La France, pour des raisons qui tiennent à une perception tardive de
l'importance des enjeux, à un affaiblissement notoire de son industrie
pharmaceutique, à
des efforts insuffisants de formation des chercheurs dans ce domaine, doit
maintenant
faire l'effort de revenir dans le jeu.
Plusieurs rapports sont produits sur l'importance du sujet et toutes les
recommandations
convergent pour inciter les responsables politiques à mobiliser des moyens
massifs sur
les programmes de recherche, le transfert des connaissances, l'incitation à la
création
d'activités nouvelles dans le domaine des biotechnologies et plus
particulièrement dans
celui des technologies médicales et du médicament.
Elle doit se lancer sans
plus
attendre dans la phase post-génomique : la protéomique.
La France a su, dans son histoire, mobiliser ses moyens pour participer
pleinement aux
enjeux scientifiques, techniques et industriels que représentaient, au cours de
ce
siècle, l'énergie et la vitesse. Les grands programmes de transport, de
maîtrise de
l'énergie, de l'aéronautique et de l'aérospatiale ont montré que la recherche,
l'industrie et le pouvoir politique savaient, quand il le fallait, se mobiliser
sur les
grands enjeux. Ceux des sciences de la vie sont de même importance. Au sein de
ce vaste
domaine, ceux de la santé et de la thérapeutique vont occuper une place majeure
dans nos
sociétés. Les responsables politiques, à tous les niveaux, doivent faire
l'effort de
compréhension du domaine et se mobiliser pour doter notre pays des moyens de la
recherche
en génomique, en protéomique et en bio-informatique. C'est un domaine
prioritaire. La
formation des étudiants, des enseignants et des chercheurs doit prendre cette
priorité
et cette urgence en compte.
EXAMEN DU RAPPORT PAR L'OFFICE EXAMEN DU RAPPORT PAR L'OFFICE
L'Office
parlementaire d'évaluation des choix scientifiques et technologiques, s'est
réuni le
mercredi 13 octobre 1999 pour examiner le rapport de M. Franck
SÉRUSCLAT,
sénateur.
M. Henri REVOL, président, sénateur, a remercié le rapporteur pour la
qualité des
informations fournies aux membres de l'Office sur la génomique et les
biotechnologies,
domaines qui méritent qu'on leur porte un vif intérêt tant pour les
partenariats qu'ils
induisent entre la recherche publique et la recherche privée, que pour les
créations
d'entreprises qu'ils peuvent susciter.
M. Jean-Yves LE DÉAUT, premier vice-président, député, a souligné les
liens
qui existent désormais entre la génomique et l'informatique. Il a rappelé les
priorités de la recherche française, fixées par le Comité interministériel de la
recherche scientifique et technologique (CIRST) dans un sens favorable aux
sciences du
vivant. Il a insisté sur la nécessité de maintenir les efforts budgétaires en
faveur
de la génomique et de la post-génomique. Rappelant l'excellence des travaux de
l'Institut national de recherche en informatique et en automatique (INRIA), il
a exprimé
le souhait que, dans un biopôle au moins, la collaboration entre la biologie et
l'informatique soit le résultat d'une démarche des informaticiens en direction
des
biologistes et non l'inverse.
Mme Michèle RIVASI, députée, a évoqué l'opportunité d'une
pluridisciplinarité
plus ouverte, ne se limitant pas à une coopération des biologistes et des
informaticiens. Elle a mis en lumière l'urgente nécessité de prendre des mesures
limitant l'accès aux résultats des tests génétiques, afin notamment que les
assureurs
ou les employeurs ne puissent pas en avoir connaissance.
À l'issue du débat, le rapport a été adopté à l'unanimité et sa publication
autorisée.
" RÉFLEXIONS POUR L'AVENIR : CONSULTATION DANS UN CABINET
MÉDICAL EN
2010 "
RÉFLEXIONS POUR
L'AVENIR :
CONSULTATION DANS UN CABINET MÉDICAL EN 2010
Faut-il proposer une charte " éthique " ?
Le recours à des médicaments génomiques entraîne-t-il des comportements
nouveaux du
médecin envers le malade ? Faut-il proposer une charte
" éthique ", sous réserve de préciser le sens de ce terme,
au
prétexte d'usage de copies conformes, chimiques et inertes, des composants du
génome
humain ?
Dans la mesure où ils ne sont que des produits chimiques corrigeant des
déviances
génétiques, comblant l'absence totale ou partielle d'un gène manquant,
chimiquement
déterminé, le médecin devrait-il avoir un comportement particulier envers celui
auquel
il apporte ses soins ? Ce médicament génétique a comme objet d'éviter la
survenue
ou d'effacer les effets d'un trouble génique détecté par comparaison entre un
génome
normal obtenu par décryptage portant sur les cellules somatiques à l'exception
des
cellules germinales, autrement dit ne corrigeant que les causes d'une maladie
vécue sans
intention d'en protéger une descendance ?
Mais peut-on être assuré de n'intervenir que sur eux, de maîtriser le parcours
du virus
vecteur, d'éviter une déviance aboutissant à un chromosome terminal ?
Cette hypothèse contraint-elle à une charte différente du serment
d'Hippocrate ?
Le respect du code de bonne conduite et la notion de responsabilité sans cause
ne
sont-ils pas les garants d'un juste exercice professionnel comme du respect des
autres
commandements en vigueur ? Les pratiques thérapeutiques comme les actes
chirurgicaux
ne sont-elles pas sujets à risques ?
L'objectif de ce rapport n'était pas la prise en compte de ces questions :
éventuellement, elles pourraient être le sujet d'un nouveau rapport consacré aux
" Sciences de la Vie et Droits de l'Homme ". Il trouverait
une partie
de son sujet dans des réponses à apporter aux préoccupations du Comité national
d'éthique qui souhaite que soient clairement distingués :
" - les diagnostics présymptomatiques qui permettent de mettre en
évidence
l'existence d'une anormalité génétique avant les manifestations cliniques qui
peuvent
en résulter ; il s'agit souvent d'une maladie monogénique ; dans
certains cas,
une action préventive et curative peut être proposée ; dans d'autres pas
(maladie
d'Huntington) ;
- les diagnostics génétiques qui ont pour objet d'évaluer le risque pour la
descendance
de l'individu testé, soit dans le cas d'une famille " à
risques "
[...], soit dans le cas d'une population plus particulièrement disposée à une
affection
déterminée ;
- les diagnostics probabilistes de prédisposition à une maladie grave qui
ont pour
objectif d'évaluer chez un individu le risque de survenue de l'affection en
comparaison
de ce risque dans la population générale "
98(
*
)
.
Lors de la conférence mondiale sur la science, à Budapest, du 26 juin au
1
er
juillet
1999, les interventions de G. KURUDJIAN, directeur de la division de
l'éthique des
sciences et des technologies comme de F. MAYOR, directeur général de
l'UNESCO, ont
donné le sentiment que le respect de la déontologie entourant les comportements
professionnels, y compris demain le recours à la thérapie génique, protège
efficacement et le médecin et le malade lors de leur rencontre professionnelle.
Au cours
de cette conférence, et notamment du débat sur
" le possible et
l'acceptable -la science face à l'éthique ",
les risques majeurs
soulignés par les intervenants ne paraissent pas devoir entrer dans une pratique
ordinaire de la vie médicale, sauf peut-être :
- les possibilités d'utilisation abusive de l'information génétique sur les
individus que peut détenir un médecin ;
- l'acceptation de la modification thérapeutique des lignées
germinales ;
- la connaissance du gène de l'origine d'une maladie permettant aux
médecins de
pratiquer le dépistage chez le foetus ou chez l'enfant ou l'adulte, qui crée des
possibilités, donc des responsabilités nouvelles.
Mais ces réponses font partie des engagements normaux de tout médecin et
n'appellent pas
une charte " éthique " particulière.
Des risques nouveaux
Le recours à cette thérapeutique génétique n'est pas exempte de surprises,
voire même
de risques, auxquels le médecin devra prêter attention :
- des effets secondaires, comme en thérapie ordinaire, peuvent se
manifester ;
le médicament étant une substance à action physiologique ce risque devrait être
plus
rare et moins spectaculaire ;
- une parfaite connaissance des effets d'un gène sera difficile à
acquérir. Le
gène responsable d'une maladie et identifié comme tel, peut, par combinaison
d'effets
avec d'autres gènes, avoir un effet protecteur envers d'autres maladies. Ainsi,
la
drépanocytose s'accompagne d'une protection contre le paludisme et un diabète
non
insulino-dépendant s'accompagne d'une résistance aux famines séculaires.
Donc, si on répare un gène, il faudra surveiller l'apparition d'effets,
différents de
ceux provoqués par la chimiothérapie conventionnelle où cette surveillance
n'est pas
nécessaire :
- l'incapacité d'être certain que le vecteur traitant ne touchera que les
cellules
cibles laisse possible des modifications de gène dans toutes les cellules y
compris les
cellules gonosomiques (c'est-à-dire porteuses de spermatozoïdes ou d'ovocytes)
et de
transmettre la mutation à l'ensemble de l'organisme ou même à la descendance.
Ces
mutations ont des effets inconnus : aucun effet ou effet tumoral voire
même de
mutation de l'espèce.
Un exemple : agir sur les blastes serait faire bénéficier d'un traitement
plus
efficace contre la mucoviscidose et plus définitif, mais les blastes, cellules à
division rapide, peuvent se révéler oncogènes ; intervenir sur les cellules
gonosomiques des parents pour éradiquer la mucoviscidose sera une tentation,
mais sans
certitude de ne pas provoquer des mutations inattendues.
- À l'étape zygotique (jusqu'à la morula par exemple), un diagnostic
génétique
est possible, les tentatives d'intervention pour corriger peuvent donner des
mutations
viables pas forcément décelables et n'étant pas sûr de leur devenir, avec le
risque de
créer des mutants viables donnant naissance à un être humain ayant un génome
anormal.
- L'homme est capable de créer des lignées de souris hypersécrétantes en
hormone
de croissance qui leur donne des muscles sans commune mesure avec les souris
ordinaire. On
peut envisager de faire de même sur l'être humain pour créer des
" super-sportifs ".
Ces diverses perspectives, et bien d'autres, invitent à confier la surveillance
de la
thérapie génique à des pharmacologues-cliniciens, mais aussi à des
généticiens ; on ne doit pas compter sur une simple surveillance clinique
et
biochimique, elle doit être aussi morphologique et de l'ADN. On ne peut se
contenter de
l'observation des individus traités : il faut s'intéresser à leur
descendance.
Le matériel technique en 2010 :
Depuis plus de 150 ans, le médecin utilise la seringue de Pravaz, depuis
moins
longtemps le stéthoscope a remplacé l'écoute à l'oreille collée sur une
serviette
blanche dans le silence de l'entourage, les appareils de radio pour scopies
pulmonaires
plus ou moins douteuses ont totalement disparu après avoir fait quelques
ravages parmi
les médecins utilisateurs et parfois les malades examinés. Les classiques
indications
des analyses du sang avec leurs kyrielles de mises en évidence et dosages
dépassant
depuis longtemps les banales glycémies, urémies ou numérations globulaires et
autres
signes caractéristiques de maladies restent utiles bien que moins nécessaires.
Les développements des techniques modernes ont, au cours des années, contribué
au
perfectionnement du diagnostic : internet, intranet, lecteurs de CD-ROM et
de
disquette, analyse sur goutte de sang ou d'urine, ordinateur et logiciels
d'aide à la
décision. Le praticien dispose d'aides techniques qui quelquefois lui font
oublier de
prendre le temps d'examiner son malade ; il fait, trop vite, recours et
confiance à
l'ECSG, l'échographie, le scanner, la RMN...
Depuis quelques années, les lecteurs de génomes ou parties de génomes sont
entrés dans
tous les cabinets médicaux : ils permettent la découverte de la moindre
altération, voire de l'absence totale de certains gènes. La lecture nécessite
une
formation plus délicate, plus élaborée que celle des radioscopies classiques
toujours
utiles. Des laboratoires spécialisés en analyse génétique permettent une étude
détaillée des risques décelables, étude suggérée par le médecin traitant
demandeur.
Des interrogations angoissantes
La connaissance de chaque gène, c'est-à-dire ce qu'il produit et ce qu'il
régit, devra
faire partie de celle du médecin ; pourra-t-il, seul, reconnaître sur un
gène la
déficience d'une protéine, ou toutes autres causes d'une maladie ou devra-t-il
demander
une analyse à des laboratoires spécialisés comme il le fait déjà pour les
composantes
chimiques du sang ? Après avoir appris à lire une radioscopie, une
radiographie,
pourra-t-il lire une cartographie du génome humain ? Des spécialistes pour
chaque
chromosome, capables de déceler les causes d'une déficience seront-il les
recours ?
" Lorsque les chercheurs auront trouvé et décodé
(" séquencé ") le gène responsable de telle maladie et ce
qui
l'active ou la désactive, les médecins devraient être en mesure de traiter la
maladie
en remplaçant certaines des cellules " défectueuses " du
malade par
d'autres dans lesquelles le gène visé sera sain... Encore embryonnaire, la
thérapie
génique est coûteuse et ne réussit pas toujours. Les gènes modifiés ne sont pas
transmis aux enfants
du patient. Cependant la modification des gènes
dans les
ovules et les spermatozoïdes (cellules germinales) d'un couple pourrait éviter
à leurs
enfants d'être atteints d'une maladie héréditaire dont l'un des parents ou les
deux
seraient porteurs. Cette manipulation des cellules terminales est actuellement
interdite
chez l'homme dans la plupart des pays... L'acceptation de la modification
thérapeutique
germinale chez les êtres humains n'est-elle qu'une question de temps après une
période
tabou initiale, comme ce fut le cas pour la fécondation
in vitro
?
La connaissance du gène à l'origine d'une maladie permet aux médecins de
pratiquer le
dépistage chez le foetus -ou chez l'enfant ou l'adulte- pour déterminer si
celui-ci en a
hérité. Mais quelle mesure faut-il alors prendre ? Certains maladies
héréditaires
ne se manifestent pas avant l'âge mûr. D'autres comme la mucoviscidose, ont des
effets
dès la naissance. Faut-il interrompre la grossesse ? Dans la négative,
faut-il dire
à l'enfant qu'il est atteindre de la maladie même si celle-ci est
incurable ? Les
compagnies d'assurance ou les employeurs doivent-ils avoir accès à l'information
génétique de leurs clients ou de leurs salariés ? Si l'interruption de
grossesse
paraît justifiée en cas de mucoviscidose, qu'en est-il de l'hémophilie ?
Et que
faut-il faire si une maladie héréditaire est liée à une caractéristique
désirable
telle que la créativité artistique ? Qui décide de ces questions ? Où
faut-il tracer la limite et quelles garanties faut-il mettre en
place ? "
99(
*
)
On peut supposer qu'une consultation hospitalière de cardiologie se déroulerait
ainsi en
2005-2010
100(
*
)
:
Jacques, 30 ans, dont le père est décédé brutalement à l'âge de
38 ans,
décide d'aller à la consultation hospitalière spécialisée, lui a-t-on dit, dans
la
recherche des causes de mort subite.
Depuis une dizaine d'année la cardiologie a bénéficié des progrès de la
génétique
moléculaire permettant la compréhension des bases moléculaires comme
l'identification
des gènes morbides impliqués dans les arythmies et cardiopathies héréditaires et
susceptibles d'être à l'origine de mort subite. Des consignes de vie et même une
thérapeutique peuvent être envisagées. L'interrogatoire médical de Jacques
suggère
l'hypothèse d'un syndrome de QTnt-long congénital dans lequel 3 gènes
codant des
canaux ioniques sont impliqués :
KvLQT 1 (gène majeur) HERG et SCN 5A, respectivement logés sur les
chromosomes 11, 7 et 3.
Un génotypage en cardiologie mérite d'être envisagé pour vérifier s'il présente
des
anomalies à l'ECG et/ou à l'échographie cardiaque et/ou des épisodes de
syncope.
L'avancée des connaissances et surtout le développement des biopuces
permettent, dans le
cadre de cette consultation cardiologique, de définir le statut de Jacques en
terme,
notamment, de risques de mort subite et même de risques d'accidents
coronariens.
Jacques ayant donné son consentement, l'analyse génétique sera réalisée dans le
cadre
d'un bilan de départ, devant tout signe clinique évocateur.
Un prélèvement de sang est effectué et envoyé au laboratoire hospitalier
d'analyses
génétiques pour en extraire l'ADN suivi de l'étude d'une batterie de gènes
identifiés
comme responsables ou modulateurs d'une pathologie héréditaire cardiaque. Une ou
plusieurs biopuces permettront l'étude des gènes impliqués dans la myocardite
hypertrophique. L'analyse, entièrement robotisée, demandera moins d'une heure,
alors
qu'avant il fallait plusieurs semaines ; Jacques peut revenir dans
48 heures. Il
quitte le service avec un peu d'angoisse.
Les résultats, sans être tout à fait rassurants, ne sont cependant pas trop
alarmants.
Le gène majeur KvLQT 1 est normal ; les deux autres, HERG et
SCN 5A
présentant des altérations, et avec les données révélées par l'étude familiale,
invitent à la mise en oeuvre d'une thérapeutique atténuée, accompagnée d'un
suivi
médical régulier. Une prescription adaptée de bêtabloquants, dont l'effet
bénéfique
est connu depuis 1991, sera le seul traitement préventif.
Une visite médicale en 2010
a été récemment décrite dans une revue
scientifique américaine
101(
*
)
" John, un diplômé de l'enseignement secondaire âgé de
23 ans, [...]
est en bonne santé mais fume un paquet de cigarettes par jour depuis six ans.
Assisté
d'un programme informatique interactif qui prend en compte les antécédents
familiaux de
John, son médecin note qu'il existe, du côté paternel, d'importants antécédents
d'infarctus du myocarde et que le père de John est décédé à l'âge de
48 ans.
Pour obtenir des informations plus précises concernant ses propres risques de
contracter
une maladie coronarienne et autres maladies dans le
futur, John accepte
d'envisager
une série de tests génétiques disponibles en 2010.
Après avoir consulté un programme informatique interactif qui explique les
bénéfices
et les risques de tels tests, John accepte (et signe la déclaration de
consentement
éclairé) de subir 15 tests génétiques qui fournissent des informations sur
le
risque de maladies pour lesquelles il existe des stratégies préventives. Il
s'oppose,
par contre, aux 10 tests supplémentaires concernant des troubles pour
lesquels
aucune intervention préventive, validée cliniquement, n'est encore disponible.
Un prélèvement d'ADN réalisé au niveau de la joue à l'aide d'un écouvillon, est
envoyé pour analyse et les résultats sont retournés dans un délai d'une
semaine.
|
RÉSULTATS DES TESTS GÉNÉTIQUES CHEZ UN PATIENT HYPOTHÉTIQUE, EN 2010 |
|||||
|
Condition |
Gènes concernés* |
Risque
|
Risque pendant la
|
||
|
Risque
réduit :
|
HPC1, HPC2,
HPC3
|
0.4
|
7
|
||
|
Risque
élevé :
|
APOB, CETP
|
2.5
|
70
|
||
|
* |
HPC1, HPC2, HPC3 |
sont les trois gènes pour le cancer de la prostate héréditaire. |
|||
|
|
APOE |
est le gène pour l'apoliproprotéine E. |
|||
|
|
FAD3 et XAD |
sont des gènes hypothétiques pour la démence d'Alzheimer familiale. |
|||
|
|
APOB |
est le gène pour l'apolipoprotéine B |
|||
|
|
CETP |
est le gène pour la protéine de transfert des esters du cholestérol. |
|||
|
|
FCC4 |
est le gène hypothétique pour le cancer du colon familial. |
|||
|
|
APC |
est le gène pour polypose adénomateuse colique. |
|||
|
|
NAT2 |
est le gène pour le N-acétyltransférase 2. |
|||
La
consultation suivante avec le médecin et une infirmière spécialisée en génétique
porte sur les conditions pour lesquelles le risque de John diffère
considérablement (par
un facteur de plus de deux) par rapport à la population générale. Comme la
plupart des
patients, ce dernier est intéressé, à la fois, par son risque relatif et son
risque
absolu.
John est heureux d'apprendre que les tests génétiques ne donnent pas toujours de
mauvaises nouvelles ; ses risques de développer un cancer de la prostate
ou une
maladie d'Alzheimer sont réduits étant donné qu'il porte des variantes à faible
risque
de plusieurs gènes dont on connaît le rôle dans ces maladies en 2010.
Cependant, John est inquiet quant aux arguments en faveur de ses risques accrus
de
contracter une maladie coronarienne, un cancer du colon et un cancer des
poumons.
Confronté à la réalité de ses propres données génétiques, il arrive à ce moment
crucial où il comprend qu'un changement dans son comportement en matière de
santé, tout
au long de sa vie et basé sur la réduction des risques spécifiques, est
possible. Et il
y a beaucoup à offrir. D'ici à l'an 2010, le domaine de la pharmacogénomique se
sera
développé et un traitement médicamenteux prophylactique basé sur la
connaissance de
John sur ses propres données génétiques peut être prescrit de façon précise
permettant de réduire son taux de cholestérol et son risque de maladie
coronarienne à
des niveaux acceptables. Son risque de cancer du colon peut être traité en
démarrant un
programme basé sur une coloscopie annuelle à partir de l'âge de 45 ans, ce
qui est
dans son cas une manière rentable de prévenir un cancer du colon. Le risque
important de
développer un cancer des
poumons lui apporte la motivation principale de
rejoindre
un groupe de soutien composé de personnes exposées à un risque génétique élevé
de
complications graves dues au tabagisme et il arrête de fumer avec succès.
Les perspectives d'une médecine préventive individualisée basée sur la génétique
sont particulièrement passionnantes, car elle pourrait apporter une profonde
contribution
à la santé humaine.
Cependant, pour réaliser un tel projet, des protections efficaces contre le
mauvais usage
des informations génétiques doivent être en place. Par ailleurs, un autre
challenge
critique, sera qu'un nombre plus important de médecins, infirmières et autres
dispensateurs de soins devront bien connaître le domaine émergent de la médecine
génétique. "
Toutefois, l'utilisation des tests génétiques mérite réflexion et
précaution.
|
" - |
les tests génétiques ne sont et ne seront qu'une arme diagnostique de plus. Ils devront être utilisés en fonction des demandes des patients et des possibilités thérapeutiques ; |
|
- |
les tests génétiques posent et poseront des problèmes sociaux et éthiques majeurs (qui ne sont pas tous forcément nouveaux mais qui prennent là une sérieuse ampleur) ; |
|
- |
les tests génétiques seront d'autant plus mal utilisés qu'il y aura moins de politique de santé publique et d'éducation sanitaire. " 102( * ) |
GLOSSAIRE GLOSSAIRE
|
Acides aminés : |
Molécules constituant les
protéines. Il en existe vingt (Alanine,
Arginine, Asparagine, Acide aspartique, Cystéine, Acide glutamique,
Glutamine, Glycine,
Histidine, Isoleucine, Leucine, Lysine, Méthionine, Phénylalanine, Proline,
Sérine,
Thréonine, Tryptophane, Tyrosine, Valine).
|
|
Acides nucléiques : |
Macromolécules biologiques, supports de l'information héréditaire, ils portent les gènes. Il en existe deux types selon la nature du sucre qui rendre dans leur constitution :l'ADN (constituant des chromosomes) et l'ARN. Ils sont caractérisés par la succession -séquence- de nucléotides.
|
|
ADN (acide désoxyribonucléique) : |
Molécule enroulée et
repliée sur elle-même, composant des chromosomes.
Elle est composée de deux brins complémentaires, en vrille l'un autour de
l'autre.
Chaque brin est une chaîne de nucléotides. Brique élémentaire de l'ADN, un
nucléotide
est composé de trois molécules : un sucre simple, un groupement
phosphate et une
des quatre bases azotées que sont l'adénine, la guanine, la cytosine et la
thymine (A,
G, C et T).
|
|
Anticorps : |
Complexe de protéines produites par certaines cellules du sang (globules blancs) et dont la double fonction consiste, d'une part, à reconnaître et à fixer toute molécule " antigène " produite par un corps étranger, d'autre part, à activer d'autres cellules participant à la défense de l'organisme.
|
|
ARN (acide ribonucléique) : |
Molécule très proche de l'ADN mais contenant le plus souvent un seul brin, formée d'un squelette phosphate et sucre ribose, le long duquel sont attachées des bases -adénine, ou cytosine, ou guanine, ou uracile- en séquence linéaire.
|
|
ARNm (ARN messager) : |
Molécule d'ARN dont le rôle consiste à transmettre la séquence des bases d'un brin de molécule ADN, donc son code génétique, à la machinerie cellulaire qui fabrique les protéines.
|
|
Apoptose : |
Dite aussi " mort programmée de la cellule ". Elle correspond à une sorte de mort douce de la cellule par implosion, qui ne cause pas de dommages à son environnement, contrairement à la nécrose, mort violente par explosion de la cellule. Le dérèglement de l'apoptose peut conduire à l'immortalisation des cellules normalement destinées à mourir, induisant ainsi la formation de tumeurs cancéreuses.
|
|
Base : |
Molécule chimique azotée qui rentre dans la composition des nucléotides de l'ADN (adénine, guanine, cytosine et thymine) ou des ribonucléotides de l'ARN (adénine, guanine, cytosine et uracile).
|
|
Chromosomes : |
Structures visibles lors de la division cellulaire, les chromosomes portent et transmettent les caractères héréditaires. Composés d'ADN et de protéines, ils portent les gènes. Chez les organismes eucaryotes (être vivants dont les cellules sont pourvues de noyaux), ils sont présents dans le noyau des cellules sous forme de paires homologues, chaque chromosome existant en deux exemplaires. L'espèce humaine en possède 23 paires par cellules. Les cellules sexuelles ne contiennent qu'un exemplaire de chaque paire.
|
|
Gènes : |
Segments d'ADN portés par les chromosomes, ils conservent et transmettent les caractères héréditaires. Un gène est un élément d'information ; caractérisé par l'ordre dans lequel s'enchaînent les bases nucléiques, contenant les instructions nécessaires à la production dans la cellule d'un type précis de protéine. On dit qu'un gène " code " (pour) une protéine. Chez l'homme on évalue à près de 100 000 le nombre de gènes.
|
|
Génome : |
Ensemble des gènes d'un organisme. Le génome d'une cellule est formé de tout l'ADN qu'elle contient.
|
|
Molécule : |
La plus petite partie d'une substance chimique qui peut exister de manière indépendante ; les molécules sont composées d'atomes.
|
|
Nucléotide : |
Motif de base de l'ADN comportant trois éléments chimiques : une des quatre bases azotées (A, C, G ou T), un sucre, le désoxyribose, et un groupement phosphate. Dans l'ARN le sucre est le ribose et la base qui remplace la thymine (T) est l'uracile (U).
|
|
Oncogène : |
Type de gène impliqué directement ou indirectement dans la croissance et la division cellulaire et dont une mutation peut mener, en concertation avec d'autres oncogènes également mutés, au cancer.
|
|
Plasmide : |
Petite molécule d'ADN circulaire qui se réplique indépendamment du chromosome principal de la bactérie.
|
|
PCR ou RPC (" polymerase chain reaction ") : |
Amplification exponentielle d'une séquence d'ADN réalisée in vitro à l'aide d'une enzyme et de deux " amorces " délimitant les bornes de la séquence à amplifier.
|
|
Protéine : |
Molécule complexe dont le squelette est formé par l'enchaînement d'acides aminés, et pouvant avoir des fonctions aussi variées que la catalyse (enzymes), la reconnaissance d'agents étrangers (anticorps) ou le transport d'énergie (globine associée au fer dans l'hémoglobine).
|
|
Recombinant : |
Terme caractérisant une molécule d'ADN hybride formée à partir d'au moins deux fragments n'ayant pas la même origine, provenant soit de deux espèces différentes d'organismes, soit de deux fragments du même chromosome qui n'étaient pas adjacents à l'origine.
|
|
Rétrovirus : |
Virus dont le génome est formé d'ARN et dont l'action particulière consiste à utiliser une enzyme forçant la cellule hôte à créer de l'ADN viral. Cet ADN viral sera alors capable de pénétrer dans le noyau de la cellule infectée et de s'intégrer aux chromosomes. Le virus du SIDA est un rétrovirus.
|
|
Virus : |
Particule formée de matériel génétique entouré d'un manteau protéo-lipidique et programmé pour se multiplier à l'intérieur et aux dépens d'un hôte, bactérie ou cellule. On distingue les " virus à ADN ", dont le génome est formé d'ADN (les adénovirus en sont une espèce particulière), et les " virus à ARN ", dont le génome est formé d'ARN (les rétrovirus en sont un exemple). |
ANNEXES : ANNEXES :
|
- Annexe n° I : |
Le problème du financement des nouvelles thérapies. Gilles JOHANET. |
|
- Annexe n° II : |
La vaccination par ADN nu. Rapport de la mission pour la science et la technologie de l'Ambassade de France aux États-Unis. Septembre 1999. |
|
- Annexe n° III : |
Réflexions de lecture du rapporteur : les dérapages possibles au cours de l'existence. |
|
- Annexe n° IV : |
Entretiens du rapporteur. |
|
- Annexe n° V : |
Lettre de saisine de l'Office parlementaire d'évaluation des choix scientifiques et technologiques |
ANNEXE N° I
ANNEXE N° I
LE PROBLÈME DU FINANCEMENT DES NOUVELLES THÉRAPIES
LE
PROBLÈME DU FINANCEMENT DES NOUVELLES THÉRAPIES
Intervention
de M. Gilles JOHANET, directeur général de la Caisse nationale d'assurance
maladie,
au colloque sur :
" Les conséquences de l'évolution des
sciences
biomédicales. Enjeux de santé et réponses politiques "
, organisé
par le
rapporteur le vendredi 12 mars 1999 au Palais du Luxembourg
103(
*
)
.
2005 est la date convenue de l'achèvement du déchiffrage des séquences du génome
humain et de l'identification des 50 à 80 000 gènes qui le
constituent. Nous
allons assister à l'irruption dans le système de soins de la médecine génétique,
actuellement encore discrète sinon modeste.
Nous aurons ainsi une connaissance des quelque 5 000 maladies
monogéniques et
nous pourrons identifier les gènes impliqués dans les maladies d'origine
polygénique.
Ce sera l'avènement de cette fameuse médecine prédictive que nous annonçaient
Jacques
RUFFIER et Jean DAUSSET, avec un champ immense de possibilités diagnostiques et
de
thérapies géniques.
Cette innovation pose des problèmes d'ordre financier pour les différentes
activités
génétiques qui s'ouvrent à nous :
En ce qui concerne les pathologies courantes, le diagnostic génétique
introduit un
risque de " trop-plein " : pathologies courantes,
bénéficiaires
nombreux, il y a donc un marché avec des entreprises qui l'ont déjà investi
puisque
nous voyons arriver des kits de diagnostics génétiques. Cela pose deux
problèmes, celui
des indications assurant la prise en charge et celui de la qualité.
Pour l'autre segment de l'activité génétique concernant cette fois les
pathologies orphelines, il est clair que le défi naît, au contraire, d'un vide
potentiel
de la recherche. Se posent alors les questions, d'une part d'un financement
spécifique de
cette recherche, du transfert et de l'activité quotidienne, et d'autre part de
l'information et du contrôle de la qualité.
À la réflexion, il me semble qu'appliqués à cette innovation majeure qu'est la
médecine génétique, les risques liés à l'extension du champ des soins et à leur
financement sont entièrement confondus avec les risques que nous courons à
poursuivre
avec le système de soins actuel.
On peut distinguer quatre risques potentiels :
Premièrement, des dépenses considérables liées à un non-choix: on assiste à un
financement de l'innovation sans redéploiement ; le maître-mot de la
politique
française est depuis le 4 octobre 1945 " toujours en plus, jamais à la
place
".
Deuxièmement, des dépenses dont l'utilité médicale serait " variable
",
c'est-à-dire un trop-plein de tests,
a fortiori
de thérapies,
inappropriés dans
leur essence comme dans leur destination.
Troisième risque, des dépenses dont la mise en oeuvre s'opérerait avec des
inégalités
considérables d'accès :
Inégalités d'ordre médical, liées à la pathologie elle-même une fraction
non
négligeable des 5 000 maladies génétiques sont des maladies
orphelines pour
lesquelles la population potentiellement bénéficiaire est extrêmement étroite.
Or une
demande faible dans notre marché des soins est une demande inexistante.
Inégalité d'ordre social, fondée sur les critères classiques, producteurs
d'inégalité croissante dans notre système de soins, ou créée par un autre
facteur de
discrimination de la population qui est l'inégalité du savoir (où sont les
bonnes
équipes, qui est vraiment efficient ?).
Quatrième risque encouru : une mise en oeuvre de ces nouvelles pratiques
sans
contrôle suffisant de la qualité.
Face à ces risques, il me semble que l'on peut se poser cinq questions:
Premièrement, sommes-nous capables d'édicter des normes ?
Des normes de sécurité et de fiabilité des produits : on sait en
général
le faire, même si c'est parfois tardif
Des normes d'indication: qui doit être bénéficiaire ou non de la prise en
charge,
sachant que dans ce domaine, les coûts sont tels que cette prise en charge est
un
discriminant quasi absolu ?
Des normes d'agrément, de référencement des laboratoires, voire des
centres de
soins, ce qui a été introduit par les lois bio-éthiques.
Des normes de qualité. A l'heure actuelle, s'agissant des laboratoires,
les normes
de qualité ne sont financées et prévues que par le programme Biomed qui est un
programme européen ; la France a considéré jusqu'ici qu'elle pouvait se
dispenser
de toute action dans ce domaine. Ce programme ne porte que sur quatre
pathologies -qui
certes concernent un nombre important de patients, puisqu'il y a la
mucoviscidose, la
myopathie de Duchenne, etc.-, alors qu'il existe 5 000 maladies
monogéniques.
De surcroît, le programme Biomed n'a pas
a priori
vocation à être
éternel. En
définitive, sommes-nous capables d'instaurer une transparence, condition d'une
réelle
qualité ?
Deuxième question : sommes-nous capables de rendre cette transparence
opposable ?
Sommes-nous capables, si nous instaurons des normes d'agrément et de
référencement, de
ne pas financer les centres qui ne sont pas agréés, ce qui serait dans nos
pratiques
assez innovant ? Sommes-nous capables, si nous posons des indications de
prise en
charge, de les respecter ? Quand je dis " nous ", je
pense bien
entendu à l'assurance maladie mais aussi aux professionnels de la santé.
Troisième grande et capitale question : sommes-nous capables d'assurer le
financement de cette activité nouvelle par redéploiement ?
Le déremboursement de produits inutiles, et il est heureux de ce point de
vue-là -et de
ce point de vue-là seulement- que nous ayons un vaste choix, est certainement
l'une des
clés du progrès médical et social.
Quatrième question : en élargissant quelque peu, peut-on imaginer et
accepter des
modes de financement et d'organisation différenciés ?
S'agissant de la médecine génétique appliquée aux pathologies courantes pour
lesquelles il existe déjà une offre prise en compte par le marché, s'agissant
des actes
et des diagnostics stabilisés à moyen terme, il me semble tout à fait possible
de
financer ces activités à l'acte.
S'agissant en revanche des activités liées aux pathologies orphelines, le mode
de
financement actuel par dotation globale, avec une intégration des laboratoires
concernés
dans les centres hospitalo-universitaires, est parfaitement inapproprié. Ces
laboratoires
étant spécialisés, ils ont une vocation et une attractivité nationales ; un
financement sur la base d'établissements voire même de régions, ne peut
manifestement
prendre en compte qu'imparfaitement cette réalité. Sommes-nous par conséquent
capables
de différencier deux modes de financement avec, pour les pathologies rares, un
réseau
national constitué de laboratoires référencés et bénéficiant d'un financement
spécifique ?
Enfin, j'ai proposé trois principes d'action pour épouser la modernité dans ce
domaine : la transparence, la sélectivité, la coresponsabilité. Peut-on
imposer
ces trois principes d'action à la médecine génétique seule, tout en continuant
d'en
exonérer le reste des activités de soins ?
ANNEXE
n° II
ANNEXE n° II
AMBASSADE DE FRANCE AUX ÉTATS-UNIS
MISSION POUR LA SCIENCE ET LA TECHNOLOGIE
LA VACCINATION PAR " ADN NU "
LA VACCINATION PAR
ADN NU
Septembre 1999
Rapport de synthèse de la Mission aux États-Unis des
Professeur Patrice DEBRÉ, professeur d'immunologie, Hôpital Pitié-Salpêtrière,
Paris ;
Dr Bernard CHARLEY, directeur de recherche INRA, Jouy-en-Josas ;
Dr Lucyna COVA, directeur de recherche, INSERM, Lyon
|
Accompagnés de : |
- Stéphane ROY, attaché pour la science et la technologie (San Francisco) ; |
|
|
- Wahid BAKOUCHE, attaché pour la science et la technologie (Washington). |
Préambule
Ce rapport présente les résultats d'une mission effectuée en juin 1999 sur la
vaccination par " ADN nu " dans le cadre d'une action de
veille en
Recherche et Développement dans le domaine des biotechnologies aux États-Unis.
Elle
rassemble le contenu d'une série d'entretiens réalisés en Californie et dans la
région
de Washington. Dans certains cas, les données ont été complétées par des
informations
issues de documents.
Elle présente les récents développements sur le thème de la recherche sur la
vaccination à " ADN nu " en analysant la situation d'un
point de vue
scientifique tout en essayant de faire le point sur la stratégie des
entreprises de
biotechnologies et leurs interactions avec le monde académique.
Cette mission s'est rendue dans les laboratoires suivants : Chiron
Corporation,
Emeryville, Californie ; Dynavax Technologies Corp., Berkeley,
Californie ;
Center for Comparative Medicine, University of California à Davis,
Californie ;
Vical, San Diego, Californie; et Agricultural Research Center, United States
Department of
Agriculture, Beltsville, Maryland.
INTRODUCTION
I. STRUCTURES VISITÉES
1. Chiron Corporation.
2. Dynavax Technologies Corporation
3. Center for Comparative Medicine, University of California à Davis
4. Vical Inc.
5 Agricultural Research Service, USDA
6 Maxygen
II. APPRÉCIATION DE LA SITUATION AUX ÉTATS-UNIS
1. Optimisme des interlocuteurs
2. Approche thérapeutique ou préventive
3. Essais cliniques chez l'être humain
4. Importance de l'approche vétérinaire
III. DÉFIS À RELEVER
1. Augmentation de la réponse immunitaire
2. Ciblage des cellules cibles
3. Aspects réglementaires
IV. STRATÉGIE DES ENTREPRISES DE BIOTECHNOLOGIE
1. Nombre limité de sociétés de biotechnologies
2. Vical et ses partenaires
3. Approche de Chiron
V. CONCLUSIONS
INTRODUCTION
Le marché de l'immunothérapie et de la vaccinologie est devenu très compétitif
et
très vaste dans la diversité des maladies ciblées (cancer, maladies
infectieuses,
neurodégénératives...). Les sociétés de biotechnologie et les grands groupes
pharmaceutiques cherchent à produire des vaccins plus efficaces. D'ores et
déjà, de
nouvelles technologies permettent :
1. de développer de nouvelles méthodes de préparation ;
2. d'envisager de nouvelles cibles et
3. d'administrer ces vaccins différemment.
Le principe de la vaccination par " ADN nu " date d'une
découverte
inattendue faite en 1989 et publiée dans Science
104(
*
)
.
Une simple administration de plasmides contenant des séquences d'ADN codant
pour des
protéines permettait la pénétration de l'ADN dans les tissus et résultait en son
expression in situ. L'expression d'une protéine étrangère codée par de l'ADN
injecté
dans les cellules d'un tissu a provoqué une réponse immunitaire. Cette nouvelle
propriété a très vite semblé offrir de grandes opportunités pour les vaccins
thérapeutiques ou prophylactiques et révolutionner la vaccinologie aux
États-Unis.
Dans le cadre de son action de veille dans le secteur des biotechnologies, la
Mission pour
la Science et Technologie aux États-Unis a choisi le thème de la vaccination par
" ADN nu ". Faisant appel à des experts français, cette
étude a
pour objet de mieux faire connaître la situation américaine, de déterminer ses
tendances en recherche et développement et d'analyser la faisabilité de cette
approche
en vaccinologie.
Dans cette optique, cette mission d'étude a consisté à la visite de centres de
recherche privés ou académiques, différents par leurs centres d'intérêts, leurs
préoccupations et leur degré d'implication. Cette note est le résultat
d'entretiens
avec des Chief Executive Officer (CEO), des Chief Scientific Officer (CSO), des
Directeurs
de recherche et des Professeurs d'Université.
Dans un premier temps, nous présentons les différents laboratoires choisis et
visités,
puis nous analysons la situation aux États-Unis à la fois sur le plan
scientifique et
stratégique avant de donner quelques éléments sur le paysage des sociétés de
biotechnologies impliquées dans cette approche.
I. STRUCTURES VISITÉES
1. Chiron Corporation.
Personnes rencontrées :
Margaret A. LIU, MD, vice-président, Vaccines and Gene Therapy
Research ;
Jeffrey B. ULMER, Ph. D., Director, Vaccines Research ;
John J. DONNELLY, Ph. D., Director, Vaccine Research.
Société de biotechnologie de grande importance, Chiron a été fondée en mai 1981
à
Emeryville, à proximité des Universités de Californie à Berkeley (UCB) et à San
Francisco (UCSF) par William RUTTER et son ancien élève Edward PENHOET. Devenue
publique
en 1983, Chiron multiplie les accords de coopération en recherche et
développement pour
pouvoir croître. C'est en 1988 qu'a lieu l'identification du virus de
l'hépatite C.
Cette découverte ouvre la voie à la fabrication de tests et à la réalisation de
vaccins et de traitements. Après de multiples rachats (Cetus en 1991, etc.),
l'entreprise
devient très vite un groupe biomédical à l'expertise reconnue. Chiron emploie
environ
7000 personnes dont 1 500 sur le site d'Emeryville. Implantée sur
l'ensemble
des États-Unis et de part le monde, Chiron a pour ambition d'être la première
entreprise de biotechnologie dont la création de produits transformera la
pratique de la
médecine. En 1998, ses bénéfices se sont montés à 220 millions de dollars.
Chiron est actif dans trois grands secteurs de l'industrie de la santé :
les
diagnostics, les vaccins et les biothérapies. La recherche et développement de
Chiron se
concentre sur la thérapie génique, les vaccins, la génomique et la découverte de
nouvelles molécules thérapeutiques.
Chiron a clairement misé sur l'approche d'immunisation par " ADN
nu "
dans le cadre de sa politique de R&D en vaccinologie. Les recherches dans
ce domaine
portent essentiellement sur les différentes modalités de cette
technologie :
- mécanismes fondamentaux, présentation antigénique, intégration de
l'ADN ;
- ciblage de l'antigène aux cellules dendritiques (recrutement à l'aide de
chimiokines) ;
- tentatives pour éviter la dégradation de l'ADN (digestion par
nucléase) ;
- approche par électro-incorporation in vivo pour en augmenter
l'intégration
cellulaire.
Cette compagnie se focalise sur deux applications essentielles : le VIH
(virus de
l'immunodéficience humaine) et l'hépatite C. Un groupe d'une quinzaine de
personnes
travaille aujourd'hui sur le sujet de la vaccination par " ADN
nu ".
2. Dynavax Technologies Corporation
Personnes rencontrées
:
Dino DINA, M.D., President & Chief Executive Officer ;
Joseph EIDEN Jr., M.D., Ph. D., Vice President, Research and
development ;
Gary VANNEST, Ph. D.
Dynavax a été fondée en 1997 par les Professeurs Eyal RAZ et Dennis CARSON de
l'Université de Californie à San Diego en association avec le Professeur
Lawrence
LICHTENSTEIN du John Hopkins School of Medicine à Baltimore (Maryland). Les
docteurs
CARSON et RAZ sont les co-découvreurs des séquences immunostimulatrices qui
forment la
base de la technologie développée par Dynavax. Dynavax est une société privée
qui ne
possède pas encore de revenus. Elle emploie 22 personnes et finance la
recherche de
9 chercheurs à l'université de Californie à San Diego.
Ce groupe qui avait parié sur l'utilisation de la vaccination par
" ADN
nu " (5 brevets déposés), en particulier dans ses applications
pour
l'asthme et l'allergie, s'est aujourd'hui reconverti vraisemblablement à cause
des prises
de brevets par les autres compagnies de biotechnologies, de l'absence
d'applications à
très court terme et des aspects réglementaires encore inexplorés. Dynavax a
choisi de
s'intéresser aux séquences d'ADN immunostimulatrices (ISS) en tant qu'adjuvant.
Dynavax espère pouvoir avoir deux de ses candidats de séquences
immunostimulatrices en
phase I d'essais cliniques en 1999.
3. Center for Comparative Medicine, University of California à Davis
Personnes rencontrées
:
Steve W. BARTHOLD, DVM, Ph. D., Professeur Directeur ;
Paul L. UCIW, Ph. D., Associate Professor ;
Gary RHODES, Ph. D. Associate Professor ;
Philippe LENA, Ph. D., Assistant Research.
Au cours de cette journée, une table ronde avait été organisée et les personnes
suivantes sont intervenues : Professeurs Murray GARDNER, Marta
MARTHAS, Peter
BARRY, Chris MILLER et Mike McCHESNEY.
Le Center for Comparative Medicine (CCM) est un centre de recherche et
d'enseignement
spécialisé dans l'étude de la pathogénie des maladies infectieuses communes aux
animaux et aux humains. Le CCM regroupe 13 chercheurs choisis à l'école de
médecine et à l'école vétérinaire. Le CCM veut développer une activité de
recherche
intégrée au modèle animal entier. De ce fait, le CCM jouxte le
" California
Regional Primate Research Center " (CRPRC), centre de primatologie
unique au
monde et se trouve a proximité du " Targeted Genomics
Laboratory "
(TGL). Le TGL a pour but de créer et maintenir une collection de souris
transgéniques,
utilisées pour étudier l'origine génétique et le développement de certaines
maladies.
Le CCM est né de la volonté d'un groupe de chercheurs dans la perspective de
regrouper
la thématique de la pathogénie des maladies infectieuses pour générer des
collaborations et des projets de recherche communs et de devenir un centre
d'excellence de
la recherche en biologie intégrée à l'animal entier.
Le programme de vaccination par " ADN nu " a débuté en 1995
avec
l'arrivée de Gary RHODES, ancien employé de Vical. Cette visite a permis de
noter
plusieurs applications des vaccins par " ADN nu " notamment
pour des
applications vétérinaires dans le domaine du FIV
105(
*
)
et du SIV
106(
*
)
. Les résultats (éventuellement
encourageants pour le FIV) nécessitent cependant largement d'être complétés
avant que
l'on puisse en tirer de réelles données, notamment sur l'avantage de cette
technique par
rapport aux vaccinations classiques en matière d'efficacité. On a eu
l'impression que
même au sein de cette structure universitaire importante, qui possède un des
plus grand
centre des primates (CRPRC), les équipes impliquées dans le développement du
vaccin par
" ADN nu " sont petites et les résultats obtenus, bien
qu'encourageants, n'ont pas permis une démonstration claire. Ces travaux ne
reflètent
pas de manière complète les progrès réalisés dans ce domaine aux États-Unis. A
titre
d'exemple, les travaux présentés sur la vaccination des macaques contre le SIV
montrent
un échec de cette approche au niveau prophylactique, c'est-à-dire des
difficultés
d'induire une immunisation. Ceci est en contradiction avec les résultats
récemment
publiés dans Nature Medicine par la prestigieuse équipe de Harriet Robinson
(Emory
University, Atlanta) qui a montré que le vaccin par " ADN
nu " induit
une immunité cellulaire protectrice contre le SIV dans ce modèle. Il est donc
très
difficile d'évaluer les progrès de recherche sur le vaccin à " ADN
nu " en se basant sur les travaux de Davis.
4. Vical Inc.
Personnes rencontrées
:
Jon A. NORMAN, Ph. D., Vice-President. Research ;
Robert H. ZAUGG, Ph. D., Vice-President, Business Development ;
Marston MANTHROPE, Ph. D., Executive Director, Cell Biology ;
Seth D. GOLDBLUM, Director, Corporate Development ;
Carl J. WHEELER, Ph. D., Director Chemistry.
Vical a été fondée en 1987 pour travailler en recherche et développement dans le
domaine de la chimie des lipides. C'est en 1989 que leurs travaux les ont
conduit à
découvrir que l'ADN nu injecté pouvait avoir un effet immunologique et qu'ils
ont
déposé un brevet. Devenue publique en 1993, Vical continue à collaborer avec de
grands
groupes pharmaceutiques pour l'utilisation de la vaccination à " ADN
nu " à usage thérapeutique. En 1998, Vical avait des revenus de
8 millions de dollars et enregistrait toutefois une perte de
7,5 millions de
dollars. La compagnie emploie 101 personnes au premier trimestre de
l'année 1999.
Cette compagnie est certainement une des plus avancées dans le domaine. Elle a
donné en
licence le brevet de la technologie de l'immunisation par " ADN
nu "
pour la majeure partie des pathologies pouvant être évoquées. A l'heure
actuelle, les
protocoles concernent d'une part des essais en matière de vaccination
anti-paludéenne en
corrélation avec le Naval Research Institute à Washington, d'autre part des
essais
expérimentaux dans le modèle murin pour déterminer les meilleurs modèles
expérimentaux d'infection. Vical a par contre conservé en développement propre
l'utilisation des séquences d'ADN pour une approche d'immunothérapie du cancer.
Vical a
ainsi 3 produits en essais cliniques: allovactin 7 (phase III),
leuvectin
(phase II) et vaxid (phase I).
Vical conserve aussi une activité de recherche fondamentale dans deux domaines
liés à
l'injection d'ADN nu. D'une part, elle se concentre sur la délivrance de
protéines
thérapeutiques à partir d'injection de gène nu, d'autre part elle se focalise
sur les
processus de transfert de gènes en thérapie génique.
5. Immunology and Disease Resistance Laboratory, Agricultural Research
Service, USDA
Personnes rencontrées
:
Joan LUNNEY, Research Leader ;
Mark JENKINS, LPSI ;
Kang Duk CHOI, LPSI ;
John and Rose HAMMOND, PSI.
Les chercheurs rencontrés travaillent sur plusieurs maladies animales contre
lesquelles
ils élaborent actuellement des stratégies de vaccination par ADN : maladies
parasitaires et virales des poules, des bovins, du porc. En matière de
vaccination contre
les parasites protozoaires, des essais sont en cours contre la cryptosporidiose
bovine et
contre les coccidioses aviaires : utilisation d'ADN administré chez les
bovins par
voie intramusculaire ou intra-mammaire (" gene-gun ")-,
injection
d'ADN autour des ganglions associés à la glande mammaire (Mark JENKINS). Chez
la poule,
vaccination ADN par voie intramusculaire avec un gène du parasite, en
association avec un
plasmide codant pour l'IL-15 aviaire (Kang Duk CHOI). Chez le porc, des
essais de
vaccination sont en cours contre une herpès-virose (maladie d'Aujeszky), ou
contre la
toxoplasmose, avec de nouveau association de plasmides codant pour un antigène,
et de
plasmides codant pour des cytokines porcines (J. LUNNEY).
Nous avons aussi rencontré, en marge de la vaccination par ADN, des chercheurs
travaillant sur l'utilisation de plantes recombinantes comme vecteurs de vaccins
(J. et R. HAMMOND).
6. Maxygen
Personne rencontrée
:
Russell J. HOWARD, Ph. D., President et CEO.
Maxygen est une entreprise privée fondée en 1997 comme spin-off de Glaxo
Wellcome
(Oxford, Angleterre) et d'Affymax (Santa Clara, Californie) par Alejandro
ZAFFARONI,
Russell HOWARD, Isaac STEINER et Pim STEMMER. Ce dernier, ancien employé
d'Affymax, a
développé le " DNA shuffling " qui constitue la base de la
technologie proposée par Maxygen. Le " DNA shuffling " est
une
approche nouvelle basée sur le " brassage " d'ADN de la
même famille
pour créer de nouveaux épitopes qui pourraient être particulièrement
intéressants
pour des nouveaux vaccins. Il est intéressant de noter que Robert
WHALEN
107(
*
)
vient tout juste de rejoindre
Maxygen pour utiliser cette
approche technologique et développer de nouveaux vaccins par " ADN
nu " avec comme objectif d'augmenter la réponse immunitaire.
II. APPRÉCIATION DE LA SITUATION AUX ÉTATS-UNIS
À partir de ces structures visitées (ce qui ne constitue qu'un échantillon des
recherches et applications industrielles en cours aux États-Unis), il apparaît
clairement que la stratégie de la vaccination par " ADN nu "
représente une approche qui, en 10 ans seulement, a mobilisé et mobilise
toujours
nombre d'équipes académiques et de structures industrielles. A titre
d'illustration, le
Keystone Symposium sur " DNA vaccines : immune responses,
mechanisms and
manipulating antigene processing " a regroupé plus de
300 personnes.
1. Optimisme des interlocuteurs
Le fait qu'une entreprise de la taille de Chiron investisse une part importante
de ses
efforts en vaccinologie sur la vaccination par " ADN nu ",
et de
surcroît ait recruté toute l'ancienne équipe de Margaret LIU chez Merck
(Jeffrey ULMER
et John DONNELLY) est à lui seul la preuve que les principaux intervenants
industriels
dans ce domaine " croient " en son succès. Le même enthousiasme par
rapport à
la vaccination par " ADN nu " se retrouve bien évidemment
chez Vical.
Les doutes principaux tiennent cependant aux délais importants requis avant le
stade de
l'application: une entreprise de petite taille comme Dynavax a de ce point de
vue fait
l'analyse qu'elle ne pouvait pas se lancer, pour des raisons scientifiques
(méthode
encore faiblement efficace) et par conséquent financières, dans des recherches
à long
terme sur la vaccination par " ADN nu ". C'est pour cette
raison
qu'elle a choisi une approche à plus court terme sur l'effet immuno-adjuvant des
plasmides eux-mêmes, et leur utilisation potentielle pour lutter contre les
allergies.
La plupart des interlocuteurs ont aussi tempéré leur optimisme sur les chances
de
succès de cette approche, en faisant le constat que la transposition des bons
résultats,
obtenus (et publiés) chez la souris, à d'autres espèces animales, y compris les
primates, posait des problèmes : il est admis par tous que le pouvoir
immunogène
des vaccins par " ADN nu " actuellement en cours d'étude est
inférieur à celui des vaccins classiques. Il est par conséquent essentiel
d'augmenter
cette immunogénicité, par plusieurs approches (voies d'immunisation; méthodes
d'injection; association ADN puis rappel par la protéine ; électroporation
in
vivo ; effet adjuvant de l'ADN lui-même, des séquences ISS ou CpG ;
formulation de l'ADN avec des adjuvants classiques; encapsulation de l'ADN,
administration
de l'ADN par des vecteurs; utilisation de cytokines à effet adjuvant
co-administrées
sous forme d'ADN...).
2. Approche thérapeutique ou préventive
L'approche vaccinale (préventive) est la plus classique et est surtout
appliquée à la
recherche de vaccins anti-infectieux chez l'homme et l'animal. Toutes les
étiologies
infectieuses sont concernées : virus, bactéries, parasites. Son efficacité
protectrice contre différents pathogènes a été clairement démontrée dans
différents
modèles animaux. Bien que la protection obtenue ne soit pas supérieure à celle
induite
par les vaccins recombinants, les vaccins à " ADN nu " ont
de
nombreux avantages. Parmi les atouts principaux de ces vaccins on peut
citer :
- synthèse permanente d'antigène dans les cellules qui permet une
stimulation
constante du système immunitaire ;
- induction d'une réponse non seulement humorale mais aussi
cellulaire ;
- modulation facile des vecteurs à base d'ADN, ce qui est particulièrement
intéressant dans le cas d'émergence des variants d'échappement ;
- faible coût, car la préparation d'ADN est peu onéreuse, et
l'immunisation par
"ADN nu" nécessite peu d'injections évitant de lourdes et coûteuses
procédures de synthèse et de purification des protéines ;
- facilité de stockage (pas besoin de réfrigération) ; cette approche
vaccinale apparaît comme particulièrement pertinente pour les pays en voie de
développement.
L'efficacité thérapeutique du vaccin à " ADN nu " est moins
bien
documentée. Outre les perspectives anti-tumorales pour lesquelles une autre
mission
d'étude est prévue, des approches thérapeutiques par utilisation de l'ADN sont
en cours
d'étude. Un des avantages essentiels du vaccin génétique comme nouvel outil
thérapeutique pour les sujets atteints d'infection chroniques est l'induction
d'une
réponse cellulaire via les molécules du CNOE de classe I. C'est un atout
majeur par
exemple pour le traitement des hépatites B chroniques, car les CTL induits
par
l'injection d'ADN nu peuvent reconnaître et lyser les cellules hépatiques
infectées.
L'approche thérapeutique est utilisée également pour le traitement d'allergie
et de
l'asthme par injection d'oligodéoxynucléotides (ODN) immunostimulants (exemple
ODN de
22 nucléotides) seuls ou conjugués chimiquement avec l'allergène (Dynavax).
D'autres possibilités d'utilisation thérapeutique de l'ADN concernent
l'injection
intra-tumorale de plasmides codant pour l'IL-2 et pour une molécule HLA de
classe I
(essais phase III chez l'homme), ou l'injection répétée de plasmides
codant pour
l'érythropoïétine chez le chien âgé (Vical).
3. Essais cliniques chez l'être humain
Les premiers essais cliniques chez les humains utilisant la vaccination par
" ADN nu " ont été effectués par PowderJect
108(
*
)
en collaboration avec Glaxo Wellcome, pour la
prophylaxie de
l'hépatite B.
Les résultats prometteurs de protection efficace obtenus récemment pour
différents
virus (influenza, VIH, HTLV I, HBV, Ebola, Cytomegalovirus, HTLV, rage...)
dans
différents modèles animaux ont été à la base des premiers essais de vaccination
par
" ADN nu " chez l'homme. Plusieurs essais de vaccination par
" ADN nu " sont conduits actuellement chez des volontaires
aux
États-Unis; Influenza (John Hopkins University, Baltimore, Maryland), malaria
(en
collaboration avec le Capitaine Steve HOFFMAN de l'U.S. Naval Medical Research
Center
(Bethesda, Maryland) et Pasteur Mérieux Connaught), hépatite B (University
of
Cincinnati, Ohio), VIH (University of Pennsylvania à Philadelphie en
collaboration avec
Merck et Vical), Herpès (University of Washington à Seattle)... Ce qui montre
l'intérêt des autorités américaines pour cette approche.
4. Importance de l'approche vétérinaire
La facilité de préparation de l'ADN, le faible coût de sa production, l'absence
de
danger et sa stabilité à la température, expliquent que de nombreuses
expérimentations
aient été menées chez les animaux domestiques, espèces cibles directes des
vaccins
ADN. Ces essais concernent toutes les espèces animales domestiques (truites,
oiseaux,
mammifères) et tous les pathogènes importants. Les structures académiques de
recherche
visitées (UC Davis et USDA à Beltsville) ont des programmes de recherche sur la
vaccination par ADN chez les animaux domestiques, chaque équipe travaillant sur
" sa " maladie " préférée ". On
retrouve les
mêmes tendances que dans les essais sur animal de laboratoire, sur primates ou
chez
l'homme : besoin d'augmenter l'immunogénicité, par exemple par l'emploi
simultané
de plasmides codant pour des cytokines (IL-15 chez le poulet contre la
coccidiose;
plasmides codant pour diverses cytokines chez le porc contre la toxoplasmose ou
contre une
herpèsvirose du porc) ; recherche des voies optimales d'administration. Un
accent
particulier est mis sur la possibilité de vacciner le jeune animal (vaccination
néonatale), en présence d'anticorps maternels, ce qui représente un net
avantage de la
vaccination par " ADN nu " par rapport aux vaccins
classiques
protéiques (UC Davis).
La situation des applications vétérinaires de la vaccination par " ADN
nu " est marquée au niveau industriel par le fait que la société
Vical a
signé avec la société Mérial (fusion des activités Santé animale de
Rhône-Mérieux
et Merck) un accord d'exclusivité limité dans le temps, au cours duquel Mérial
évalue
les domaines d'application. Au terme de cette période, Mérial devra définir les
maladies et les espèces animales pour lesquels il aura l'exclusivité de
l'exploitation
du brevet Vical.
III. DÉFIS À RELEVER
1. Augmentation de la réponse immunitaire
Le besoin évident d'accroître l'immunogénicité des vaccins ADN se traduit en
termes de
pistes de recherche par :
- variations autour de la voie d'administration (sous cutanée,
intramusculaire,
mucosale) et méthode d'injection (seringue, systèmes sous pression avec
l'exemple de
Bioject ; par le " gene gun " avec l'exemple de
PowderJect®
System) ;
- différentes présentations de l'ADN (en association avec des
microparticules, des
acides gras; vecteur réplicatif type alphavirus; association avec des adjuvants
classiques (alun) ...) ;
- effets adjuvants de cytokines co-injectées sous forme plasmidique
(notamment
GM-CSF en essai chez l'homme et les animaux domestiques) ;
- association vaccin " ADN nu " et rappel par
l'antigène
protéique ;
- modification des séquences immunostimulantes du plasmide lui-même ou
injection
simultanée d'oligonucléotides.
2. Ciblage des cellules-cibles
Le constat qu'une part importante de l'ADN injecté est perdu, dégradé par des
macrophages résidents, et donc ne transfecte pas suffisamment de cellules
justifie la
recherche de moyens susceptibles d'augmenter in vivo la transfection des
cellules
musculaires (injection intramusculaire) : utilisation d'un système à usage
unique
d'injection et électroporation in vivo (Chiron).
3. Aspects réglementaires
Personne rencontrée
:
Imran KHAN, Ph D., Bio-Trends International, Inc., Research Leader.
En médecine vétérinaire, le correspondant de Biotrends International comme ceux
de
Vical en relation avec Mérial considèrent qu'il n'y aura pas de frein majeur à
l'utilisation de l'ADN en vaccination des animaux, à certaines conditions:
absence de
marqueur de résistance aux antibiotiques à l'exception du marqueur
kanamycine ;
absence de diffusion du plasmide injecté, dans les organes génitaux, le liquide
séminal; absence d'insertion dans la lignée germinale.
En médecine humaine, les essais effectués par Vical ont donné lieu à une
approche
soigneusement évaluée avant de soumettre le protocole expérimental à la FDA
(Federal
Drug Administration).
IV. STRATÉGIE DES ENTREPRISES DE BIOTECHNOLOGIE
1. Nombre limité de sociétés de biotechnologies
Le nombre de sociétés de biotechnologies impliquées dans la recherche et
développement
en vaccination par " ADN nu " est assez limité. Outre les
sociétés
visitées, il est intéressant de noter que PowderJect Vaccine Inc., a une
activité très
importante dans ce domaine en utilisant sa plate-forme technologique d'injection
PowderJect®. Ainsi au mois de janvier 1999, PowderJect et Glaxo Wellcome
(Oxford,
Angleterre) ont étendu leur collaboration sur la vaccination par " ADN
nu " pour l'hépatite B, le VIH, des maladies infectieuses et
certains
cancers. Cette extension de la collaboration fait suite aux premiers résultats
prometteurs des tests cliniques pour le vaccin de l'hépatite B. Il semble
aussi que
la compétition soit croissante dans le domaine de la vaccination par
" ADN
nu ". En effet, de nombreuses sociétés de biotechnologies (Chiron,
Vical,
Merck, PowderJect...) sont activement impliquées dans l'application de cette
approche et
le domaine est couvert par des prises des brevets.
Compte tenu que le domaine de vaccination par " ADN nu "
dans l'état
actuel est largement couvert par des brevets et que le développement de
nouvelles
approches requière un investissement important en recherche, on comprend que des
start-up, comme Dynavax et Apollon
109(
*
)
, ne
soient pas
très compétitives et préfèrent changer d'orientation. Par contre, on note
simultanément l'implication d'autres petites compagnies de biotechnologie dans
le domaine
du vaccin à " ADN nu ", mais qui proposent des stratégies
très
différentes. C'est le cas par exemple de Maxygen qui veut commercialiser la
technique de
" DNA shuffling ".
Enfin, il est intéressant de noter qu'au cours de ces entretiens, les
différents acteurs
impliqués dans la R&D sur la vaccination par " ADN nu "
aux
États-Unis ont été très souvent " débauchés " par
différentes
compagnies qui se sont offertes ainsi une expertise. Ainsi Dino DINA, CEO et
Président de
Dynavax, était au préalable employé par Chiron Corporation depuis 1982 où il a
dirigé
le département Vaccins. Il a été remplacé par Margaret LIU qui est à l'origine
de
l'alliance de Merk et de Vical. Enfin, le passage de Gary RHODES (Professeur à
UC Davis)
de Vical au milieu académique a permis le démarrage de cette activité sur le
campus.
2. Vical et ses partenaires
Vical a compris très tôt l'intérêt du développement de la vaccination par
" ADN nu ". Très rapidement Vical a couvert par les brevets
la
vaccination par ADN pour les différents virus (VHB, VIH, Influenza...),
bactéries,
parasites et cancer.
Vical a ensuite accordé des licences à de grand groupes pharmaceutiques pour
l'utilisation de sa technologie d'immunisation par " ADN
nu ". Dès
1991, Merck Research Laboratories a obtenu une licence pour utiliser cette
approche dans
le traitement des maladies infectieuses (pour HBV, HIV et HCV), et une stratégie
similaire a été adoptée pour Pasteur Mérieux Connaught. Mérial (à l'époque Rhône
Mérieux) a obtenu la licence de cette technologie pour des applications
vétérinaires en
1995. En 1997, Merck a repris une licence pour les maladies cardio-vasculaires
et
Rhône-Poulenc Rorer pour les maladies neurodégénératives. Tout dernièrement, en
1999,
Pfizer vient de donner en licence la technique de délivrance des gènes pour des
applications en santé animale
110(
*
)
.
Un accord de " cross-licensing " a été signé entre Dynavax
et Vical
au début de l'année 1998 donnant l'accès aux technologies respectives des deux
partenaires. Ainsi, Dynavax a accès à certains brevets qui couvrent
l'immunisation par
ADN dans le domaine des maladies de l'asthme et de l'allergie. En retour Vical
obtient
l'accès à la technologie développée par Dynavax pour l'immunostimulation de
certaines
séquences d'ADN pour utiliser dans le domaine des maladies infectieuses et du
cancer.
Enfin, Vical garde uniquement la mise au point des vaccins pour les thérapies
du cancer.
3. Approche de Chiron
La situation de Chiron est différente car cette très importante société de
biotechnologie a beaucoup investi dans le vaccin à " ADN
nu " contre
HIV et l'hépatite C. Chiron a " débauché " de Merck
d'abord
Margaret LIU et ensuite toute son équipe avec des chercheurs de très haut
niveau comme
Jeffrey ULMER et John DONNELY qui sont une référence dans le domaine
d'immunisation
génétique. Cependant, comme la licence est détenue par Merck, le problème de
rachat va
être posé au moment de la commercialisation du vaccin.
Sous la houlette de Margaret LIU, Chiron a donc très nettement concentré ses
efforts sur
l'aspect technologique de la vaccination à " ADN nu ". De
l'avis
même de nos interlocuteurs, une telle approche est nécessaire pour d'une part
augmenter
l'efficacité de l'immunisation et d'autre part contourner les brevets déposés
par Vical
qui semblent verrouiller le marché. Le but est de résoudre les problèmes
technologiques
afin d'utiliser cette approche vaccinologique pour différentes maladies.
IV. CONCLUSIONS
Cette mission a permis de montrer l'intérêt de cette technologie et son
développement
dans l'industrie des biotechnologies. Tous les interlocuteurs s'accordent à
dire que la
vaccination à " ADN nu " a de nombreux avantages dont la
possibilité
de prédire/combattre les changements observés dans les souches de nombreux
agents
infectieux et de présenter les antigènes sous leur forme native, la stabilité
de la
réponse immunitaire, la simplicité de production en utilisant des procédés de
fermentation bactérienne, et la lutte contre l'immunosénescence (activité
développée
par Pharmadigm Inc).
Cependant, cette méthode demande encore à être comparée aux vaccins
traditionnels,
étudiés en matière de stratégie vaccinale (technique " prime
boost ") et des recherches dans le domaine fondamental avant de
pouvoir
clairement définir sa place et son avenir en vaccinologie présentative et/ou
thérapeutique.
Cette mission a aussi permis d'apprécier l'intérêt et l'enthousiasme des
sociétés de
biotechnologies pour la vaccination par " ADN nu ". Le
domaine est
très largement couvert par les brevets ce qui rend difficile l'émergence de
start-up, à
l'exception de celles qui proposent des approches méthodologiques innovantes.
L'immunisation par " ADN nu " est considérée aux États-Unis
comme
une approche d'avenir qui est en train de révolutionner la vaccinologie et
l'immunothérapie comme l'attestent des publications dans les journaux de très
haut
niveau et des nombreux essais chez l'homme. L'appréciation de la recherche dans
le
domaine fondamental est difficile en se basant sur les structures académiques
visitées
et devrait être complétée par la visite des laboratoires de pointe sur la côte
Est.
ANNEXE N°
III
ANNEXE N° III
Réflexions de lecture du rapporteur :
Réflexions
de lecture du rapporteur :
LES DÉRAPAGES POSSIBLES AU COURS DE L'EXISTENCE
LES
DÉRAPAGES POSSIBLES AU COURS DE L'EXISTENCE
De génération
en génération, les informations contenues dans le noyau, 2x23 chromosomes
pour
chacun de nous, sont les copies de celles que possédaient notre ancêtre,
l'
Homo
sapiens
sapiens
, modifiées lors des recopiages, au fil des divisions
cellulaires, en mal souvent, en bien exceptionnellement.
Au cours de la vie de chacun, le patrimoine génétique initial assure les
renouvellements
nécessaires aux remplacements des cellules qui disparaissent normalement, qu'il
s'agisse
des cellules de la peau comme de tous nos organes sauf en ce qui concerne le
muscle
cardiaque et le cerveau. Ces deux organes, essentiels à la vie, ne bénéficient
pas de
ces possibilités de renouvellement : leur usure provient de l'apparition,
dans la
cellule, de facteurs capables d'engager l'apotpose cellulaire.
Il est reconnu et admis que si des cellules germinales (blastes) sexuelles,
(début de la
lignée ovocytaire ou au début de la lignée des spermatozoïdes) subissent une
altération du génome, cela peut donner une mutation ou une maladie transmise
génétiquement (trisomie 21)
Si ces cellules sont des cellules germinales d'une lignée à renouvellement
rapide
(derme, lymphoblastes), la mutation va créer une maladie à retentissement
important
puisqu'un seul blaste malade sera le " père " de nombreuses
cellules
matures après multiples division (cancers cutanés, leucémies). Ce système de
renouvellement continue est destiné à pallier une usure naturelle (peau).
Si ces cellules sont des cellules très différenciées à renouvellement faible ou
nul
(neurones myocardiocytes), le plus souvent cela aboutit à la mort de la cellule
et à la
dégénérescence progressive de l'organe ; si cette destruction ou cette
erreur de
retranscription du génome touche la partie codant le mécanisme de division
cellulaire
(mécanisme inhibé dans l'exemple du neurone et du coeur), le réveil de ces
mécanismes
peut provoquer la naissance d'une tumeur (cancer) constitué d'une masse de
filles de
cette cellule qui n'aurait jamais dû retrouver la capacité de se diviser.
L'apoptose est due à l'exécution du message de leur mort que contiennent des
cellules
essentielles à la vie. C'est probablement une partie du génome qui est réprimée
(cachée à la rNa transcriptase pendant la vie cellulaire) : à un moment
donné, il
va être exprimé donnant naissance à des protéines toxiques entraînant
l'autodestruction de la cellule. L'apoptose est un phénomène cellulaire
autonome, ce
n'est pas une attaque extérieure de la cellule. Elle serait donc autoprogrammée
pour se
détruire.
Il y une organisation duale des données génétiques. Ce sont les allèles :
un
gène de prédisposition à une longue vie (gène du centenaire) serait aussi
porteur
d'une prédisposition à l'infarctus du myocarde vers 40 ans. Lequel sera
dominant ? et pourquoi ? Les conditions de vie contribuaient-elles à
la
victoire de l'un ou de l'autre ?
Même si la réalité n'est pas aussi simple , il n'en reste pas moins que des
transformations passagères, plus ou moins durable, peuvent devenir
transmissibles et
rompre, modifier l'équilibre initial ; des maladies peuvent apparaître
sans pour
autant être automatiquement transmissibles.
" Un pourcentage élevé du potentiel génétique humain reste
silencieux.
L'expression des gènes est régulée de façon physiologique en réponse à des
facteurs
externes (condition de nutrition, stress variés, infection virales etc.) ou
internes
(mutations, actions hormonales) lors de processus tumoraux ou au cours du
vieillissement.
Que sait-on de l'organisation du noyau, des compartiments nucléaires où
s'activent ou
bien se répriment les gènes, se maturent les ARN, s'assemblent les ribosomes,
s'organisent l'import et l'export des ARN et des protéines ? Ces questions
relevant
de l'architecture du noyau vont devenir de plus en plus essentielles.
La cellule contient des horloges internes (cyclines et protéines kinases qui
régulent le
cycle cellulaire, les télomères qui régulent le nombre de cycles, des programmes
d'apoptose et de vieillissement, qui régulent la survie cellulaire).
Connaître la fonction des gènes est une tâche de grande ampleur et
d'importance, car
cette connaissance constitue la base d'une physiologie intégrée. On sait que
les grandes
fonctions de base et les machineries cellulaires sont conservées de la levure
de bière
à l'homme ". (D'après M. YANIC et A. SENTENAC Rapport
Académie des Sciences).
Une autre source de modification se produit lors de l'appariement des
chromosomes
d'origine maternelle et d'origine paternelle, intervenant 18 heures après
la
fécondation et aboutissant à la formation du zygote ; nous avons tous la
même
quantité de " livres " dont le sujet est imposé à
tous :
contribuer à la naissance d'un être humain complet. Mais chacun des parents
n'apporte
pas exactement le même texte sur le même sujet ; par exemple le père donne
la
recette des yeux bleus, la mère peut être sans projet ou apporter celle des yeux
marrons, recette dominante qui s'imposera aux yeux bleus. Pour avoir les yeux
bleus, il
faut soit que les parents aient tous deux la recette des yeux bleus, soit que
la mère
n'ait pas de recette ou porte une recette illisible.
Enfin la " consommation constante " des protéines finalement
éliminées après usage sous forme d'urée, exige leur fabrication également
constante : au cours de celle-ci des incidents, des erreurs peuvent
apparaître ; comme dans le fonctionnement d'une automobile se dégrade par
usures ; des incidents, des accidents, des maladies, non transmissibles,
peuvent,
chez les êtres vivants, se manifester au cours d'une existence.
1. Comme chacun, dans un dictionnaire, en connaissance des 26 lettres
alphabétiques,
peut lire les mots utiles au langage des hommes entre eux, comprendre le sens
de chacun
d'entre eux, selon sa place dans une phrase, dans des ouvrages ou dans des
discours, les
généticiens, suffisamment expérimentés, peuvent lire dans ce dictionnaire
humain, le
génome, à condition d'avoir la connaissance des 24 lettres
111(
*
)
de notre alphabet d'humain. Leurs places, leurs présences sur les gènes
.donnent un
sens, un rôle, une activité particulière aux comportements de chacun de nous,
avec une
particularité essentielle : chaque bibliothèque est propre à chacun, même
chez
des jumeaux parfaits.
Malgré cette diversité certaine entre les individus, les être humains ne
reproduisent
que des êtres humains se ressemblant alors que dans le règne végétal et animal
les
différences aboutissent à des espèces qui, entre elles, de l'arbre majestueux au
pissenlit ou à la violette, de l'éléphant au virus du SIDA, paraissent ne pas
avoir un
génome initiateur commun, différent ,bien évidemment, entre celui du règne
végétal
celui du règne animal.
2. La composition des éléments constitutifs de l'être humain, l'ADN comme de
tous les
constituants des cellules humaines, est connue ; leur origine est soit la
chimie
minérale soit la chimie organique. Tout ce que les hommes créent, manipulent,
reste
cependant inerte et sans avenir propre comme ces mêmes substances le sont dans
le corps
humain. Les généticiens-biologistes ne savent pas les " mettre en
vie ", ne savent pas comment ces mêmes substances accourent dès la
naissance
chez les êtres vivants comme dans le règne végétal et leur permettent de
croître puis
de mourir
" Qu'est-ce que la vie ? Ce problème s'est posé à l'homme
depuis des
millénaires, et malgré les immenses progrès de la biologie, le statut du
vivant, reste,
quoi qu'on en pense, toujours aussi incertain : les tentatives de
réduction de
l'organisme au physico-chimique laissent toujours un résidu inexplicable,
tandis que les
définitions du vivant hésitent entre la tautologie et l'irrationnel. L'étude du
vivant,
contrairement aux autres sciences, ne peut se passer de l'idée de finalité...
Ni la
finalité théologique ni la réduction de la vie à la matière, ni les théories
vitalistes n'apportent une réponse satisfaisante à l'énigme de la finalité
biologique.
Quant à l'explication du rôle du hasard, même associé à la sélection naturelle,
une
analyse probabiliste sérieuse montre qu'elle se heurte à des difficultés
insurmontables.
Le fait que la vie soit compréhensible reste donc incompréhensible. C'est une
leçon
d'humilité pour la raison, mais n'empêche pas la biologie de fonctionner et de
progresser "
112(
*
)
.
Les hommes d'aujourd'hui restent pantois, le souffle coupé par l'émotion, la
surprise en
découvrant cette merveille d'organisation minutieuse, astucieuse dans ses
moindres
détails qui permet à l'homme d'assurer les fonctions multiples et diverses de
son
esprit, de son corps, de tous ses organes ; ils en découvrent, en même
temps les
fragilités et les possibilités de remédier à des conséquences qui peuvent être
désastreuses. Ils s'interrogent sur son origine et craignent de n'en point
trouver une
explication satisfaisante. Les uns se réfugient dans la tranquillité d'un
créateur
omnipuissant, omniscient, d'autres acceptent une évolution depuis l'apparition
de la
première cellule dans la soupe originelle après le big-bang ; ils
s'appuient sur
les étapes connues depuis l'
Homo
erectus
jusqu'à l'
Homo
sapiens
sapiens
ou sur les évolutions des plantes comme les angiospermes
ANNEXE N° IV
ANNEXE N° IV
ENTRETIENS DU RAPPORTEUR ET PARTICIPATION À DES COLLOQUES
ENTRETIENS
DU RAPPORTEUR ET PARTICIPATION À DES COLLOQUES
Le rapporteur a été reçu, le 15 juin 1999 par M. Claude ALLÈGRE, ministre de l'éducation nationale, de la recherche et de la technologie et le 29 juin 1999, par M. Bernard KOUCHNER, secrétaire d'État à la Santé.
|
24/09/1998 |
Centre de recherche
de Synthélabo à Bagneux. Visite organisée par
le Syndicat national de l'industrie pharmaceutique :
|
|
06/10/1998 |
- M. F. Sauer, directeur exécutif de l'EMEA (European Agency for the Evaluation of Medicinal Products).
|
|
12/10/1998 |
" Les inventions biotechnologiques : protection et exploitation ". Colloque organisé par l'Institut de recherche en propriété intellectuelle Henri-Desbois en collaboration avec l'Académie de droit européen de Trèves.
|
|
13/10/1998 |
" Publique et privée, quelles conditions pour une recherche commune ? ". Cercle institutionnel Rhône-Poulenc Rorer.
|
|
16/10/1998 |
" Les médicaments de demain ". Colloque organisé par le Conseil de l'ordre des pharmaciens.
|
|
25/11/1998 |
- M. F. Meyer, directeur général de la recherche. Rhône-Poulenc Rorer.
|
|
27/11/1998 |
" Économie-santé " IVe forum international de la gestion de la santé organisé par le groupe " Les Échos ".
|
|
10/12/1998 |
- Dr O. Amédée-Manesme, directeur des affaires scientifiques, pharmaceutiques et médicales. Syndicat national de l'industrie pharmaceutique.
|
|
15/12/1998 |
Petit déjeuner de
presse :
|
|
16/12/1998 |
- Pr J.-L . Salzmann, professeur des universités, praticien hospitalier à l'UFR-SMBH de Bobigny ; co-fondateur de la société Génopoïétic.
|
|
21 et 22/01/1999 |
Déplacement à
Lyon :
|
|
27/01/1999 |
- M. B. Tocqué, président directeur général de la société ExonHit Therapeutics.
|
|
10/02/1999 |
- Dr F. Thomas, société Bioserve.
|
|
11/02/1999 |
Déplacement à
Strasbourg :
|
|
17/02/1999 |
- Mme G. Berger, directrice du département bio-ingénierie au ministère de l'éducation nationale, de la recherche et de la technologie.
|
|
09/03/1999 |
- Pr. A. Revol, professeur émérite à l'université Lyon I, biologiste honoraire des hôpitaux de Lyon.
|
|
11/03/1999 |
Réunion à l'Académie des sciences pour l'étude du pré-rapport " Développement et applications de la génomique - L'après-génome ", sous la présidence du professeur François Gros, secrétaire perpétuel de l'Académie des sciences.
|
|
12/03/1999 |
Colloque organisé par le
rapporteur au Palais du Luxembourg sur :
" Les conséquences de l'évolution des sciences
biomédicales : enjeux de
santé et réponse politiques " avec la participation de :
|
|
16/03/1999 |
- Dr P.-H. Gouyon, professeur de génétique à l'université de Paris-Sud-Orsay.
|
|
19/03/1999 |
Visite du Génopôle
d'Évry :
|
|
28/03 au 02/04/1999 |
Déplacement aux États-Unis : |
|
|
Boston :
|
|
|
Philadelphie :
|
|
|
San
Francisco :
|
|
16/04/1999 |
Déplacement à
Grenoble :
|
|
05/05/1999 |
- Pr. D. Strosberg, vice président de la Société
Hybrigenics et vice-président de l'association France Bio-Tech ;
|
|
18/05/1999 |
- Pr. B.-P. Roques, membre de l'Académie des sciences, directeur de l'unité de pharmacochimie moléculaire et structurale (Paris V).
|
|
03/06/1999 |
Déplacement à
Lille :
|
|
01/07/1999 |
- M. Capdeville, président de la Fédération nationale des syndicats pharmaceutiques.
|
|
01/07/1999 |
- M. J.-R. Fourtou, président directeur général
de
Rhône-Poulenc
|
|
|
|
ANNEXE
n° V : Lettre de saisine de l'Office
ANNEXE
n° V : Lettre de saisine de l'Office

1
P. KOURILSKY, Préface " Des gènes aux protéines ".
Bibliothèque Pour la Science, diffusion Belin.
2
Catherine VINCENT.
Le Monde
. Numéro spécial : Le
Siècle.
3
Oui 2. Denoël. 1971.
4
Adénine, Thymine, Guanine, Cytosine.
5
Le Séquençage. Document fourni par le Centre national de
séquençage.
6
Le Monde
- 3 juin 1998.
7
Cf. entretien du rapporteur avec M. Manfred ZORN. Center for
bioinformatics and computational genomics. Université de Berkeley, Californie.
2 avril 1999.
8
Le Monde
. 24 mars 1999.
9 Le génome des organismes eucaryotes tels que les plantes, les animaux, les
champignons,
les levures, est protégé par une membrane à l'intérieur du noyau de la cellule.
10
Jean WEISSENBACH. ENA Mensuel n° 285.
11
Françoise FOURY. Revue Gène. 195.1.1997.
12
ENA Mensuel n° 285.
13
Pierre CASELLAS, Directeur du centre d'immunologie de Sanofi
Recherche à
Montpellier.
14
Fiche " Informatique et séquençage ".
Génoscope.
15
Laboratoire d'électronique, de technologie et d'instrumentation.
16
PCR :
Polymerase chain reaction
ou réaction de
polymérisation
en chaîne. Ce procédé permet de copier en grande quantité une séquence
quelconque
d'ADN en un temps très bref.
17
AMIGO : Présentation générale - Février 1999. LETI/CEA.
18
AMIGO : Présentation générale - Février 1999. LETI/CEA.
19
La société Affymetrix a intenté un procès à Incyte
Pharmaceuticals et
à Synteni. Elle est elle-même attaquée par Hyseq pour deux de ses brevets...
20
J. DANGOUMAU, Professeur des universités, praticien hospitalier,
pharmacologie.
21
Cf. la thèse de Line BOUREL : " Synthèse
combinatoire
appliquée à la découverte de nouveaux médicaments ". Université de
Lille.
1997.
22
Pour la science
. n° 241 Novembre 1997.
23
Pour la Science
. n° 241. Novembre 1997.
24
B. MERRIFIELD a reçu pour ces travaux le prix Nobel de chimie en
1984.
25
André TARTAR. Directeur de la CEREP.
L'Usine nouvelle.
Hors série.
Novembre 1998.
26
Michel KACZOREK. Président de Syntem.
L'Usine Nouvelle.
Hors série.
Novembre 1998.
27
" Une nouvelle stratégie de recherche ".
Synthélabo.
28
" Quels antibactériens pour demain ? ",
J.-F. DESNOTTES.
La Recherche
n° 314. Novembre 1998.
29
Rapport de l'OTA (Office of Technology Assessment). 1984.
30
Le kilobase (kb) est une unité de taille correspondant à
1000 paires
de bases d'ADN. La taille moyenne d'un gène est de 1 à 2 kb.
31
La transduction est le transfert de séquences d'ADN entre
différents
génomes par l'intermédiaire d'un virus.
32
Séquences situées avant ou après le gène proprement dit sur le
filament
d'ADN et impliquées dans l'efficacité de la production des protéines.
33
Pr Alain FISCHER.
34
AFM. Chronique des mille jours de la thérapie génique. N° 6.
Novembre 1998.
35
Dr. LEBOULCH (Harvard Medical School, Massachusetts) et
Dr. BACHELOT (Centre Léon Bérard. Lyon).
36
Société Gencell RPR.
37
Chronique des mille jours de la thérapie génique. N° 3 mars
1998 ; AFM.
38
" Thérapie génique : thérapie du gène et gène
médicament ". Axel KAHN.
39
Source :
www.wiley/genetherapy.com
. Transgène février
1999.
40
Source : L'Usine nouvelle - Hors série - Mars 1999.
41
" Targeted gene correction : a new strategy for
molecular
medicine ". S. YE, A. COLE-STRAUSS, B. FRANK,
E. KMIEC.
Molecular
Medicine Today
. Oct. 1998.
42
Afp, 15 novembre 1998 - Comptes-rendus de l'Académie des
Sciences.
43
Note de presse : CEA-INSERM - 30 mars 1998.
44
Les cellules dendritiques sont des cellules spécialisées dans la
présentation d'antigènes.
45
" Immunité antitumorale et perspectives
d'immunothérapie ". Marina OSTANKOVITCH, Agnès BUZYN, Sacha GNJATIC,
Jeannine
CHOPPIN, Jean-Gérard GUILLET (Laboratoire d'immunologie des pathologies
infectieuses et
tumorales. INSERM, U 445. Médecine thérapeutique. Vol 3. n° 9.
Novembre
1997.
46
Le pistolet à gènes ou " gene gun " est un
appareil
permettant de faire pénétrer, à l'intérieur des cellules, l'ADN fixé sur des
microparticules d'or.
47
Pour la Science
. N° 263. Septembre 1999.
48
On appelle antigène toute molécule capable d'être reconnue par le
système immunitaire et de provoquer une réponse de ce système.
49
" Qu'apporte la connaissance du génome de Helicobacter
Pylori ? " Agnès LABIGNE, Peter JENKS, Véronique MAZARIN,
Marie-Josée
QUENTIN-MILLET.
La lettre de l'infectiologue
. 1998.
50
" Attenuation of virulence by disruption of
Mycobacterium
tuberculosis erp gene ". F-X BERTHET, M. LAGRANDERIE,
P. GOUVION,
C. LAURENT-WINTER, D. ENSERGUEIX, P. CHAVAROT, F. HOURON,
E. MARANGHI, V. PELICIE, D. PORTNOÏ, G. MARCHAL,
B. GICQUEL.
Science
,
23 octobre 1998.
51
Source : The Scientific future of DNA for immunization.
Colloquium
of the American Academy of Microbiology. Juin 1996.
52
Définition de la pharmacogénomique par le Professeur George
POSTE.
53
Tableau et citations extraits et traduits de l'article
" This
drug's for you ".
Business Week.
18 janvier 1999.
54
Tableau et citation extraits et traduits de l'article
" This
drug's for you ".
Business Week
. 18 janvier 1999.
55
J. DANGOUMAU, Professeur des universités, praticien hospitalier,
pharmacologie.
56
Le technoscope de Biofutur
. N° 166. Avril 1997.
57
Gènes considérés comme indispensables au développement d'une
tumeur.
58
New Drug Discovery Technologies. Lehman Brothers.
59
" Publique et privée : quelles conditions pour une
recherche
commune ? " Intervention de François MEYER, directeur de la
recherche de
Rhône Poulenc Rorer. Cercle institutionnel, 13 octobre 1998.
60
Source :
Med Ad News,
Mai 1997.
Millenium Predictive Medicine.
61
World Review 1999. The Pharmaceutical Market. IMS ELF,
Londres.
62
" Les alliés de la biotech. Laboratoires et compagnies
de
biotechnologie ". Dr. François THOMAS. Pharmaceutiques. Octobre 1998.
N° 60.
63
Courrier ANVAR. N° 113. Septembre 1998.
64
" Biotechnologies et Santé ".
Publication
Eurasanté. Lille 1999.
65
Source : Les Chiffres clés de la science et de la technologie
1998-1999,
Observatoire des
sciences et des techniques
,
Economica, 1998.
66
Institut Pasteur : Rapport 1997.
67
Institut Pasteur : Rapport 1998.
68
Claude GRISCELLI.
La Revue Parlementaire
. Supplément.
3
e
trimestre
1998.
69
" La technologie et l'innovation " Henri
GUILLAUME.
Coll. des rapports officiels. Doc. fr. 1998.
70
Propos de M. J. BAIXERAS, adjoint du délégué aux
entreprises du
CNRS, cités dans l'article " Un nouveau statut pour le chercheur
créateur
d'entreprise ".
Les ÉCHOS
. 13/01/1999.
71
Dossier Valorisation. Biofutur n° 183. Novembre 1998.
72
Le Monde
. 22 septembre 1999.
73
Entretiens du rapporteur avec P. CHAMBON, professeur au
Collège de
France, directeur de l'Institut de génétique et de biologie moléculaire et
cellulaire.
Strasbourg. 11 février 1999.
74
" ADN et évolution " Richard MOXON et
Christopher
WILLS.
Pour la Science,
n° 257. Mars 1999.
75
David BALTIMORE. Prix Nobel de médecine. Intervention au Forum
Biovision.
Lyon. Mars 1999.
76
Nature
. 1998. 279.
77
" Principe de la lecture électronique de l'hybridation
de l'ADN
sur support solide ". E. SOUTEYRAND, J.-P. CLOAREC,
C. CHICHER,
J.-R. MARTIN.
78
Source : Association France-Biotech. Octobre 1999.
79
Les biopoles font bouger l'Europe.
L'Usine nouvelle
. Hors
série.
Mars 1999.
80
Courrier ANVAR
. N° 113. Septembre 1998.
81
L'Usine nouvelle
. Hors série. Mars 1999.
82
L'Usine nouvelle
. Hors série. Mars 1999.
83
Source : Association Alsace Biovalley.
84
Elle devra être transposée dans le droit national des États
membres au
plus tard le 30 juillet 2000.
85
Article 5 de la directive 98/44.
86
La protection juridique des inventions biotechnologiques après
l'adoption
de la directive européenne. Laurence TELLIER-LONIEWSKI.
Gazette du
Palais.
21
janvier 1999.
87
" Patent on gene fragments sends researchers a mixed
message ". Tony REICHARDT.
Nature
. Vol. 396. Dec 1998.
88
Déclaration d'Arthur HOLDEN, président directeur général du SNP
Consortium. 15 avril 1999.
89
Les nouveaux métiers des biotechnologies. L. BOURLANDE.
La
Recherche
. Septembre 1998.
90
Washington Post
. ST Presse de l'Ambassade de France.
7 Juin
1999.
91
www.infobiogen.fr
92
La chorée de Huntington est une maladie neurodégénérative
incurable.
Elle débute en moyenne vers 40-50 ans, provoquant des mouvements anormaux,
des
troubles du comportement, un démence et la mort, au bout d'une quinzaine
d'années.
93
Jeremy RIFKIN. Dossier Biofutur,. n° 181. Septembre 1998.
94
Axel KAHN. Génétique, médecine et société. ENA Mensuel, N° 285.
95
La Découverte
. Paris. 1998.
96
R. COORSH.. Health Insurance Association of America.
97
SNP : Single nucleotide polymorphism.
98
La France face au défi des biotechnologies : quels enjeux
pour
l'avenir ? Rapport présenté par M. ROUVILLOIS et M. LE FUR.
Conseil
économique et social. 1999.
99
In Bulletin Unesco. Compte rendu conférence mondiale sur la
science,
26 juin-1
er
juillet 1999.
100
D'après les réflexions de Mme Claire RODRIGUEZ-LAFRASSE,
maître de
conférences des universités, particien hospitalier en biochimie et biologie
moléculaire. CHU de Lyon.
101
" A hypothetic case in 2010 ".
The New
England
Journal of Medicine
. 1
er
juillet 1999.
102
Jacques Dangoumau, Professeur des universités, praticien
hospitalier,
pharmacologie.
103
Ethica. N° 28/29. Juin 1999.
104
Wolff et al. (1990)
Science
247: 1465.
105
FIV : feline immunodeficiency virus.
106
SIV : simian, immunodeficiency virus.
107
Ancien vaccinologiste du CNRS.
108
PowderJect Pharmaceutical a été fondée en 1993 pour exploiter les
recherches réalisées à l'Université d'Oxford (Angleterre) et se trouve basée à
Madison, Wisconsin.
109
Petite start-up créée en 1992 en Pennsylvanie, Apollon n'a jamis
réussi
à devenir publique. Son approche technologique rentrait en conflit avec les
brevets de
Vical.
110
Cette collaboration s'élève à 40 millions de dollars.
Source :
Bioworld Today
. 26 janvier 1999.
111
4 bases et 20 acides aminés.
112
Le Paradoxe de la Vie. Francis Kaplan La Découverte.
Janvier 1995.