1.1.4. LA CHIMIE COMBINATOIRE ET LE CRIBLAGE À HAUT DÉBIT
" L'apparition de nouvelles technologies a
bouleversé
la recherche de nouveaux médicaments dans sa phase initiale. Celle-ci
inclut tout d'abord la synthèse et l'isolement de nouvelles
molécules puis leur essai sur des systèmes biologiques permettant
de présupposer d'un intérêt thérapeutique
éventuel. Cette phase était classiquement longue et
pénible. La synthèse chimique relevait d'un art difficile ;
au départ, le choix d'une structure de base se faisait sans guide. Les
essais sur les animaux entiers ou les organes isolés étaient
longs et complexes. Au total, malgré des progrès au fil des
années, le processus relevait plus de la " pêche à la
ligne " que de la démarche rationnelle. Trois approches ont
profondément transformé cette recherche.
1.
Les techniques conformationnelles
La théorie des récepteurs postule que c'est l'union de la
molécule de médicament avec une macromolécule (une
protéine en général) qui est à l'origine de l'effet
pharmacodynamique et plus généralement de la réponse
thérapeutique. Cette union est fortement spécifique : seules
quelques molécules privilégiées en sont capables. On dit
que le médicament est comme " la clé dans la serrure ".
On sait maintenant déterminer la conformation dans l'espace des
protéines, notamment grâce à la radiocristallographie aux
rayons X, donc celle des récepteurs. On peut donc prévoir quelles
structures devront présenter les molécules pour pouvoir s'unir
à eux. Cette recherche est aidée par les programmes informatiques
qui permettent de visualiser les molécules et de les faire tourner dans
l'espace (conception assistée par ordinateur).
Bien que hautement sophistiquée et évidemment plus ardue que ces
quelques lignes pourraient le laisser croire, cette approche permet de ne plus
s'en remettre au hasard dans la recherche des séries chimiques
intéressantes. On voit, cependant, qu'il est indispensable de
connaître au départ le récepteur pertinent,
c'est-à-dire d'avoir une hypothèse physio-pathologique et d'avoir
été capable d'identifier et d'isoler la protéine qui le
porte. Là aussi, des progrès décisifs ont
été faits dans l'isolement des protéines et, mieux
-encore, dans le repérage et le clonage des gènes qui commandent
leur synthèse.
2.
La chimie combinatoire
Il est désormais possible de synthétiser en une seule
opération plusieurs centaines de molécules c'est ce que l'on
appelle la chimie combinatoire. On part de la structure de base
déterminée a priori comme il vient d'être dit et on
génère systématiquement toutes les variations possibles en
greffant des radicaux chimiques, des chaînes latérales, en
modifiant le squelette, etc. Ceci se fait non plus étape par
étape, mais en mettant en présence les réactifs
nécessaires. On obtient ainsi d'un seul coup plusieurs centaines de
molécules. Toutes les opérations, synthèse, isolement et
identification, sont miniaturisées et robotisées. Le gain de
temps et l'abaissement des coûts sont considérables. On peut ainsi
constituer une bibliothèque de plusieurs milliers de
dérivés en quelques mois.
3.
Le criblage à haut débit
Le problème est alors d'identifier parmi toutes ces molécules
celles qui sont pourvues des propriétés biologiques les plus
intéressantes. Le gain de temps et l'augmentation de la
productivité apportés par la chimie combinatoire l'auraient
été en vain si la productivité de cette phase de
repérage appelée " criblage " n'avait pas
été aussi améliorée. Aux essais longs et
limités de la pharmacologie expérimentale classique, a
succédé une technique qui permet d'essayer dans le minimum de
temps des milliers de molécules.
Le test consiste à mette en présence la substance à tester
et un système biochimique (une enzyme par exemple) et de mesurer
l'importance de la réaction éventuelle. L'essai peut être
fait simultanément avec un grand nombre de systèmes, de
significations très diverses. En fait, tout est miniaturisé et
robotisé. Les opérations se déroulent sans intervention
humaine et les résultats sont lus sur l'imprimante.
Tout dépend de ce que l'on met dans les tubes et, une fois de plus, on
ne trouvera que ce que l'on cherche. C'est dire l'importance des
hypothèses physiopathologiques. Les systèmes biologiques
testés ne sont pas indifférents : ce sont ceux dont on pense
qu'ils interviennent de manière cruciale dans le déterminisme de
la maladie. On voit donc bien que cette recherche appliquée, dont les
progrès sont si remarquables, est totalement tributaire de la recherche
fondamentale d'amont.
L'opération finale est celle du choix. La plupart du temps, toutes les
molécules intéressantes ne peuvent pas passer en
développement. Il faut donc sélectionner les plus prometteuses,
compte tenu de leurs résultats aux tests, mais aussi d'autres
considérations comme leur facilité de synthèse ou leur
pharmacocinétique. Si les opérations répétitives
ont été automatisées, l'inventivité de l'esprit
humain intervient donc toujours dans les choix des hypothèses, des tests
et in fine des molécules. "
( 20( * ) )
1.1.4.1. Chimie combinatoire et constitution de chimiothèques
La chimie combinatoire
Le principe de la chimie combinatoire est d'accroître les chances de trouver une molécule active, un candidat-médicament, en multipliant le nombre de molécule étudiées 21( * ) .
La synthèse
C'est
une sorte de jeu de construction qui permet de synthétiser de
très nombreuses molécules par combinaison. Les chimistes, en
cela, imitent la nature qui, à partir de quelques molécules
élémentaires, a construit toute la variété des
gènes et des protéines.
Au cours des années 1980, les immuno-chimistes cherchaient à
établir la structure des fragments des protéines virales
(peptides viraux) qui déclenchent les réactions du système
immunitaire d'un organisme infecté par un virus. Pour synthétiser
de nombreux peptides de 5 à 12 acides aminés, ils ont mis au
point une méthode appelée synthèse en parallèle.
La synthèse en parallèle
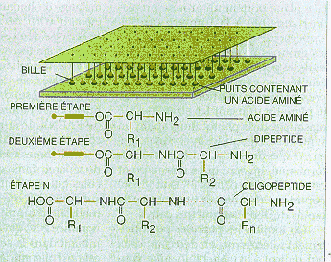
Dans la
synthèse en parallèle, une petite bille de polystyrène est
fixée à une tige de polyéthylène ou
" pin "
. Chacun des puits de la plaque contient un seul acide
aminé.
On plonge tous les pins dans les puits, et l'acide aminé présent
dans chaque puits se greffe sur la bille immergée, par
l'intermédiaire d'un " bras " (le cylindre). Puis on change
les acides aminés contenus dans les puits, et les billes sont à
nouveau immergées. Le nouvel acide aminé se lie au premier,
formant un dipeptide. Après
n
étapes, on obtient un
oligopeptide qui est libéré dans le puits après rupture du
bras qui le reliait à la bille de polystyrène.
Par cette méthode, on obtient autant de peptides différents qu'il
y a de puits, et ces molécules sont formées par la succession des
acides aminés qui ont été successivement introduits dans
les puits.
(22( * ))
La
méthode combinatoire est dérivée de cette synthèse
mais elle est beaucoup plus puissante car elle produit un nombre de
molécules croissant exponentiellement à chaque étape de la
synthèse. Il s'agit alors d'une synthèse dite divergente ou
dissociée.
LES DIFFÉRENTES MÉTHODES DE SYNTHÈSE DES BANQUES
COMBINATOIRES
La
synthèse en parallèle divergente
La synthèse par répartition et mélange
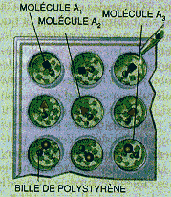


Étape 1
On dispose d'un jeu de trois molécules A 1 , A 2 , A 3 . Ces molécules sont fixées sur des billes de polystyrène différentes.
Étape 2
On ajoute dans chacun des puits le mélange des trois molécules A 1 , A 2 , A 3 . On obtient les molécules A 1 A 1 , A 1 A 2 , A 1 A 3 ; A 2 A 1 , A 2 A 2 , A 2 A 3 ; A 3 A 1 , A 3 A 2 , A 3 A 3 (pour simplifier, on n'a représenté qu'un type de molécules par puits, mais, en réalité, les trois types de molécules sont mélangés, sur chaque ligne).
Étapes suivantes
Au bout
de deux étapes, on a synthétisé 3
2
, soit neuf
molécules différentes (en recommençant
n
fois la
même opération, on obtient 3
n
molécules).
Toutefois, les billes portent toutes un mélange de molécules, ce
qui rend l'identification de la molécule active difficile.
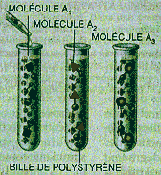
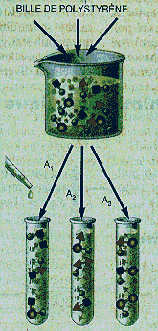
MOLÉCULES
A
1
A
1
A
1
A
2
A
1
A
3
A
2
A
1
A
2
A
2
A
2
A
3
A
3
A
1
A
3
A
2
A
3
A
3
Étape 1
Des billes de polystyrène sont introduites dans les tubes à essai. Les molécules A 1 sont placées dans le premier tube, les molécules A 2 dans le deuxième, etc. On n'a représenté que trois tubes, mais on peut en utiliser des dizaines.
Étape 2
Le contenu des tubes est filtré et rincé, et toutes les billes sont regroupées et mélangées.
Étape 3
Le mélange est partagé en trois, et des molécules A 1 , sont introduites dans le tube 1, des molécules A 2 dans le tube 2, etc.
Étapes suivantes
On recommence les opérations de mélange et de partage autant de fois qu'il est nécessaire pour ajouter toutes les sous-unités voulues. Une bille ne porte qu'un seul type de molécules, car, à chaque étape, les billes ne sont en contact qu'avec un seul réactif. Un test colorimétrique identifie la bille qui porte la molécule active.
(23( * ))
Ce type
de synthèse a été rendu possible par le fait que
B. MERRIFIELD
24(
*
)
a
réussi à développer des peptides sur de petites billes de
polymère chimiquement inertes.
La chimie combinatoire utilise ces billes de polymère de la façon
suivante : placées dans un solvant, les billes gonflent, de sorte
que l'intérieur devient accessible aux réactifs dissous dans ce
solvant. Chaque bille porte environ 10
13
amorces qui se combinent
avec les réactifs. Par réactions successives on obtient des
oligomères, des molécules constituées d'un
enchaînement de plusieurs sous-unités.
Le marquage des molécules
La
seconde étape consiste à " étiqueter " les
molécules au fur et à mesure de leur élaboration, de
façon à pouvoir identifier chacune d'elles à la fin du
processus. Plusieurs techniques sont utilisées :
- La première est l'ajout, à chaque stade de la
synthèse, d'un composé inerte sur un site distinct de la
bille ; cela crée une sorte de signature chimique de la
molécule ;
- La seconde est l'ajout, à chaque stade également, d'une
base (A, T, G ou C) qui permettra ultérieurement de reconnaître la
molécule en lisant la séquence après amplification par PCR
(
polymerase chain reaction
) ;
- La société française CEREP utilise des codes barres
pour assurer la traçabilité des produits.
" Chaque
opération, chaque produit est référencé.
L'ordinateur lit les codes et permet au chimiste d'accéder directement
aux informations qu'il recherche ou au robot de savoir quels
échantillons il traite "
.
25(
*
)
- La société californienne Irori a lancé, en juin
1996, le système Accutag : il s'agit de développer des
molécules sur des billes contenues dans une capsule où se trouve
également un microprocesseur. La paroi de chaque capsule est comme un
filet qui laisse passer les réactifs chimiques nécessaires
à la synthèse, mais retient les billes de polymère.
À chaque étape de la synthèse, un signal radio est
envoyé à chaque microprocesseur afin d'enregistrer en
détail la réaction à laquelle la bille est exposée.
Ces données peuvent ensuite être lues sur les microprocesseurs
lorsque les molécules en sont arrivées à leur stade final.
Toutefois, le système électronique utilisé reste trop
volumineux pour permettre de produire des centaines de milliers de
composés.
C'est pourquoi, Irori a signé des accords avec Rhône-Poulenc Rorer
et Bristol-Myers Squibb pour mettre au point un nouveau procédé
qui devrait, à terme, permettre de synthétiser près de
25 000 composés par semaine. L'idée est d'utiliser des
étiquettes code à barres à deux dimensions et à
lecture optique au lieu du système des étiquettes à
radiofréquence c'est-à-dire des " volumineux "
microprocesseurs contenus dans la capsule.
Les nouvelles molécules créées par la chimie combinatoire
sont rassemblées dans de vastes chimiothèques. Les
chimiothèques " généralistes " fournissent une
grande diversité de structures moléculaires. Toutefois, ces
molécules sont d'un intérêt inégal et il est
indispensable de les trier pour déterminer les plus actives et quel type
de cible elles peuvent atteindre.
UNE
NOUVELLE FAÇON DE CRÉER DES MOLÉCULES
INÉDITES :
LES MOLÉCULES DARWINIENNES
" Faisant appel aux principes de la sélection
naturelle, un logiciel engendre des molécules-modèles
inédites, futures candidates au statut de médicament.
Plusieurs modèles chimiques permettent déjà de trouver
informatiquement de nouvelles molécules actives. Le dernier logiciel en
date propose d'appliquer la sélection naturelle darwinienne sur des
modèles de molécules. Les résultats de David NOEVER lui
ont valu le titre d'innovator de l'année 1998, décerné par
le magazine américain Discover. Grâce à cette idée,
David NOEVER a fondé une start-up qui travaille notamment avec le
National Cancer Institute (NCI) et une dizaine d'entreprises privées.
Son logiciel part d'une molécule fournie par un laboratoire, connue pour
son action sur certaines maladies. Il en construit ensuite jusqu'à
trente " parents ", des molécules élaborées
automatiquement selon des critères géométriques,
physico-chimiques ou autres.
A chaque molécule est associé un " Chromosome " fictif
représentant ses caractéristiques. L'algorithme déclenche
alors un processus de reproduction sur cette population de chromosomes, en
autorisant les mutations (changement ponctuel et aléatoire) et les
cross-over (échange d'une séquence entre deux chromosomes).
À chaque génération, le logiciel choisit les meilleurs
reproducteurs, par exemple les molécules ayant la configuration de plus
basse énergie. En calculant quelques milliers de
générations, on voit apparaître de nouvelles
molécules, parmi lesquelles seules les meilleures
" survivent ". David NOEVER précise que, " lors des
expériences réalisées pour le National Cancer Institute,
les experts du NCI ont toujours vérifié que les molécules
proposées n'avaient jamais été observées
auparavant ". En jouant sur les paramètres de sélection,
NOEVER peut aussi modéliser la fixation de ligands sur une
protéine, ou encore prédire la forme exacte d'une molécule
d'après sa formule chimique, en un temps record: quelques minutes sur
une station de travail. "
La Recherche. N° 313 . Octobre 1998 .
1.1.4.2. Le criblage à haut débit
Les laboratoires pharmaceutiques ont tout intérêt à restreindre leur champ d'investigation expérimentale en le ciblant correctement pour aboutir à de nouveaux médicaments le plus vite possible à un moindre coût. C'est pourquoi ils passent au crible les nouvelles molécules ; ce criblage doit être réalisé à haut débit.
Le criblage virtuel
Cette technique relativement récente est notamment mise en oeuvre par la société Syntem. L'ordinateur aide le biochimiste à faire le tri parmi les molécules. " Par une évaluation rationnelle de l'activité biologique des molécules, on peut diminuer le nombre de molécules à synthétiser en laboratoire d'un facteur mille ou plus " 26( * ) . Puis les nouvelles molécules sont modélisées : leurs propriétés globales (exemple : affinité pour les graisses, solubilité...) sont analysées et ces descriptions sont communiquées à l'ordinateur qui réalise un premier tri, permettant de sélectionner les molécules qui, compte tenu de ces propriétés, ont le plus de chance d'être actives en fonction de telle ou telle cible.
Le criblage biologique
C'est
actuellement le procédé le plus utilisé. Il consiste
à identifier les molécules actives par mise en contact avec la
cible biologique. Ces cibles peuvent par exemple être des
protéines dont on a identifié expérimentalement
l'implication dans tel ou tel processus pathologique.
Jusqu'au début des années 1990, les essais traditionnels en
éprouvette permettaient à une personne de tester
2000 composés par an. L'emploi de microplaques à
96 puits a ensuite permis de réaliser 6 000 essais par
jour et par robot sur une même cible. Aujourd'hui les microplaques
comportent 384 puits, certaines pouvant aller jusqu'à
1 536 puits. Ces progrès ont permis de multiplier les tests
et, également, de réduire les coûts car les essais sont
" miniaturisés " et utilisent des volumes
d'échantillons très réduits.
Le criblage à haut débit se caractérise par l'utilisation
de robots capables de gérer simultanément de grands nombres de
microplaques, facilement manipulables et stockables, où sont
effectués des milliers de tests par jour.
Le criblage à haut débit est issu des progrès de la
robotique. Il repose sur des systèmes capables de réaliser des
taches séquentielles indépendantes telles que dilution, pipettage
et répartition de composés dans des cupules ou puits, agitation,
incubation, lecture de résultats. Ils sont pilotés par des
logiciels spécifiquement adaptés au type d'analyse à
réaliser. Les essais sont effectués dans des microplaques
standards identifiées par un code à barres et manipulées
par la " main " d'un robot : celle-ci prend la plaque vide,
ajoute les réactifs nécessaires et les composés à
tester, gère le temps de la réaction puis passe la plaque
à un lecteur pour connaître le résultat. Pour visualiser
les réactions issues de la mise en contact, dans les puits, de la
molécule et de la cible, on utilise des méthodes basées
sur la radioactivité ou, bien souvent, la fluorescence. Ces
résultats sont stockés et analysés grâce à
des ordinateurs.